+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the endogenous core TIM23 complex from S. cerevisiae | |||||||||
 マップデータ マップデータ | Unsharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | TRANSLOCASE-IMMUNE SYSTEM complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報TIM23 mitochondrial import inner membrane translocase complex / protein import into mitochondrial matrix / protein transmembrane transporter activity / protein-folding chaperone binding / mitochondrial inner membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
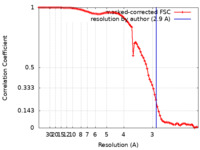
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Sim SI / Park E | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2023 ジャーナル: Nature / 年: 2023タイトル: Structural basis of mitochondrial protein import by the TIM23 complex. 著者: Sue Im Sim / Yuanyuan Chen / Diane L Lynch / James C Gumbart / Eunyong Park /  要旨: Mitochondria import nearly all of their approximately 1,000-2,000 constituent proteins from the cytosol across their double-membrane envelope. Genetic and biochemical studies have shown that the ...Mitochondria import nearly all of their approximately 1,000-2,000 constituent proteins from the cytosol across their double-membrane envelope. Genetic and biochemical studies have shown that the conserved protein translocase, termed the TIM23 complex, mediates import of presequence-containing proteins (preproteins) into the mitochondrial matrix and inner membrane. Among about ten different subunits of the TIM23 complex, the essential multipass membrane protein Tim23, together with the evolutionarily related protein Tim17, has long been postulated to form a protein-conducting channel. However, the mechanism by which these subunits form a translocation path in the membrane and enable the import process remains unclear due to a lack of structural information. Here we determined the cryo-electron microscopy structure of the core TIM23 complex (heterotrimeric Tim17-Tim23-Tim44) from Saccharomyces cerevisiae. Contrary to the prevailing model, Tim23 and Tim17 themselves do not form a water-filled channel, but instead have separate, lipid-exposed concave cavities that face in opposite directions. Our structural and biochemical analyses show that the cavity of Tim17, but not Tim23, forms the protein translocation path, whereas Tim23 probably has a structural role. The results further suggest that, during translocation of substrate polypeptides, the nonessential subunit Mgr2 seals the lateral opening of the Tim17 cavity to facilitate the translocation process. We propose a new model for the TIM23-mediated protein import and sorting mechanism, a central pathway in mitochondrial biogenesis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27825.map.gz emd_27825.map.gz | 32.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27825-v30.xml emd-27825-v30.xml emd-27825.xml emd-27825.xml | 27.1 KB 27.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27825_fsc.xml emd_27825_fsc.xml | 9.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27825.png emd_27825.png | 27.2 KB | ||
| Filedesc metadata |  emd-27825.cif.gz emd-27825.cif.gz | 7.5 KB | ||
| その他 |  emd_27825_additional_1.map.gz emd_27825_additional_1.map.gz emd_27825_half_map_1.map.gz emd_27825_half_map_1.map.gz emd_27825_half_map_2.map.gz emd_27825_half_map_2.map.gz | 54.3 MB 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27825 http://ftp.pdbj.org/pub/emdb/structures/EMD-27825 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27825 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27825 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8e1mMC  8scxC  24885  24886 M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27825.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27825.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Sharpened map
| ファイル | emd_27825_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_27825_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_27825_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Core TIM23 complex (endogenous)
| 全体 | 名称: Core TIM23 complex (endogenous) |
|---|---|
| 要素 |
|
-超分子 #1: Core TIM23 complex (endogenous)
| 超分子 | 名称: Core TIM23 complex (endogenous) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Mitochondrial import inner membrane translocase subunit TIM17
| 分子 | 名称: Mitochondrial import inner membrane translocase subunit TIM17 タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 16.602156 KDa |
| 配列 | 文字列: MSADHSRDPC PIVILNDFGG AFAMGAIGGV VWHGIKGFRN SPLGERGSGA MSAIKARAPV LGGNFGVWGG LFSTFDCAVK AVRKREDPW NAIIAGFFTG GALAVRGGWR HTRNSSITCA CLLGVIEGVG LMFQRYAAWQ AKPMAPPLPE APSSQPLQA UniProtKB: Mitochondrial import inner membrane translocase subunit TIM17 |
-分子 #2: Mitochondrial import inner membrane translocase subunit TIM23
| 分子 | 名称: Mitochondrial import inner membrane translocase subunit TIM23 タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 23.266527 KDa |
| 配列 | 文字列: MSWLFGDKTP TDDANAAVGG QDTTKPKELS LKQSLGFEPN INNIISGPGG MHVDTARLHP LAGLDKGVEY LDLEEEQLSS LEGSQGLIP SRGWTDDLCY GTGAVYLLGL GIGGFSGMMQ GLQNIPPNSP GKLQLNTVLN HITKRGPFLG NNAGILALSY N IINSTIDA ...文字列: MSWLFGDKTP TDDANAAVGG QDTTKPKELS LKQSLGFEPN INNIISGPGG MHVDTARLHP LAGLDKGVEY LDLEEEQLSS LEGSQGLIP SRGWTDDLCY GTGAVYLLGL GIGGFSGMMQ GLQNIPPNSP GKLQLNTVLN HITKRGPFLG NNAGILALSY N IINSTIDA LRGKHDTAGS IGAGALTGAL FKSSKGLKPM GYSSAMVAAA CAVWCSVKKR LLEK UniProtKB: Mitochondrial import inner membrane translocase subunit TIM23 |
-分子 #3: Mitochondrial import inner membrane translocase subunit TIM44
| 分子 | 名称: Mitochondrial import inner membrane translocase subunit TIM44 タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 48.933418 KDa |
| 配列 | 文字列: MHRSTFIRTS GTSSRTLTAR YRSQYTGLLV ARVLFSTSTT RAQGGNPRSP LQIFRDTFKK EWEKSQELQE NIKTLQDASG KLGESEAYK KAREAYLKAQ RGSTIVGKTL KKTGETMEHI ATKAWESELG KNTRKAAAAT AKKLDESFEP VRQTKIYKEV S EVIDDGES ...文字列: MHRSTFIRTS GTSSRTLTAR YRSQYTGLLV ARVLFSTSTT RAQGGNPRSP LQIFRDTFKK EWEKSQELQE NIKTLQDASG KLGESEAYK KAREAYLKAQ RGSTIVGKTL KKTGETMEHI ATKAWESELG KNTRKAAAAT AKKLDESFEP VRQTKIYKEV S EVIDDGES SRYGGFITKE QRRLKRERDL ASGKRHRAVK SNEDAGTAVV ATNIESKESF GKKVEDFKEK TVVGRSIQSL KN KLWDESE NPLIVVMRKI TNKVGGFFAE TESSRVYSQF KLMDPTFSNE SFTRHLREYI VPEILEAYVK GDVKVLKKWF SEA PFNVYA AQQKIFKEQD VYADGRILDI RGVEIVSAKL LAPQDIPVLV VGCRAQEINL YRKKKTGEIA AGDEANILMS SYAM VFTRD PEQIDDDETE GWKILEFVRG GSRQFT UniProtKB: Mitochondrial import inner membrane translocase subunit TIM44 |
-分子 #4: Antibody Fab fragment light chain
| 分子 | 名称: Antibody Fab fragment light chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 26.173201 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEKDTLLLWV LLLWVPGSTG DIVLTQSPAS LAVSLGQRAT ISCRASESVD IYGISFMNWF QQKPGQPPKL LIYATSNQGS GVPARFSGS GSGTDFSLNI HPMEEDDTAM YFCQQSKEVP RTFGGGTKLE IKRADAAPTV SIFPPSSEQL TSGGASVVCF L NNFYPKDI ...文字列: MEKDTLLLWV LLLWVPGSTG DIVLTQSPAS LAVSLGQRAT ISCRASESVD IYGISFMNWF QQKPGQPPKL LIYATSNQGS GVPARFSGS GSGTDFSLNI HPMEEDDTAM YFCQQSKEVP RTFGGGTKLE IKRADAAPTV SIFPPSSEQL TSGGASVVCF L NNFYPKDI NVKWKIDGSE RQNGVLNSWT DQDSKDSTYS MSSTLTLTKD EYERHNSYTC EATHKTSTSP IVKSFNRNEC |
-分子 #5: Antibody Fab fragment heavy chain
| 分子 | 名称: Antibody Fab fragment heavy chain / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 25.49499 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAVLVLLLCL VTFPSCVLSQ VQLKQSGPGL VQPSQSLSIT CTVSGFSLTT YGVHWVRQSP GKGLEWLGVM WRGGSTDFNA AFMSRLSIT KDNSKSQVFF KMNSLQADDT AIYYCARYGN YDAMDYWGQG TSVTVSSAKT TPPSVYPLAP GSAAQTNSMV T LGCLVKGY ...文字列: MAVLVLLLCL VTFPSCVLSQ VQLKQSGPGL VQPSQSLSIT CTVSGFSLTT YGVHWVRQSP GKGLEWLGVM WRGGSTDFNA AFMSRLSIT KDNSKSQVFF KMNSLQADDT AIYYCARYGN YDAMDYWGQG TSVTVSSAKT TPPSVYPLAP GSAAQTNSMV T LGCLVKGY FPEPVTVTWN SGSLSSGVHT FPAVLQSDLY TLSSSVTVPS SPRPSETVTC NVAHPASSTK VDKKIVPRDC |
-分子 #6: CARDIOLIPIN
| 分子 | 名称: CARDIOLIPIN / タイプ: ligand / ID: 6 / コピー数: 1 / 式: CDL |
|---|---|
| 分子量 | 理論値: 1.464043 KDa |
| Chemical component information |  ChemComp-CDL: |
-分子 #7: PHOSPHATIDYLETHANOLAMINE
| 分子 | 名称: PHOSPHATIDYLETHANOLAMINE / タイプ: ligand / ID: 7 / コピー数: 1 / 式: PTY |
|---|---|
| 分子量 | 理論値: 734.039 Da |
| Chemical component information |  ChemComp-PTY: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 40 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)