+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Local refinement around RCKW of LRRK1 | |||||||||||||||
 マップデータ マップデータ | Local refinement of RCKW of LRRK1 | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | monomer / TRANSFERASE | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
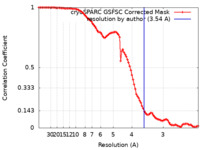
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.54 Å | |||||||||||||||
 データ登録者 データ登録者 | Reimer JM / Mathea S / Knapp S / Leschziner AE | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Structure of LRRK1 and mechanisms of autoinhibition and activation. 著者: Janice M Reimer / Andrea M Dickey / Yu Xuan Lin / Robert G Abrisch / Sebastian Mathea / Deep Chatterjee / Elizabeth J Fay / Stefan Knapp / Matthew D Daugherty / Samara L Reck-Peterson / Andres E Leschziner /   要旨: Leucine Rich Repeat Kinase 1 and 2 (LRRK1 and LRRK2) are homologs in the ROCO family of proteins in humans. Despite their shared domain architecture and involvement in intracellular trafficking, ...Leucine Rich Repeat Kinase 1 and 2 (LRRK1 and LRRK2) are homologs in the ROCO family of proteins in humans. Despite their shared domain architecture and involvement in intracellular trafficking, their disease associations are strikingly different: LRRK2 is involved in familial Parkinson's disease while LRRK1 is linked to bone diseases. Furthermore, Parkinson's disease-linked mutations in LRRK2 are typically autosomal dominant gain-of-function while those in LRRK1 are autosomal recessive loss-of-function. Here, to understand these differences, we solved cryo-EM structures of LRRK1 in its monomeric and dimeric forms. Both differ from the corresponding LRRK2 structures. Unlike LRRK2, which is sterically autoinhibited as a monomer, LRRK1 is sterically autoinhibited in a dimer-dependent manner. LRRK1 has an additional level of autoinhibition that prevents activation of the kinase and is absent in LRRK2. Finally, we place the structural signatures of LRRK1 and LRRK2 in the context of the evolution of the LRRK family of proteins. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27814.map.gz emd_27814.map.gz | 86 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27814-v30.xml emd-27814-v30.xml emd-27814.xml emd-27814.xml | 17 KB 17 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27814_fsc.xml emd_27814_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27814.png emd_27814.png | 34.3 KB | ||
| Filedesc metadata |  emd-27814.cif.gz emd-27814.cif.gz | 6 KB | ||
| その他 |  emd_27814_half_map_1.map.gz emd_27814_half_map_1.map.gz emd_27814_half_map_2.map.gz emd_27814_half_map_2.map.gz | 84.6 MB 84.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27814 http://ftp.pdbj.org/pub/emdb/structures/EMD-27814 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27814 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27814 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27814_validation.pdf.gz emd_27814_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27814_full_validation.pdf.gz emd_27814_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_27814_validation.xml.gz emd_27814_validation.xml.gz | 17.4 KB | 表示 | |
| CIF形式データ |  emd_27814_validation.cif.gz emd_27814_validation.cif.gz | 21.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27814 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27814 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27814 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27814 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27814.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27814.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Local refinement of RCKW of LRRK1 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.16 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map B LRRK1, focused refinement on RCKW
| ファイル | emd_27814_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B LRRK1, focused refinement on RCKW | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
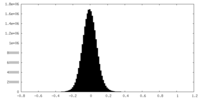
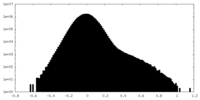
| 密度ヒストグラム |
-ハーフマップ: Half map A LRRK1, focused refinement on RCKW
| ファイル | emd_27814_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A LRRK1, focused refinement on RCKW | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
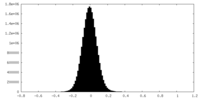
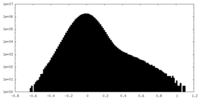
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Monomeric structure of LRRK1
| 全体 | 名称: Monomeric structure of LRRK1 |
|---|---|
| 要素 |
|
-超分子 #1: Monomeric structure of LRRK1
| 超分子 | 名称: Monomeric structure of LRRK1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Leucine-rich repeat kinase 1
| 分子 | 名称: Leucine-rich repeat kinase 1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SAVCPERAME TLNGAGDTGG KPSTRGGDPA ARSRRTEGIR AAYRRGDRGG ARDLLEEACD QCASQLEKGQ LLSIPAAYGD LEMVRYLLSK RLVELPTEPT DDNPAVVAAY FGHTAVVQEL LESLPGPCSP QRLLNWMLAL ACQRGHLGVV KLLVLTHGAD PESYAVRKNE ...文字列: SAVCPERAME TLNGAGDTGG KPSTRGGDPA ARSRRTEGIR AAYRRGDRGG ARDLLEEACD QCASQLEKGQ LLSIPAAYGD LEMVRYLLSK RLVELPTEPT DDNPAVVAAY FGHTAVVQEL LESLPGPCSP QRLLNWMLAL ACQRGHLGVV KLLVLTHGAD PESYAVRKNE FPVIVRLPLY AAIKSGNEDI AIFLLRHGAY FCSYILLDSP DPSKHLLRKY FIEASPLPSS YPGKTALRVK WSHLRLPWVD LDWLIDISCQ ITELDLSANC LATLPSVIPW GLINLRKLNL SDNHLGELPG VQSSDEIICS RLLEIDISSN KLSHLPPGFL HLSKLQKLTA SKNCLEKLFE EENATNWIGL RKLQELDISD NKLTELPALF LHSFKSLNSL NVSRNNLKVF PDPWACPLKC CKASRNALEC LPDKMAVFWK NHLKDVDFSE NALKEVPLGL FQLDALMFLR LQGNQLAALP PQEKWTCRQL KTLDLSRNQL GKNEDGLKTK RIAFFTTRGR QRSGTEAASV LEFPAFLSES LEVLCLNDNH LDTVPPSVCL LKSLSELYLG NNPGLRELPP ELGQLGNLWQ LDTEDLTISN VPAEIQKEGP KAMLSYLRAQ LRKAEKCKLM KMIIVGPPRQ GKSTLLEILQ TGRAPQVVHG EATIRTTKWE LQRPAGSRAK VESVEFNVWD IGGPASMATV NQCFFTDKAL YVVVWNLALG EEAVANLQFW LLNIEAKAPN AVVLVVGTHL DLIEAKFRVE RIATLRAYVL ALCRSPSGSR ATGFPDITFK HLHEISCKSL EGQEGLRQLI FHVTCSMKDV GSTIGCQRLA GRLIPRSYLS LQEAVLAEQQ RRSRDDDVQY LTDRQLEQLV EQTPDNDIKD YEDLQSAISF LIETGTLLHF PDTSHGLRNL YFLDPIWLSE CLQRIFNIKG SRSVAKNGVI RAEDLRMLLV GTGFTQQTEE QYFQFLAKFE IALPVANDSY LLPHLLPSKP GLDTHGMRHP TANTIQRVFK MSFVPVGFWQ RFIARMLISL AEMDLQLFEN KKNTKSRNRK VTIYSFTGNQ RNRCSTFRVK RNQTIYWQEG LLVTFDGGYL SVESSDVNWK KKKSGGMKIV CQSEVRDFSA MAFITDHVNS LIDQWFPALT ATESDGTPLM EQYVPCPVCE TAWAQHTDPS EKSEDVQYFD MEDCVLTAIE RDFISCPRHP DLPVPLQELV PELFMTDFPA RLFLENSKLE HSEDEGSVLG QGGSGTVIYR ARYQGQPVAV KRFHIKKFKN FANVPADTML RHLRATDAMK NFSEFRQEAS MLHALQHPCI VALIGISIHP LCFALELAPL SSLNTVLSEN ARDSSFIPLG HMLTQKIAYQ IASGLAYLHK KNIIFCDLKS DNILVWSLDV KEHINIKLSD YGISRQSFHE GALGVEGTPG YQAPEIRPRI VYDEKVDMFS YGMVLYELLS GQRPALGHHQ LQIAKKLSKG IRPVLGQPEE VQFRRLQALM MECWDTKPEK RPLALSVVSQ MKDPTFATFM YELCCGKQTA FFSSQGQEYT VVFWDGKEES RNYTVVNTEK GLMEVQRMCC PGMKVSCQLQ VQRSLWTATE DQKIYIYTLK GMCPLNTPQQ ALDTPAVVTC FLAVPVIKKN SYLVLAGLAD GLVAVFPVVR GTPKDSCSYL CSHTANRSKF SIADEDARQN PYPVKAMEVV NSGSEVWYSN GPGLLVIDCA SLEICRRLEP YMAPSMVTSV VCSSEGRGEE VVWCLDDKAN SLVMYHSTTY QLCARYFCGV PSPLRDMFPV RPLDTEPPAA SHTANPKVPE GDSIADVSIM YSEELGTQIL IHQESLTDYC SMSSYSSSPP RQAARSPSSL PSSPASSSSV PFSTDCEDSD MLHTPGAASD RSEHDLTPMD GETFSQHLQA VKILAVRDLI WVPRRGGDVI VIGLEKDSGA QRGRVIAVLK ARELTPHGVL VDAAVVAKDT VVCTFENENT EWCLAVWRGW GAREFDIFYQ SYEELGRLEA CTRKRR |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 詳細: 50 mM HEPES pH 7.4, 150 mM NaCl, 5% glycerol, 0.5 mM TCEP, 20 uM GDP |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 51.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.124 µm 最小 デフォーカス(公称値): 1.2630000000000001 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)