+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative stain EM map of the heterodimeric p110gamma-p84 complex | |||||||||
 マップデータ マップデータ | Negative stain EM ab initio model of the heterodimeric p110gamma-p84 complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | PI3K / p110 / PIK3CG / phosphoinositide / PIK3R6 / p84/p87 / 3-kinase / PIP3 / IMMUNE SYSTEM / TRANSFERASE | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
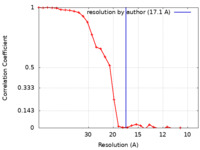
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 17.1 Å | |||||||||
 データ登録者 データ登録者 | Burke JE / Dalwadi U / Rathinaswamy MK / Yip CK / Nam SE | |||||||||
| 資金援助 |  カナダ, 1件 カナダ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2023 ジャーナル: Cell Rep / 年: 2023タイトル: Molecular basis for differential activation of p101 and p84 complexes of PI3Kγ by Ras and GPCRs. 著者: Manoj K Rathinaswamy / Meredith L Jenkins / Benjamin R Duewell / Xuxiao Zhang / Noah J Harris / John T Evans / Jordan T B Stariha / Udit Dalwadi / Kaelin D Fleming / Harish Ranga-Prasad / ...著者: Manoj K Rathinaswamy / Meredith L Jenkins / Benjamin R Duewell / Xuxiao Zhang / Noah J Harris / John T Evans / Jordan T B Stariha / Udit Dalwadi / Kaelin D Fleming / Harish Ranga-Prasad / Calvin K Yip / Roger L Williams / Scott D Hansen / John E Burke /    要旨: Class IB phosphoinositide 3-kinase (PI3Kγ) is activated in immune cells and can form two distinct complexes (p110γ-p84 and p110γ-p101), which are differentially activated by G protein-coupled ...Class IB phosphoinositide 3-kinase (PI3Kγ) is activated in immune cells and can form two distinct complexes (p110γ-p84 and p110γ-p101), which are differentially activated by G protein-coupled receptors (GPCRs) and Ras. Using a combination of X-ray crystallography, hydrogen deuterium exchange mass spectrometry (HDX-MS), electron microscopy, molecular modeling, single-molecule imaging, and activity assays, we identify molecular differences between p110γ-p84 and p110γ-p101 that explain their differential membrane recruitment and activation by Ras and GPCRs. The p110γ-p84 complex is dynamic compared with p110γ-p101. While p110γ-p101 is robustly recruited by Gβγ subunits, p110γ-p84 is weakly recruited to membranes by Gβγ subunits alone and requires recruitment by Ras to allow for Gβγ activation. We mapped two distinct Gβγ interfaces on p101 and the p110γ helical domain, with differences in the C-terminal domain of p84 and p101 conferring sensitivity of p110γ-p101 to Gβγ activation. Overall, our work provides key insight into the molecular basis for how PI3Kγ complexes are activated. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27738.map.gz emd_27738.map.gz | 1.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27738-v30.xml emd-27738-v30.xml emd-27738.xml emd-27738.xml | 16.8 KB 16.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27738_fsc.xml emd_27738_fsc.xml | 3.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27738.png emd_27738.png | 46 KB | ||
| Filedesc metadata |  emd-27738.cif.gz emd-27738.cif.gz | 4.8 KB | ||
| その他 |  emd_27738_half_map_1.map.gz emd_27738_half_map_1.map.gz emd_27738_half_map_2.map.gz emd_27738_half_map_2.map.gz | 1.3 MB 1.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27738 http://ftp.pdbj.org/pub/emdb/structures/EMD-27738 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27738 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27738 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27738_validation.pdf.gz emd_27738_validation.pdf.gz | 535.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27738_full_validation.pdf.gz emd_27738_full_validation.pdf.gz | 535.3 KB | 表示 | |
| XML形式データ |  emd_27738_validation.xml.gz emd_27738_validation.xml.gz | 8.2 KB | 表示 | |
| CIF形式データ |  emd_27738_validation.cif.gz emd_27738_validation.cif.gz | 10.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27738 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27738 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27738 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27738 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27738.map.gz / 形式: CCP4 / 大きさ: 1.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27738.map.gz / 形式: CCP4 / 大きさ: 1.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain EM ab initio model of the heterodimeric p110gamma-p84 complex | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.67 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_27738_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
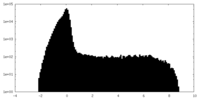
| 投影像・断面図 |
| ||||||||||||
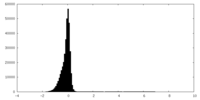
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_27738_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
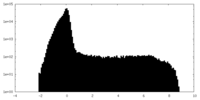
| 投影像・断面図 |
| ||||||||||||
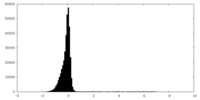
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of p110 gamma with p84
| 全体 | 名称: Ternary complex of p110 gamma with p84 |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of p110 gamma with p84
| 超分子 | 名称: Ternary complex of p110 gamma with p84 / タイプ: complex / ID: 1 / 親要素: 0 / 詳細: Both p110 and p84 subunits are from Homo sapiens |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.02 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8.5 構成要素:
詳細: Freshly prepared gel filtration buffer, filtered through 0.22 um filter and degassed | |||||||||||||||
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Formate 詳細: Negative stain EM samples prepares by adsorbing samples on grid for 15 second, followed by blotting of sample, 2 washes with water, 1 wash with stain, and a final 30 second soak in stain and blot. | |||||||||||||||
| グリッド | モデル: Homemade / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS | |||||||||||||||
| 詳細 | Specimen was a 1:1 molar ratio of p110g to p84, purified to homogeneity by gel filtration. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI SPIRIT |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI EAGLE (4k x 4k) / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 50 / 平均露光時間: 1.0 sec. / 平均電子線量: 25.0 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 6.3 mm / 最大 デフォーカス(公称値): 1.2 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 49000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC / ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai Spirit / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X