+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | eEF1A(GDP)aa-tRNA stalled on the rabbit 80S ribosome by ternatin-4 | |||||||||
 マップデータ マップデータ | RELION Refine3D, pixel calibrated | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Ribosome / eEF1A / ternatin / translation | |||||||||
| 生物種 |  | |||||||||
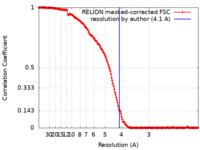
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.1 Å | |||||||||
 データ登録者 データ登録者 | Brown A / Shao S / Rundlet EJ / Juette MF / Carelli JD / Taunton J / Blanchard SC | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2022 ジャーナル: Elife / 年: 2022タイトル: Didemnin B and ternatin-4 differentially inhibit conformational changes in eEF1A required for aminoacyl-tRNA accommodation into mammalian ribosomes. 著者: Manuel F Juette / Jordan D Carelli / Emily J Rundlet / Alan Brown / Sichen Shao / Angelica Ferguson / Michael R Wasserman / Mikael Holm / Jack Taunton / Scott C Blanchard /   要旨: Rapid and accurate mRNA translation requires efficient codon-dependent delivery of the correct aminoacyl-tRNA (aa-tRNA) to the ribosomal A site. In mammals, this fidelity-determining reaction is ...Rapid and accurate mRNA translation requires efficient codon-dependent delivery of the correct aminoacyl-tRNA (aa-tRNA) to the ribosomal A site. In mammals, this fidelity-determining reaction is facilitated by the GTPase elongation factor-1 alpha (eEF1A), which escorts aa-tRNA as an eEF1A(GTP)-aa-tRNA ternary complex into the ribosome. The structurally unrelated cyclic peptides didemnin B and ternatin-4 bind to the eEF1A(GTP)-aa-tRNA ternary complex and inhibit translation but have different effects on protein synthesis in vitro and in vivo. Here, we employ single-molecule fluorescence imaging and cryogenic electron microscopy to determine how these natural products inhibit translational elongation on mammalian ribosomes. By binding to a common site on eEF1A, didemnin B and ternatin-4 trap eEF1A in an intermediate state of aa-tRNA selection, preventing eEF1A release and aa-tRNA accommodation on the ribosome. We also show that didemnin B and ternatin-4 exhibit distinct effects on the dynamics of aa-tRNA selection that inform on observed disparities in their inhibition efficacies and physiological impacts. These integrated findings underscore the value of dynamics measurements in assessing the mechanism of small-molecule inhibition and highlight potential of single-molecule methods to reveal how distinct natural products differentially impact the human translation mechanism. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27732.map.gz emd_27732.map.gz | 337.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27732-v30.xml emd-27732-v30.xml emd-27732.xml emd-27732.xml | 20.1 KB 20.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27732_fsc.xml emd_27732_fsc.xml | 16.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27732.png emd_27732.png | 140.7 KB | ||
| Filedesc metadata |  emd-27732.cif.gz emd-27732.cif.gz | 4.7 KB | ||
| その他 |  emd_27732_additional_1.map.gz emd_27732_additional_1.map.gz emd_27732_half_map_1.map.gz emd_27732_half_map_1.map.gz emd_27732_half_map_2.map.gz emd_27732_half_map_2.map.gz | 38.4 MB 338.2 MB 338 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27732 http://ftp.pdbj.org/pub/emdb/structures/EMD-27732 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27732 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27732 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27732_validation.pdf.gz emd_27732_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27732_full_validation.pdf.gz emd_27732_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_27732_validation.xml.gz emd_27732_validation.xml.gz | 23.8 KB | 表示 | |
| CIF形式データ |  emd_27732_validation.cif.gz emd_27732_validation.cif.gz | 30.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27732 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27732 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27732 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27732 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27732.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27732.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RELION Refine3D, pixel calibrated | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.045 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: RELION Postprocessed, pixel calibrated
| ファイル | emd_27732_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RELION Postprocessed, pixel calibrated | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
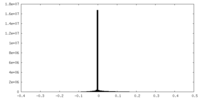
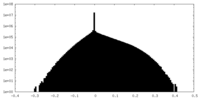
| 密度ヒストグラム |
-ハーフマップ: Pixel calibrated
| ファイル | emd_27732_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Pixel calibrated | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
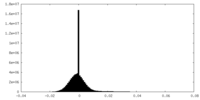
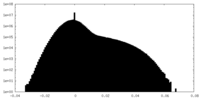
| 密度ヒストグラム |
-ハーフマップ: Pixel calibrated
| ファイル | emd_27732_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Pixel calibrated | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : eEF1A(GDP)aa-tRNA stalled on the rabbit 80S ribosome by ternatin-4
| 全体 | 名称: eEF1A(GDP)aa-tRNA stalled on the rabbit 80S ribosome by ternatin-4 |
|---|---|
| 要素 |
|
-超分子 #1: eEF1A(GDP)aa-tRNA stalled on the rabbit 80S ribosome by ternatin-4
| 超分子 | 名称: eEF1A(GDP)aa-tRNA stalled on the rabbit 80S ribosome by ternatin-4 タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #2: eEF1A(GDP)-aatRNA
| 超分子 | 名称: eEF1A(GDP)-aatRNA / タイプ: complex / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: Peptidyl-tRNA
| 超分子 | 名称: Peptidyl-tRNA / タイプ: complex / ID: 3 / 親要素: 1 / 詳細: P-site tRNA |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #4: Deacyl-tRNA
| 超分子 | 名称: Deacyl-tRNA / タイプ: complex / ID: 4 / 親要素: 1 / 詳細: E-site tRNA |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #5: 40S subunit
| 超分子 | 名称: 40S subunit / タイプ: complex / ID: 5 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #6: 60S subunit
| 超分子 | 名称: 60S subunit / タイプ: complex / ID: 6 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 構成要素:
詳細: Included 1 uM terntin-4 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/2 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 5 / 前処理 - タイプ: GLOW DISCHARGE | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK III | |||||||||||||||
| 詳細 | Pull downs of ribosomes from in vitro translation reactions of a transcript encoding 3x Flag-tagged KRas in rabbit reticulocyte lysate (RRL) in the presence of 1 uM ternatin-4. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: COUNTING / 実像数: 1422 / 平均露光時間: 1.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 9.200000000000001 µm 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 135000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)