+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SARS-CoV-2 Wuhan-hu-1-Spike-RBD bound to computationally engineered ACE2 mimetic CVD293 | ||||||||||||
 マップデータ マップデータ | SARS-CoV-2 Wuhan-hu-1-Spike-RBD bound to computationally engineered ACE2 mimetic CVD293 | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | SARS-CoV-2 / Spike / Receptor-binding domain / ACE2 receptor traps / ANTIVIRAL PROTEIN / VIRAL PROTEIN-ANTIVIRAL PROTEIN complex | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報immunoglobulin complex / positive regulation of amino acid transport / angiotensin-converting enzyme 2 / positive regulation of L-proline import across plasma membrane / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; 金属プロテアーゼ / angiotensin-mediated drinking behavior / regulation of systemic arterial blood pressure by renin-angiotensin / positive regulation of gap junction assembly / tryptophan transport / regulation of cardiac conduction ...immunoglobulin complex / positive regulation of amino acid transport / angiotensin-converting enzyme 2 / positive regulation of L-proline import across plasma membrane / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; 金属プロテアーゼ / angiotensin-mediated drinking behavior / regulation of systemic arterial blood pressure by renin-angiotensin / positive regulation of gap junction assembly / tryptophan transport / regulation of cardiac conduction / maternal process involved in female pregnancy / peptidyl-dipeptidase activity / regulation of vasoconstriction / transporter activator activity / Metabolism of Angiotensinogen to Angiotensins / carboxypeptidase activity / angiotensin maturation / Attachment and Entry / receptor-mediated endocytosis of virus by host cell / metallocarboxypeptidase activity / viral life cycle / positive regulation of cardiac muscle contraction / regulation of cytokine production / blood vessel diameter maintenance / negative regulation of smooth muscle cell proliferation / brush border membrane / negative regulation of ERK1 and ERK2 cascade / endocytic vesicle membrane / positive regulation of reactive oxygen species metabolic process / metallopeptidase activity / regulation of cell population proliferation / virus receptor activity / regulation of inflammatory response / endopeptidase activity / symbiont-mediated disruption of host tissue / Maturation of spike protein / adaptive immune response / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / viral translation / host extracellular space / symbiont-mediated-mediated suppression of host tetherin activity / Potential therapeutics for SARS / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell / membrane fusion / Attachment and Entry / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / host cell surface receptor binding / cilium / symbiont-mediated suppression of host innate immune response / apical plasma membrane / membrane raft / receptor ligand activity / endocytosis involved in viral entry into host cell / endoplasmic reticulum lumen / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / symbiont entry into host cell / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / cell surface / negative regulation of transcription by RNA polymerase II / extracellular space / extracellular exosome / extracellular region / zinc ion binding / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | ||||||||||||
 データ登録者 データ登録者 | QCRG Structural Biology Consortium / Remesh SG / Merz GE / Brilot AF / Chio U / Verba KA | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2023 ジャーナル: Structure / 年: 2023タイトル: Computational pipeline provides mechanistic understanding of Omicron variant of concern neutralizing engineered ACE2 receptor traps. 著者: Soumya G Remesh / Gregory E Merz / Axel F Brilot / Un Seng Chio / Alexandrea N Rizo / Thomas H Pospiech / Irene Lui / Mathew T Laurie / Jeff Glasgow / Chau Q Le / Yun Zhang / Devan Diwanji / ...著者: Soumya G Remesh / Gregory E Merz / Axel F Brilot / Un Seng Chio / Alexandrea N Rizo / Thomas H Pospiech / Irene Lui / Mathew T Laurie / Jeff Glasgow / Chau Q Le / Yun Zhang / Devan Diwanji / Evelyn Hernandez / Jocelyne Lopez / Hevatib Mehmood / Komal Ishwar Pawar / Sergei Pourmal / Amber M Smith / Fengbo Zhou / / Joseph DeRisi / Tanja Kortemme / Oren S Rosenberg / Anum Glasgow / Kevin K Leung / James A Wells / Kliment A Verba /  要旨: The SARS-CoV-2 Omicron variant, with 15 mutations in Spike receptor-binding domain (Spike-RBD), renders virtually all clinical monoclonal antibodies against WT SARS-CoV-2 ineffective. We recently ...The SARS-CoV-2 Omicron variant, with 15 mutations in Spike receptor-binding domain (Spike-RBD), renders virtually all clinical monoclonal antibodies against WT SARS-CoV-2 ineffective. We recently engineered the SARS-CoV-2 host entry receptor, ACE2, to tightly bind WT-RBD and prevent viral entry into host cells ("receptor traps"). Here we determine cryo-EM structures of our receptor traps in complex with stabilized Spike ectodomain. We develop a multi-model pipeline combining Rosetta protein modeling software and cryo-EM to allow interface energy calculations even at limited resolution and identify interface side chains that allow for high-affinity interactions between our ACE2 receptor traps and Spike-RBD. Our structural analysis provides a mechanistic rationale for the high-affinity (0.53-4.2 nM) binding of our ACE2 receptor traps to Omicron-RBD confirmed with biolayer interferometry measurements. Finally, we show that ACE2 receptor traps potently neutralize Omicron and Delta pseudotyped viruses, providing alternative therapeutic routes to combat this evolving virus. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27731.map.gz emd_27731.map.gz | 398.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27731-v30.xml emd-27731-v30.xml emd-27731.xml emd-27731.xml | 24.6 KB 24.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27731_fsc.xml emd_27731_fsc.xml | 16.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27731.png emd_27731.png | 114.3 KB | ||
| Filedesc metadata |  emd-27731.cif.gz emd-27731.cif.gz | 8.1 KB | ||
| その他 |  emd_27731_additional_1.map.gz emd_27731_additional_1.map.gz emd_27731_half_map_1.map.gz emd_27731_half_map_1.map.gz emd_27731_half_map_2.map.gz emd_27731_half_map_2.map.gz | 398.9 MB 391.3 MB 391.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27731 http://ftp.pdbj.org/pub/emdb/structures/EMD-27731 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27731 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27731 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27731_validation.pdf.gz emd_27731_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27731_full_validation.pdf.gz emd_27731_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_27731_validation.xml.gz emd_27731_validation.xml.gz | 24.6 KB | 表示 | |
| CIF形式データ |  emd_27731_validation.cif.gz emd_27731_validation.cif.gz | 32.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27731 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27731 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27731 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27731 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8dv2MC  8dv1C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27731.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27731.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | SARS-CoV-2 Wuhan-hu-1-Spike-RBD bound to computationally engineered ACE2 mimetic CVD293 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.008 Å | ||||||||||||||||||||||||||||||||||||
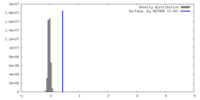
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: SARS-CoV-2 Wuhan-hu-1-Spike-RBD bound to computationally engineered ACE2 mimetic...
| ファイル | emd_27731_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | SARS-CoV-2 Wuhan-hu-1-Spike-RBD bound to computationally engineered ACE2 mimetic CVD293 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: SARS-CoV-2 Wuhan-hu-1-Spike-RBD bound to computationally engineered ACE2 mimetic...
| ファイル | emd_27731_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | SARS-CoV-2 Wuhan-hu-1-Spike-RBD bound to computationally engineered ACE2 mimetic CVD293 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: SARS-CoV-2 Wuhan-hu-1-Spike-RBD bound to computationally engineered ACE2 mimetic...
| ファイル | emd_27731_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | SARS-CoV-2 Wuhan-hu-1-Spike-RBD bound to computationally engineered ACE2 mimetic CVD293 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV-2 Wuhan-hu-1-Spike bound to linker variant of affinity m...
| 全体 | 名称: SARS-CoV-2 Wuhan-hu-1-Spike bound to linker variant of affinity matured ACE2 mimetic CVD293 |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 Wuhan-hu-1-Spike bound to linker variant of affinity m...
| 超分子 | 名称: SARS-CoV-2 Wuhan-hu-1-Spike bound to linker variant of affinity matured ACE2 mimetic CVD293 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #2: Spike glycoprotein
| 超分子 | 名称: Spike glycoprotein / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: Angiotensin-converting enzyme 2,Immunoglobulin gamma-1 heavy chai...
| 超分子 | 名称: Angiotensin-converting enzyme 2,Immunoglobulin gamma-1 heavy chain fusion タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|
-分子 #1: Spike glycoprotein
| 分子 | 名称: Spike glycoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 133.75325 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDG VYFASTEKSN IIRGWIFGTT LDSKTQSLLI VNNATNVVIK VCEFQFCNDP FLGVYYHKNN KSWMESEFRV Y SSANNCTF ...文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDG VYFASTEKSN IIRGWIFGTT LDSKTQSLLI VNNATNVVIK VCEFQFCNDP FLGVYYHKNN KSWMESEFRV Y SSANNCTF EYVSQPFLMD LEGKQGNFKN LREFVFKNID GYFKIYSKHT PINLVRDLPQ GFSALEPLVD LPIGINITRF QT LLALHRS YLTPGDSSSG WTAGAAAYYV GYLQPRTFLL KYNENGTITD AVDCALDPLS ETKCTLKSFT VEKGIYQTSN FRV QPTESI VRFPNITNLC PFGEVFNATR FASVYAWNRK RISNCVADYS VLYNSASFST FKCYGVSPTK LNDLCFTNVY ADSF VIRGD EVRQIAPGQT GKIADYNYKL PDDFTGCVIA WNSNNLDSKV GGNYNYLYRL FRKSNLKPFE RDISTEIYQA GSTPC NGVE GFNCYFPLQS YGFQPTNGVG YQPYRVVVLS FELLHAPATV CGPKKSTNLV KNKCVNFNFN GLTGTGVLTE SNKKFL PFQ QFGRDIADTT DAVRDPQTLE ILDITPCSFG GVSVITPGTN TSNQVAVLYQ DVNCTEVPVA IHADQLTPTW RVYSTGS NV FQTRAGCLIG AEHVNNSYEC DIPIGAGICA SYQTQTNSPG SASSVASQSI IAYTMSLGAE NSVAYSNNSI AIPTNFTI S VTTEILPVSM TKTSVDCTMY ICGDSTECSN LLLQYGSFCT QLNRALTGIA VEQDKNTQEV FAQVKQIYKT PPIKDFGGF NFSQILPDPS KPSKRSFIED LLFNKVTLAD AGFIKQYGDC LGDIAARDLI CAQKFNGLTV LPPLLTDEMI AQYTSALLAG TITSGWTFG AGAALQIPFA MQMAYRFNGI GVTQNVLYEN QKLIANQFNS AIGKIQDSLS STASALGKLQ DVVNQNAQAL N TLVKQLSS NFGAISSVLN DILSRLDPPE AEVQIDRLIT GRLQSLQTYV TQQLIRAAEI RASANLAATK MSECVLGQSK RV DFCGKGY HLMSFPQSAP HGVVFLHVTY VPAQEKNFTT APAICHDGKA HFPREGVFVS NGTHWFVTQR NFYEPQIITT DNT FVSGNC DVVIGIVNNT VYDPLQPELD SFKEELDKYF KNHTSPDVDL GDISGINASV VNIQKEIDRL NEVAKNLNES LIDL QELGK YEQ UniProtKB: Spike glycoprotein |
-分子 #2: Angiotensin-converting enzyme 2,Immunoglobulin gamma-1 heavy chai...
| 分子 | 名称: Angiotensin-converting enzyme 2,Immunoglobulin gamma-1 heavy chain fusion タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: angiotensin-converting enzyme 2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 113.86925 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QSTIEEQAKT FLDFFNIQAE DLFYQSSLAS WNYNTNITEE NVQNMNNAGD KWSAFLKEQS TLAQMYPLQE IQNLTVKLQL QALQQNGSS VLSEDKSKRL NTILNTMSTI YSTGKVCNPD NPQECLLLEP GLNEIMANSL DYNERLWAWE SWRSEVGKQL R PLYEEYVV ...文字列: QSTIEEQAKT FLDFFNIQAE DLFYQSSLAS WNYNTNITEE NVQNMNNAGD KWSAFLKEQS TLAQMYPLQE IQNLTVKLQL QALQQNGSS VLSEDKSKRL NTILNTMSTI YSTGKVCNPD NPQECLLLEP GLNEIMANSL DYNERLWAWE SWRSEVGKQL R PLYEEYVV LKNEMARANH YEDYGDYWRG DYEVNGVDGY DYSRGQLIED VEHTFEEIKP LYEHLHAYVR AKLMNAYPSY IS PIGCLPA HLLGDMWGRF WTNLYSLTVP FGQKPNIDVT DAMVDQAWDA QRIFKEAEKF FVSVGLPNMT QGFWENSMLT DPG NVQKAV CHPTAWDLGK GDFRILMCTK VTMDDFLTAH HEMGHIQYDM AYAAQPFLLR NGANEGFHEA VGEIMSLSAA TPKH LKSIG LLSPDFQEDN ETEINFLLKQ ALTIVGTLPF TYMLEKWRWM VFKGEIPKDQ WMKKWWEMKR EIVGVVEPVP HDETY CDPA SLFHVSNDYS FIRYYTRTLY QFQFQEALCQ AAKHEGPLHK CDISNSTEAG QKLFNMLRLG KSEPWTLALE NVVGAK NMN VRPLLNYFEP LFTWLKDQNK NSFVGWSTDW SPYADQSIKV RISLKSALGD KAYEWNDNEM YLFRSSVAYA MRQYFLK VK NQMILFGEED VRVANLKPRI SFNFFVTAPK NVSDIIPRTE VEKAIRMSRS RINDAFRLND NSLEFLGIQP TLGPPNQP P VSTSSGGGGE NLYFQSSGGG SGGGEPKSCD KTHTCPPCPA PELLGGPSVF LFPPKPKDTL MISRTPEVTC VVVDVSHED PEVKFNWYVD GVEVHNAKTK PREEQYNSTY RVVSVLTVLH QDWLNGKEYK CKVSNKALPA PIEKTISKAK GQPREPQVYT LPPSRDELT KNQVSLTCLV KGFYPSDIAV EWESNGQPEN NYKTTPPVLD SDGSFFLYSK LTVDKSRWQQ GNVFSCSVMH E ALHNHYTQ KSLSLSPGKG GGGSGLNDIF EAQKIEWHEG UniProtKB: Angiotensin-converting enzyme 2, Immunoglobulin gamma-1 heavy chain |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 7 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. / 前処理 - 雰囲気: AIR | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.0 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 3 / 実像数: 10685 / 平均電子線量: 66.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)