+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Helical reconstruction of A92E HIV capsid in complex with CPSF6 construct (filtered by local resolution) | |||||||||
 マップデータ マップデータ | Helical reconstruction of A92E HIV capsid in complex with CPSF6 construct (filtered by local resolution) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Complex / VIRAL PROTEIN | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | |||||||||
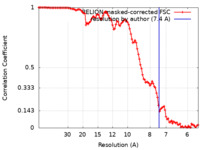
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 7.4 Å | |||||||||
 データ登録者 データ登録者 | Iqbal N / Asturias F / Kvaratskhelia M | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Prion-like low complexity regions enable avid virus-host interactions during HIV-1 infection. 著者: Guochao Wei / Naseer Iqbal / Valentine V Courouble / Ashwanth C Francis / Parmit K Singh / Arpa Hudait / Arun S Annamalai / Stephanie Bester / Szu-Wei Huang / Nikoloz Shkriabai / Lorenzo ...著者: Guochao Wei / Naseer Iqbal / Valentine V Courouble / Ashwanth C Francis / Parmit K Singh / Arpa Hudait / Arun S Annamalai / Stephanie Bester / Szu-Wei Huang / Nikoloz Shkriabai / Lorenzo Briganti / Reed Haney / Vineet N KewalRamani / Gregory A Voth / Alan N Engelman / Gregory B Melikyan / Patrick R Griffin / Francisco Asturias / Mamuka Kvaratskhelia /  要旨: Cellular proteins CPSF6, NUP153 and SEC24C play crucial roles in HIV-1 infection. While weak interactions of short phenylalanine-glycine (FG) containing peptides with isolated capsid hexamers have ...Cellular proteins CPSF6, NUP153 and SEC24C play crucial roles in HIV-1 infection. While weak interactions of short phenylalanine-glycine (FG) containing peptides with isolated capsid hexamers have been characterized, how these cellular factors functionally engage with biologically relevant mature HIV-1 capsid lattices is unknown. Here we show that prion-like low complexity regions (LCRs) enable avid CPSF6, NUP153 and SEC24C binding to capsid lattices. Structural studies revealed that multivalent CPSF6 assembly is mediated by LCR-LCR interactions, which are templated by binding of CPSF6 FG peptides to a subset of hydrophobic capsid pockets positioned along adjoining hexamers. In infected cells, avid CPSF6 LCR-mediated binding to HIV-1 cores is essential for functional virus-host interactions. The investigational drug lenacapavir accesses unoccupied hydrophobic pockets in the complex to potently impair HIV-1 inside the nucleus without displacing the tightly bound cellular cofactor from virus cores. These results establish previously undescribed mechanisms of virus-host interactions and antiviral action. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27619.map.gz emd_27619.map.gz | 27.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27619-v30.xml emd-27619-v30.xml emd-27619.xml emd-27619.xml | 15.6 KB 15.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27619_fsc.xml emd_27619_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27619.png emd_27619.png | 37.4 KB | ||
| Filedesc metadata |  emd-27619.cif.gz emd-27619.cif.gz | 5.1 KB | ||
| その他 |  emd_27619_half_map_1.map.gz emd_27619_half_map_1.map.gz emd_27619_half_map_2.map.gz emd_27619_half_map_2.map.gz | 80.5 MB 80.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27619 http://ftp.pdbj.org/pub/emdb/structures/EMD-27619 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27619 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27619 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27619_validation.pdf.gz emd_27619_validation.pdf.gz | 930.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27619_full_validation.pdf.gz emd_27619_full_validation.pdf.gz | 929.9 KB | 表示 | |
| XML形式データ |  emd_27619_validation.xml.gz emd_27619_validation.xml.gz | 17.8 KB | 表示 | |
| CIF形式データ |  emd_27619_validation.cif.gz emd_27619_validation.cif.gz | 23.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27619 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27619 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27619 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27619 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27619.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27619.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Helical reconstruction of A92E HIV capsid in complex with CPSF6 construct (filtered by local resolution) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.8 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half Map 1
| ファイル | emd_27619_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
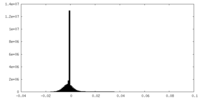
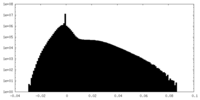
| 注釈 | Half Map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map 2
| ファイル | emd_27619_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
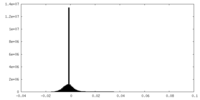
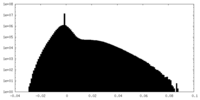
| 注釈 | Half Map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : HIV Capsid Protein Hexamer
| 全体 | 名称: HIV Capsid Protein Hexamer |
|---|---|
| 要素 |
|
-超分子 #1: HIV Capsid Protein Hexamer
| 超分子 | 名称: HIV Capsid Protein Hexamer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
-分子 #1: HIV Capsid
| 分子 | 名称: HIV Capsid / タイプ: protein_or_peptide / ID: 1 / 光学異性体: DEXTRO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: PIVQNLQGQM VHQCISPRTL NAWVKVVEEK AFSPEVIPMF SALSCGATPQ DLNTMLNTVG GHQAAMQMLK ETINEEAAEW DRLHPVHAGP IAPGQMREPR GSDIAGTTST LQEQIGWMTH NPPIPVGEIY KRWIILGLNK IVRMYSPTSI LDIRQGPKEP FRDYVDRFYK ...文字列: PIVQNLQGQM VHQCISPRTL NAWVKVVEEK AFSPEVIPMF SALSCGATPQ DLNTMLNTVG GHQAAMQMLK ETINEEAAEW DRLHPVHAGP IAPGQMREPR GSDIAGTTST LQEQIGWMTH NPPIPVGEIY KRWIILGLNK IVRMYSPTSI LDIRQGPKEP FRDYVDRFYK TLRAEQASQE VKNAATETLL VQNANPDCKT ILKALGPGAT LEEMMTACQG VGGPGHKARV L |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 1M NaCl, 50mM Tris pH 8, 0.2mM IP6 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4090 pixel / 撮影したグリッド数: 2 / 実像数: 4706 / 平均電子線量: 45.65 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 28000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 28000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)