+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The structure of S. epidermidis Cas10-Csm bound to target RNA | |||||||||
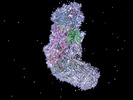
 マップデータ マップデータ | Cas10-Csm complex bound to crRNA and target RNA. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR / Cas10 / Type III / Csm / RNA BINDING PROTEIN / RNA BINDING PROTEIN-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Paraan M / Stagg SM / Dunkle JA | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: PLoS One / 年: 2023 ジャーナル: PLoS One / 年: 2023タイトル: The structure of a Type III-A CRISPR-Cas effector complex reveals conserved and idiosyncratic contacts to target RNA and crRNA among Type III-A systems. 著者: Mohammadreza Paraan / Mohamed Nasef / Lucy Chou-Zheng / Sarah A Khweis / Allyn J Schoeffler / Asma Hatoum-Aslan / Scott M Stagg / Jack A Dunkle /  要旨: Type III CRISPR-Cas systems employ multiprotein effector complexes bound to small CRISPR RNAs (crRNAs) to detect foreign RNA transcripts and elicit a complex immune response that leads to the ...Type III CRISPR-Cas systems employ multiprotein effector complexes bound to small CRISPR RNAs (crRNAs) to detect foreign RNA transcripts and elicit a complex immune response that leads to the destruction of invading RNA and DNA. Type III systems are among the most widespread in nature, and emerging interest in harnessing these systems for biotechnology applications highlights the need for detailed structural analyses of representatives from diverse organisms. We performed cryo-EM reconstructions of the Type III-A Cas10-Csm effector complex from S. epidermidis bound to an intact, cognate target RNA and identified two oligomeric states, a 276 kDa complex and a 318 kDa complex. 3.1 Å density for the well-ordered 276 kDa complex allowed construction of atomic models for the Csm2, Csm3, Csm4 and Csm5 subunits within the complex along with the crRNA and target RNA. We also collected small-angle X-ray scattering data which was consistent with the 276 kDa Cas10-Csm architecture we identified. Detailed comparisons between the S. epidermidis Cas10-Csm structure and the well-resolved bacterial (S. thermophilus) and archaeal (T. onnurineus) Cas10-Csm structures reveal differences in how the complexes interact with target RNA and crRNA which are likely to have functional ramifications. These structural comparisons shed light on the unique features of Type III-A systems from diverse organisms and will assist in improving biotechnologies derived from Type III-A effector complexes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27593.map.gz emd_27593.map.gz | 230.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27593-v30.xml emd-27593-v30.xml emd-27593.xml emd-27593.xml | 21.5 KB 21.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_27593.png emd_27593.png | 71.8 KB | ||
| Filedesc metadata |  emd-27593.cif.gz emd-27593.cif.gz | 6.5 KB | ||
| その他 |  emd_27593_half_map_1.map.gz emd_27593_half_map_1.map.gz emd_27593_half_map_2.map.gz emd_27593_half_map_2.map.gz | 226.8 MB 226.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27593 http://ftp.pdbj.org/pub/emdb/structures/EMD-27593 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27593 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27593 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27593_validation.pdf.gz emd_27593_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27593_full_validation.pdf.gz emd_27593_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_27593_validation.xml.gz emd_27593_validation.xml.gz | 15.9 KB | 表示 | |
| CIF形式データ |  emd_27593_validation.cif.gz emd_27593_validation.cif.gz | 18.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27593 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27593 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27593 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27593 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8do6MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27593.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27593.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cas10-Csm complex bound to crRNA and target RNA. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.846 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map A
| ファイル | emd_27593_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_27593_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cas10-Csm complex bound to target RNA
| 全体 | 名称: Cas10-Csm complex bound to target RNA |
|---|---|
| 要素 |
|
-超分子 #1: Cas10-Csm complex bound to target RNA
| 超分子 | 名称: Cas10-Csm complex bound to target RNA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌) |
| 分子量 | 理論値: 300 KDa |
-分子 #1: CRISPR system Cms endoribonuclease Csm3
| 分子 | 名称: CRISPR system Cms endoribonuclease Csm3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌)株: ATCC 35984 / RP62A |
| 分子量 | 理論値: 24.033975 KDa |
| 組換発現 | 生物種:  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌) |
| 配列 | 文字列: MYSKIKISGT IEVVTGLHIG GGGESSMIGA IDSPVVRDLQ TKLPIIPGSS IKGKMRNLLA KHFGLKMKQE SHNQDDERVL RLFGSSEKG NIQRARLQIS DAFFSEKTKE HFAQNDIAYT ETKFENTINR LTAVANPRQI ERVTRGSEFD FVFIYNVDEE S QVEDDFEN ...文字列: MYSKIKISGT IEVVTGLHIG GGGESSMIGA IDSPVVRDLQ TKLPIIPGSS IKGKMRNLLA KHFGLKMKQE SHNQDDERVL RLFGSSEKG NIQRARLQIS DAFFSEKTKE HFAQNDIAYT ETKFENTINR LTAVANPRQI ERVTRGSEFD FVFIYNVDEE S QVEDDFEN IEKAIHLLEN DYLGGGGTRG NGRIQFKDTN IETVVGEYDS TNLKIK UniProtKB: CRISPR system Cms endoribonuclease Csm3 |
-分子 #4: CRISPR system Cms protein Csm2
| 分子 | 名称: CRISPR system Cms protein Csm2 / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌) |
| 分子量 | 理論値: 16.809471 KDa |
| 組換発現 | 生物種:  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌) |
| 配列 | 文字列: MILAKTKSGK TIDLTFAHEV VKSNVKNVKD RKGKEKQVLF NGLTTSKLRN LMEQVNRLYT IAFNSNEDQL NEEFIDELEY LKIKFYYEA GREKSVDEFL KKTLMFPIID RVIKKESKKF FLDYCKYFEA LVAYAKYYQK ED UniProtKB: UNIPROTKB: A0A8G7QML1 |
-分子 #5: CRISPR system Cms protein Csm4
| 分子 | 名称: CRISPR system Cms protein Csm4 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌)株: ATCC 35984 / RP62A |
| 分子量 | 理論値: 34.551938 KDa |
| 組換発現 | 生物種:  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌) |
| 配列 | 文字列: MTLATKVFKL SFKTPVHFGK KRLSDGEMTI TADTLFSALF IETLQLGKDT DWLLNDLIIS DTFPYENELY YLPKPLIKID SKEEDNHKA FKKLKYVPVH HYNQYLNGEL SAEDATDLND IFNIGYFSLQ TKVSLIAQET DSSADSEPYS VGTFTFEPEA G LYFIAKGS ...文字列: MTLATKVFKL SFKTPVHFGK KRLSDGEMTI TADTLFSALF IETLQLGKDT DWLLNDLIIS DTFPYENELY YLPKPLIKID SKEEDNHKA FKKLKYVPVH HYNQYLNGEL SAEDATDLND IFNIGYFSLQ TKVSLIAQET DSSADSEPYS VGTFTFEPEA G LYFIAKGS EETLDHLNNI MTALQYSGLG GKRNAGYGQF EYEIINNQQL SKLLNQNGKH SILLSTAMAK KEEIESALKE AR YILTKRS GFVQSTNYSE MLVKKSDFYS FSSGSVFKNI FNGDIFNVGH NGKHPVYRYA KPLWLEV UniProtKB: CRISPR system Cms protein Csm4 |
-分子 #6: CRISPR system Cms protein Csm5
| 分子 | 名称: CRISPR system Cms protein Csm5 / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌)株: ATCC 35984 / RP62A |
| 分子量 | 理論値: 39.449125 KDa |
| 組換発現 | 生物種:  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌) |
| 配列 | 文字列: MTIKNYEVVI KTLGPIHIGS GQVMKKQDYI YDFYNSKVYM INGNKLVKFL KRKNLLYTYQ NFLRYPPKNP RENGLKDYLD AQNVKQSEW EAFVSYSEKV NQGKKYGNTR PKPLNDLHLM VRDGQNKVYL PGSSIKGAIK TTLVSKYNNE KNKDIYSKIK V SDSKPIDE ...文字列: MTIKNYEVVI KTLGPIHIGS GQVMKKQDYI YDFYNSKVYM INGNKLVKFL KRKNLLYTYQ NFLRYPPKNP RENGLKDYLD AQNVKQSEW EAFVSYSEKV NQGKKYGNTR PKPLNDLHLM VRDGQNKVYL PGSSIKGAIK TTLVSKYNNE KNKDIYSKIK V SDSKPIDE SNLAIYQKID INKSEKSMPL YRECIDVNTE IKFKLTIEDE IYSINEIEQS IQDFYKNYYD KWLVGFKETK GG RRFALEG GIPDVLNQNI LFLGAGTGFV SKTTHYQLKN RKQAKQDSFE ILTKKFRGTY GKMKEIPSNV PVALKGTTNQ SRH TSYQQG MCKVSFQELN NEVL UniProtKB: CRISPR system Cms protein Csm5 |
-分子 #2: crRNA
| 分子 | 名称: crRNA / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌) |
| 分子量 | 理論値: 11.895168 KDa |
| 配列 | 文字列: ACGAGAACAC GUAUGCCGAA GUAUAUAAAU CAUCAGU |
-分子 #3: Target RNA
| 分子 | 名称: Target RNA / タイプ: rna / ID: 3 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌) |
| 分子量 | 理論値: 13.599979 KDa |
| 配列 | 文字列: CUUUGUACUG AUGAUUUAUA UACUUCGGCA UACGUUCUCU AAA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 構成要素:
| ||||||
|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 78.0 K / 最高: 92.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 5000 / 平均露光時間: 1.0 sec. / 平均電子線量: 44.7 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 1.5 µm / 最小 デフォーカス(補正後): 1.0 µm / 倍率(補正後): 81000 / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 0.001 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)