+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of SARS-CoV-2 Gamma (P.1) spike protein in complex with Fab 4A8 | |||||||||
 マップデータ マップデータ | structure of SARS-CoV-2 Gamma (P.1) spike protein in complex with Fab 4A8 | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
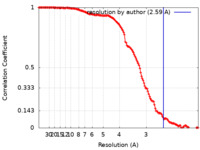
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.59 Å | |||||||||
 データ登録者 データ登録者 | Zhu X / Mannar D / Saville JW / Srivastava SS / Berezuk AM / Zhou S / Tuttle KS / Subramaniam S | |||||||||
| 資金援助 |  カナダ, 2件 カナダ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: SARS-CoV-2 variants of concern: spike protein mutational analysis and epitope for broad neutralization. 著者: Dhiraj Mannar / James W Saville / Zehua Sun / Xing Zhu / Michelle M Marti / Shanti S Srivastava / Alison M Berezuk / Steven Zhou / Katharine S Tuttle / Michele D Sobolewski / Andrew Kim / ...著者: Dhiraj Mannar / James W Saville / Zehua Sun / Xing Zhu / Michelle M Marti / Shanti S Srivastava / Alison M Berezuk / Steven Zhou / Katharine S Tuttle / Michele D Sobolewski / Andrew Kim / Benjamin R Treat / Priscila Mayrelle Da Silva Castanha / Jana L Jacobs / Simon M Barratt-Boyes / John W Mellors / Dimiter S Dimitrov / Wei Li / Sriram Subramaniam /   要旨: Mutations in the spike glycoproteins of SARS-CoV-2 variants of concern have independently been shown to enhance aspects of spike protein fitness. Here, we describe an antibody fragment (V ab6) that ...Mutations in the spike glycoproteins of SARS-CoV-2 variants of concern have independently been shown to enhance aspects of spike protein fitness. Here, we describe an antibody fragment (V ab6) that neutralizes all major variants including the recently emerged BA.1 and BA.2 Omicron subvariants, with a unique mode of binding revealed by cryo-EM studies. Further, we provide a comparative analysis of the mutational effects within previously emerged variant spikes and identify the structural role of mutations within the NTD and RBD in evading antibody neutralization. Our analysis shows that the highly mutated Gamma N-terminal domain exhibits considerable structural rearrangements, partially explaining its decreased neutralization by convalescent sera. Our results provide mechanistic insights into the structural, functional, and antigenic consequences of SARS-CoV-2 spike mutations and highlight a spike protein vulnerability that may be exploited to achieve broad protection against circulating variants. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27513.map.gz emd_27513.map.gz | 123 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27513-v30.xml emd-27513-v30.xml emd-27513.xml emd-27513.xml | 18.3 KB 18.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27513_fsc.xml emd_27513_fsc.xml | 13.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27513.png emd_27513.png | 66 KB | ||
| その他 |  emd_27513_half_map_1.map.gz emd_27513_half_map_1.map.gz emd_27513_half_map_2.map.gz emd_27513_half_map_2.map.gz | 226.3 MB 226.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27513 http://ftp.pdbj.org/pub/emdb/structures/EMD-27513 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27513 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27513 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27513_validation.pdf.gz emd_27513_validation.pdf.gz | 922.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27513_full_validation.pdf.gz emd_27513_full_validation.pdf.gz | 922.1 KB | 表示 | |
| XML形式データ |  emd_27513_validation.xml.gz emd_27513_validation.xml.gz | 19.4 KB | 表示 | |
| CIF形式データ |  emd_27513_validation.cif.gz emd_27513_validation.cif.gz | 25.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27513 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27513 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27513 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27513 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8dliC  8dljC  8dlkC  8dllC  8dlmC  8dlnC  8dloC  8dlpC  8dlqC  8dlrC  8dlsC  8dltC  8dluC  8dlvC  8dlwC  8dlxC  8dlyC  8dlzC  8dm0C C: 同じ文献を引用 ( |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27513.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27513.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | structure of SARS-CoV-2 Gamma (P.1) spike protein in complex with Fab 4A8 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half Map 1
| ファイル | emd_27513_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map 2
| ファイル | emd_27513_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV-2 Gamma (P.1) spike protein in complex with Fab 4A8
| 全体 | 名称: SARS-CoV-2 Gamma (P.1) spike protein in complex with Fab 4A8 |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 Gamma (P.1) spike protein in complex with Fab 4A8
| 超分子 | 名称: SARS-CoV-2 Gamma (P.1) spike protein in complex with Fab 4A8 タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #2: SARS-CoV-2 Gamma (P.1) spike protein
| 超分子 | 名称: SARS-CoV-2 Gamma (P.1) spike protein / タイプ: complex / キメラ: Yes / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: Fab 4A8
| 超分子 | 名称: Fab 4A8 / タイプ: complex / キメラ: Yes / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: SARS-CoV-2 Gamma (P.1) spike protein
| 分子 | 名称: SARS-CoV-2 Gamma (P.1) spike protein / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: MFVFLVLLPL VSSQCVNFTN RTQLPSAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDGV YFASTEKSNI IRGWIFGTTL DSKTQSLLIV NNATNVVIKV CEFQFCNYPF LGVYYHKNNK SWMESEFRVY SSANNCTFEY ...文字列: MFVFLVLLPL VSSQCVNFTN RTQLPSAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDGV YFASTEKSNI IRGWIFGTTL DSKTQSLLIV NNATNVVIKV CEFQFCNYPF LGVYYHKNNK SWMESEFRVY SSANNCTFEY VSQPFLMDLE GKQGNFKNLS EFVFKNIDGY FKIYSKHTPI NLVRDLPQGF SALEPLVDLP IGINITRFQT LLALHRSYLT PGDSSSGWTA GAAAYYVGYL QPRTFLLKYN ENGTITDAVD CALDPLSETK CTLKSFTVEK GIYQTSNFRV QPTESIVRFP NITNLCPFGE VFNATRFASV YAWNRKRISN CVADYSVLYN SASFSTFKCY GVSPTKLNDL CFTNVYADSF VIRGDEVRQI APGQTGTIAD YNYKLPDDFT GCVIAWNSNN LDSKVGGNYN YLYRLFRKSN LKPFERDIST EIYQAGSTPC NGVKGFNCYF PLQSYGFQPT YGVGYQPYRV VVLSFELLHA PATVCGPKKS TNLVKNKCVN FNFNGLTGTG VLTESNKKFL PFQQFGRDIA DTTDAVRDPQ TLEILDITPC SFGGVSVITP GTNTSNQVAV LYQGVNCTEV PVAIHADQLT PTWRVYSTGS NVFQTRAGCL IGAEYVNNSY ECDIPIGAGI CASYQTQTNS PGSASSVASQ SIIAYTMSLG AENSVAYSNN SIAIPTNFTI SVTTEILPVS MTKTSVDCTM YICGDSTECS NLLLQYGSFC TQLNRALTGI AVEQDKNTQE VFAQVKQIYK TPPIKDFGGF NFSQILPDPS KPSKRSPIED LLFNKVTLAD AGFIKQYGDC LGDIAARDLI CAQKFNGLTV LPPLLTDEMI AQYTSALLAG TITSGWTFGA GPALQIPFPM QMAYRFNGIG VTQNVLYENQ KLIANQFNSA IGKIQDSLSS TPSALGKLQD VVNQNAQALN TLVKQLSSNF GAISSVLNDI LSRLDPPEAE VQIDRLITGR LQSLQTYVTQ QLIRAAEIRA SANLAAIKMS ECVLGQSKRV DFCGKGYHLM SFPQSAPHGV VFLHVTYVPA QEKNFTTAPA ICHDGKAHFP REGVFVSNGT HWFVTQRNFY EPQIITTDNT FVSGNCDVVI GIVNNTVYDP LQPELDSFKE ELDKYFKNHT SPDVDLGDIS GINASFVNIQ KEIDRLNEVA KNLNESLIDL QELGKYEQGS GYIPEAPRDG QAYVRKDGEW VLLSTFLGRS LEVLFQGPGH HHHHHHHSAW SHPQFEKGGG SGGGGSGGSA WSHPQFEK |
-分子 #2: Fab 4A8 heavy chain
| 分子 | 名称: Fab 4A8 heavy chain / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDWTWRVFCL LAVAPGAHSE VQLVESGAEV KKPGASVKVS CKVSGYTLTE LSMHWVRQAP GKGLEWMGGF DPEDGETMYA QKFQGRVTMT EDTSTDTAYM ELSSLRSEDT AVYYCATSTA VAGTPDLFDY YYGMDVWGQG TTVTVSSAST KGPSVFPLAP SSKSTSGGTA ...文字列: MDWTWRVFCL LAVAPGAHSE VQLVESGAEV KKPGASVKVS CKVSGYTLTE LSMHWVRQAP GKGLEWMGGF DPEDGETMYA QKFQGRVTMT EDTSTDTAYM ELSSLRSEDT AVYYCATSTA VAGTPDLFDY YYGMDVWGQG TTVTVSSAST KGPSVFPLAP SSKSTSGGTA ALGCLVKDYF PEPVTVSWNS GALTSGVHTF PAVLQSSGLY SLSSVVTVPS SSLGTQTYIC NVNHKPSNTK VDKKVEPKSC GSHHHHHH |
-分子 #3: Fab 4A8 light chain
| 分子 | 名称: Fab 4A8 light chain / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MVLQTQVFIS LLLWISGAYG EIVMTQSPLS SPVTLGQPAS ISCRSSQSLV HSDGNTYLSW LQQRPGQPPR LLIYKISNRF SGVPDRFSGS GAGTDFTLKI SRVEAEDVGV YYCTQATQFP YTFGQGTKVD IKGQPKANPT VTLFPPSSEE LQANKATLVC LISDFYPGAV ...文字列: MVLQTQVFIS LLLWISGAYG EIVMTQSPLS SPVTLGQPAS ISCRSSQSLV HSDGNTYLSW LQQRPGQPPR LLIYKISNRF SGVPDRFSGS GAGTDFTLKI SRVEAEDVGV YYCTQATQFP YTFGQGTKVD IKGQPKANPT VTLFPPSSEE LQANKATLVC LISDFYPGAV TVAWKADGSP VKAGVETTKP SKQSNNKYAA SSYLSLTPEQ WKSHRSYSCQ VTHEGSTVEK TVAPTECS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)