+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2717 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Electron cryo-microscopy of T7 bacteriophage tail after DNA ejection | |||||||||
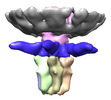
 マップデータ マップデータ | Reconstruction of T7 tail after DNA ejection | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | bacteriophage / virus infection / DNA ejection / tail | |||||||||
| 生物種 |   Enterobacteria phage T7 (ファージ) Enterobacteria phage T7 (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 20.0 Å | |||||||||
 データ登録者 データ登録者 | Gonzalez-Garcia VA / Pulido-Cid M / Garcia-Doval C / van Raaij MJ / Martin-Benito J / Cuervo A / Carrascosa JL | |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2015 ジャーナル: J Biol Chem / 年: 2015タイトル: Conformational changes leading to T7 DNA delivery upon interaction with the bacterial receptor. 著者: Verónica A González-García / Mar Pulido-Cid / Carmela Garcia-Doval / Rebeca Bocanegra / Mark J van Raaij / Jaime Martín-Benito / Ana Cuervo / José L Carrascosa /  要旨: The majority of bacteriophages protect their genetic material by packaging the nucleic acid in concentric layers to an almost crystalline concentration inside protein shells (capsid). This highly ...The majority of bacteriophages protect their genetic material by packaging the nucleic acid in concentric layers to an almost crystalline concentration inside protein shells (capsid). This highly condensed genome also has to be efficiently injected into the host bacterium in a process named ejection. Most phages use a specialized complex (often a tail) to deliver the genome without disrupting cell integrity. Bacteriophage T7 belongs to the Podoviridae family and has a short, non-contractile tail formed by a tubular structure surrounded by fibers. Here we characterize the kinetics and structure of bacteriophage T7 DNA delivery process. We show that T7 recognizes lipopolysaccharides (LPS) from Escherichia coli rough strains through the fibers. Rough LPS acts as the main phage receptor and drives DNA ejection in vitro. The structural characterization of the phage tail after ejection using cryo-electron microscopy (cryo-EM) and single particle reconstruction methods revealed the major conformational changes needed for DNA delivery at low resolution. Interaction with the receptor causes fiber tilting and opening of the internal tail channel by untwisting the nozzle domain, allowing release of DNA and probably of the internal head proteins. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2717.map.gz emd_2717.map.gz | 784.8 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2717-v30.xml emd-2717-v30.xml emd-2717.xml emd-2717.xml | 10.5 KB 10.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2717.png emd_2717.png | 89.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2717 http://ftp.pdbj.org/pub/emdb/structures/EMD-2717 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2717 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2717 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2717_validation.pdf.gz emd_2717_validation.pdf.gz | 236.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2717_full_validation.pdf.gz emd_2717_full_validation.pdf.gz | 235.5 KB | 表示 | |
| XML形式データ |  emd_2717_validation.xml.gz emd_2717_validation.xml.gz | 4.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2717 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2717 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2717 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2717 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2717.map.gz / 形式: CCP4 / 大きさ: 825.2 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2717.map.gz / 形式: CCP4 / 大きさ: 825.2 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of T7 tail after DNA ejection | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5.5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
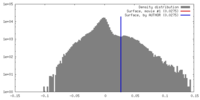
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : T7 tail after DNA ejection
| 全体 | 名称: T7 tail after DNA ejection |
|---|---|
| 要素 |
|
-超分子 #1000: T7 tail after DNA ejection
| 超分子 | 名称: T7 tail after DNA ejection / タイプ: sample / ID: 1000 集合状態: Oligomer build of 5 components: gp8 (12mer), gp11 (12mer), gp12 (6mer), gp17 (3mer), gp10 (n/a) Number unique components: 5 |
|---|
-分子 #1: gp8
| 分子 | 名称: gp8 / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage T7 (ファージ) Enterobacteria phage T7 (ファージ) |
-分子 #2: gp11
| 分子 | 名称: gp11 / タイプ: protein_or_peptide / ID: 2 / コピー数: 12 / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage T7 (ファージ) Enterobacteria phage T7 (ファージ) |
-分子 #3: gp12
| 分子 | 名称: gp12 / タイプ: protein_or_peptide / ID: 3 / コピー数: 6 / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage T7 (ファージ) Enterobacteria phage T7 (ファージ) |
-分子 #4: gp17
| 分子 | 名称: gp17 / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage T7 (ファージ) Enterobacteria phage T7 (ファージ) |
-分子 #5: gp10
| 分子 | 名称: gp10 / タイプ: protein_or_peptide / ID: 5 / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage T7 (ファージ) Enterobacteria phage T7 (ファージ) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.8 詳細: 50 mM TrisHCl, 10 mM MgCl2, 100 mM NaCl, 0.25mg/ml LPS from E. coli EH 100 Ra mutant |
| グリッド | 詳細: R2/2 Quantifoil coated with a thin carbon layer |
| 凍結 | 凍結剤: ETHANE / 装置: LEICA EM CPC 手法: Samples were applied to grids for 1 minute, blotted, and plunged into liquid ethane. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 温度 | 平均: 93 K |
| 日付 | 2012年10月11日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GATAN K2 (4k x 4k) / デジタル化 - サンプリング間隔: 2 µm / 実像数: 463 / 平均電子線量: 10 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.26 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 108696 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | The particles were manually selected |
|---|---|
| CTF補正 | 詳細: Each micrograph |
| 最終 再構成 | 想定した対称性 - 点群: C6 (6回回転対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 20.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: XMIPP, EMAN / 詳細: Final map was calculated from 3 averaged data sets / 使用した粒子像数: 1781 |
| 最終 2次元分類 | クラス数: 4 |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)