+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | VWF tubule derived from dimeric D1-A2 | ||||||||||||
 マップデータ マップデータ | Map output from Relion refinement (half maps included) autosharpened in Phenix. | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | VWF / tubule / Blood clotting | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
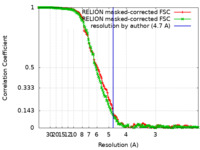
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 4.7 Å | ||||||||||||
 データ登録者 データ登録者 | Anderson JR / Li J / Springer TA / Brown A | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Blood / 年: 2022 ジャーナル: Blood / 年: 2022タイトル: Structures of VWF tubules before and after concatemerization reveal a mechanism of disulfide bond exchange. 著者: Jacob R Anderson / Jing Li / Timothy A Springer / Alan Brown /  要旨: von Willebrand factor (VWF) is an adhesive glycoprotein that circulates in the blood as disulfide-linked concatemers and functions in primary hemostasis. The loss of long VWF concatemers is ...von Willebrand factor (VWF) is an adhesive glycoprotein that circulates in the blood as disulfide-linked concatemers and functions in primary hemostasis. The loss of long VWF concatemers is associated with the excessive bleeding of type 2A von Willebrand disease (VWD). Formation of the disulfide bonds that concatemerize VWF requires VWF to self-associate into helical tubules, yet how the helical tubules template intermolecular disulfide bonds is not known. Here, we report electron cryomicroscopy (cryo-EM) structures of VWF tubules before and after intermolecular disulfide bond formation. The structures provide evidence that VWF tubulates through a charge-neutralization mechanism and that the A1 domain enhances tubule length by crosslinking successive helical turns. In addition, the structures reveal disulfide states before and after disulfide bond-mediated concatemerization. The structures and proposed assembly mechanism provide a foundation to rationalize VWD-causing mutations. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27158.map.gz emd_27158.map.gz | 197.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27158-v30.xml emd-27158-v30.xml emd-27158.xml emd-27158.xml | 18.3 KB 18.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27158_fsc.xml emd_27158_fsc.xml emd_27158_fsc_2.xml emd_27158_fsc_2.xml | 13.6 KB 13.7 KB | 表示 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27158.png emd_27158.png | 121.6 KB | ||
| マスクデータ |  emd_27158_msk_1.map emd_27158_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-27158.cif.gz emd-27158.cif.gz | 6.3 KB | ||
| その他 |  emd_27158_half_map_1.map.gz emd_27158_half_map_1.map.gz emd_27158_half_map_2.map.gz emd_27158_half_map_2.map.gz | 171.4 MB 171.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27158 http://ftp.pdbj.org/pub/emdb/structures/EMD-27158 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27158 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27158 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27158_validation.pdf.gz emd_27158_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27158_full_validation.pdf.gz emd_27158_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_27158_validation.xml.gz emd_27158_validation.xml.gz | 21.3 KB | 表示 | |
| CIF形式データ |  emd_27158_validation.cif.gz emd_27158_validation.cif.gz | 28 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27158 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27158 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27158 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27158 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27158.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27158.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map output from Relion refinement (half maps included) autosharpened in Phenix. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_27158_msk_1.map emd_27158_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: 2 of 2 half maps of D1-A2 VWF
| ファイル | emd_27158_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 2 of 2 half maps of D1-A2 VWF | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: 1 of 2 half maps of D1-A2 VWF
| ファイル | emd_27158_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 1 of 2 half maps of D1-A2 VWF | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Von Willebrand Factor tubule derived from dimeric D1-A2
| 全体 | 名称: Von Willebrand Factor tubule derived from dimeric D1-A2 |
|---|---|
| 要素 |
|
-超分子 #1: Von Willebrand Factor tubule derived from dimeric D1-A2
| 超分子 | 名称: Von Willebrand Factor tubule derived from dimeric D1-A2 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 14.0 kDa/nm |
-分子 #1: Von Willebrand Factor (D1-A2)
| 分子 | 名称: Von Willebrand Factor (D1-A2) / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MIPARFAGVL LALALILPGT LCAEGTRGRS STARCSLFGS DFVNTFDGSM YSFAGYCSYL LAGGCQKRSF SIIGDFQNGK RVSLSVYLGE FFDIHLFVNG TVTQGDQRVS MPYASKGLYL ETEAGYYKLS GEAYGFVARI DGSGNFQVLL SDRYFNKTCG LCGNFNIFAE ...文字列: MIPARFAGVL LALALILPGT LCAEGTRGRS STARCSLFGS DFVNTFDGSM YSFAGYCSYL LAGGCQKRSF SIIGDFQNGK RVSLSVYLGE FFDIHLFVNG TVTQGDQRVS MPYASKGLYL ETEAGYYKLS GEAYGFVARI DGSGNFQVLL SDRYFNKTCG LCGNFNIFAE DDFMTQEGTL TSDPYDFANS WALSSGEQWC ERASPPSSSC NISSGEMQKG LWEQCQLLKS TSVFARCHPL VDPEPFVALC EKTLCECAGG LECACPALLE YARTCAQEGM VLYGWTDHSA CSPVCPAGME YRQCVSPCAR TCQSLHINEM CQERCVDGCS CPEGQLLDEG LCVESTECPC VHSGKRYPPG TSLSRDCNTC ICRNSQWICS NEECPGECLV TGQSHFKSFD NRYFTFSGIC QYLLARDCQD HSFSIVIETV QCADDRDAVC TRSVTVRLPG LHNSLVKLKH GAGVAMDGQD VQLPLLKGDL RIQHTVTASV RLSYGEDLQM DWDGRGRLLV KLSPVYAGKT CGLCGNYNGN QGDDFLTPSG LAEPRVEDFG NAWKLHGDCQ DLQKQHSDPC ALNPRMTRFS EEACAVLTSP TFEACHRAVS PLPYLRNCRY DVCSCSDGRE CLCGALASYA AACAGRGVRV AWREPGRCEL NCPKGQVYLQ CGTPCNLTCR SLSYPDEECN EACLEGCFCP PGLYMDERGD CVPKAQCPCY YDGEIFQPED IFSDHHTMCY CEDGFMHCTM SGVPGSLLPD AVLSSPLSHA SASLSCRPPM VKLVCPADNL RAEGLECAKT CQNYDLECMS MGCVSGCLCP PGMVRHENRC VALERCPCFH QGKEYAPGET VKIGCNTCVC RDRKWNCTDH VCDATCSTIG MAHYLTFDGL KYLFPGECQY VLVQDYCGSN PGTFRILVGN KGCSHPSVKC KKRVTILVEG GEIELFDGEV NVKRPMKDET HFEVVESGRY IILLLGKALS VVWDRHLSIS VVLKQTYQEK VCGLCGNFDG IQNNDLTSSN LQVEEDPVDF GNSWKVSSQC ADTRKVPLDS SPATCHNNIM KQTMVDSSCR ILTSDVFQDC NKLVDPEPYL DVCIYDTCSC ESIGDCACFC DTIAAYAHVC AQHGKVVTWR TATLCPQSCE ERNLRENGYE CEWRYNSCAP ACQVTCQHPE PLACPVQCVE GCHAHCPPGK ILDELLQTCV DPEDCPVCEV AGRRFASGKK VTLNPSDPEH CQICHCDVVN LTCEACQEPG GLVVPPTDAP VSPTTLYVED ISEPPLHDFY CSRLLDLVFL LDGSSRLSEA EFEVLKAFVV DMMERLRISQ KWVRVAVVEY HDGSHAYIGL KDRKRPSELR RIASQVKYAG SQVASTSEVL KYTLFQIFSK IDRPEASRIA LLLMASQEPQ RMSRNFVRYV QGLKKKKVIV IPVGIGPHAN LKQIRLIEKQ APENKAFVLS SVDELEQQRD EIVSYLCDLA PEAPPPTLPP DMAQVTVGPG LLGVSTLGPK RNSMVLDVAF VLEGSDKIGE ADFNRSKEFM EEVIQRMDVG QDSIHVTVLQ YSYMVTVEYP FSEAQSKGDI LQRVREIRYQ GGNRTNTGLA LRYLSDHSFL VSQGDREQAP NLVYMVTGNP ASDEIKRLPG DIQVVPIGVG PNANVQELER IGWPNAPILI QDFETLPREA PDLVLQRCCS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 5.2 構成要素:
詳細: 100 mM Sodium Cacodylate at pH 5.2, 10 mM CaCl2, and 100 mM NaCl. | ||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 2 | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 55.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)