+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 1F8 mAb in complex with the computationally optimized broadly reactive H1 influenza hemagglutinin P1 | |||||||||
 マップデータ マップデータ | CryoEM map of COBRA P1 HA in complex with 1F8 mAb | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Hemagglutinin / influenza virus / COBRA / monoclonal antibody / IMMUNE SYSTEM / Viral Protein-IMMUNE SYSTEM complex | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Influenza A virus (A型インフルエンザウイルス) / Influenza A virus (A型インフルエンザウイルス) /  | |||||||||
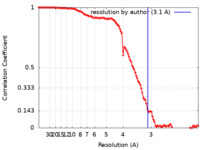
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Dzimianski JV / DuBois RM / Ward A | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2023 ジャーナル: Commun Biol / 年: 2023タイトル: Structural insights into the broad protection against H1 influenza viruses by a computationally optimized hemagglutinin vaccine. 著者: John V Dzimianski / Julianna Han / Giuseppe A Sautto / Sara M O'Rourke / Joseph M Cruz / Spencer R Pierce / Jeffrey W Ecker / Michael A Carlock / Kaito A Nagashima / Jarrod J Mousa / Ted M ...著者: John V Dzimianski / Julianna Han / Giuseppe A Sautto / Sara M O'Rourke / Joseph M Cruz / Spencer R Pierce / Jeffrey W Ecker / Michael A Carlock / Kaito A Nagashima / Jarrod J Mousa / Ted M Ross / Andrew B Ward / Rebecca M DuBois /  要旨: Influenza virus poses an ongoing human health threat with pandemic potential. Due to mutations in circulating strains, formulating effective vaccines remains a challenge. The use of computationally ...Influenza virus poses an ongoing human health threat with pandemic potential. Due to mutations in circulating strains, formulating effective vaccines remains a challenge. The use of computationally optimized broadly reactive antigen (COBRA) hemagglutinin (HA) proteins is a promising vaccine strategy to protect against a wide range of current and future influenza viruses. Though effective in preclinical studies, the mechanistic basis driving the broad reactivity of COBRA proteins remains to be elucidated. Here, we report the crystal structure of the COBRA HA termed P1 and identify antigenic and glycosylation properties that contribute to its immunogenicity. We further report the cryo-EM structure of the P1-elicited broadly neutralizing antibody 1F8 bound to COBRA P1, revealing 1F8 to recognize an atypical receptor binding site epitope via an unexpected mode of binding. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26983.map.gz emd_26983.map.gz | 118.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26983-v30.xml emd-26983-v30.xml emd-26983.xml emd-26983.xml | 23.6 KB 23.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26983_fsc.xml emd_26983_fsc.xml | 11.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26983.png emd_26983.png | 74.9 KB | ||
| マスクデータ |  emd_26983_msk_1.map emd_26983_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-26983.cif.gz emd-26983.cif.gz | 7.6 KB | ||
| その他 |  emd_26983_half_map_1.map.gz emd_26983_half_map_1.map.gz emd_26983_half_map_2.map.gz emd_26983_half_map_2.map.gz | 116 MB 116 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26983 http://ftp.pdbj.org/pub/emdb/structures/EMD-26983 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26983 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26983 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26983_validation.pdf.gz emd_26983_validation.pdf.gz | 977.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26983_full_validation.pdf.gz emd_26983_full_validation.pdf.gz | 977.4 KB | 表示 | |
| XML形式データ |  emd_26983_validation.xml.gz emd_26983_validation.xml.gz | 18.8 KB | 表示 | |
| CIF形式データ |  emd_26983_validation.cif.gz emd_26983_validation.cif.gz | 23.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26983 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26983 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26983 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26983 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26983.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26983.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM map of COBRA P1 HA in complex with 1F8 mAb | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.045 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_26983_msk_1.map emd_26983_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: CryoEM half map B of COBRA P1 HA in complex with 1F8 mAb
| ファイル | emd_26983_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM half map B of COBRA P1 HA in complex with 1F8 mAb | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: CryoEM half map A of COBRA P1 HA in complex with 1F8 mAb
| ファイル | emd_26983_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM half map A of COBRA P1 HA in complex with 1F8 mAb | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : COBRA P1 HA in complex with 1F8 mAb
| 全体 | 名称: COBRA P1 HA in complex with 1F8 mAb |
|---|---|
| 要素 |
|
-超分子 #1: COBRA P1 HA in complex with 1F8 mAb
| 超分子 | 名称: COBRA P1 HA in complex with 1F8 mAb / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #2: COBRA P1 hemagglutinin
| 超分子 | 名称: COBRA P1 hemagglutinin / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 詳細: HA antigen designed from human H1N1 viruses from 1933-1957 and 2009-2011, and swine H1N1 viruses from 1931-1998. |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス) |
-超分子 #3: 1F8 monoclonal antibody
| 超分子 | 名称: 1F8 monoclonal antibody / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 詳細: Recombinantly expressed Fab fragment of an IgG antibody. |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: COBRA P1 HA
| 分子 | 名称: COBRA P1 HA / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス) |
| 分子量 | 理論値: 63.264535 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DTICIGYHAN NSTDTVDTVL EKNVTVTHSV NLLEDSHNGK LCKLKGIAPL QLGKCNIAGW LLGNPECESL LSARSWSYIV ETPNSENGT CYPGDFIDYE ELREQLSSVS SFERFEIFPK ESSWPNHNTT KGVTAACSHA GKSSFYRNLL WLTKKGGSYP K LSKSYVNN ...文字列: DTICIGYHAN NSTDTVDTVL EKNVTVTHSV NLLEDSHNGK LCKLKGIAPL QLGKCNIAGW LLGNPECESL LSARSWSYIV ETPNSENGT CYPGDFIDYE ELREQLSSVS SFERFEIFPK ESSWPNHNTT KGVTAACSHA GKSSFYRNLL WLTKKGGSYP K LSKSYVNN KGKEVLVLWG VHHPSTSTDQ QSLYQNENAY VSVVSSNYNR RFTPEIAERP KVRGQAGRMN YYWTLLEPGD TI IFEATGN LIAPWYAFAL SRGSGSGIIT SNASMHECNT KCQTPQGAIN SSLPFQNIHP VTIGECPKYV RSTKLRMVTG LRN IPSIQS RGLFGAIAGF IEGGWTGMID GWYGYHHQNE QGSGYAADQK STQNAINGIT NKVNSVIEKM NTQFTAVGKE FNNL EKRME NLNKKVDDGF LDIWTYNAEL LVLLENERTL DFHDSNVKNL YEKVKSQLRN NAKEIGNGCF EFYHKCDNEC MESVK NGTY DYPKYSEESK LNREKIDGVK LESMGVYGSG YIPEAPRDGQ AYVRKDGEWV LLSTFLGLND IFEAQKIEWH EGHHHH HH |
-分子 #2: 1F8 light chain
| 分子 | 名称: 1F8 light chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 23.918271 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: DIVLTQSPAS LAVSLGQRAT ISCKASQSVD FDGDTYMSWY QQKPGQPPKL LIYAASKLES GIPARFSGSG SGTDFTLNIH PVEEEDAAT YFCQQSNEDP WTFGGGTKLE IKRADAAPTV SIFPPSSEQL TSGGASVVCF LNNFYPKDIN VKWKIDGSER Q NGVLNSWT ...文字列: DIVLTQSPAS LAVSLGQRAT ISCKASQSVD FDGDTYMSWY QQKPGQPPKL LIYAASKLES GIPARFSGSG SGTDFTLNIH PVEEEDAAT YFCQQSNEDP WTFGGGTKLE IKRADAAPTV SIFPPSSEQL TSGGASVVCF LNNFYPKDIN VKWKIDGSER Q NGVLNSWT DQDSKDSTYS MSSTLTLTKD EYERHNSYTC EATHKTSTSP IVKSFNRNEC |
-分子 #3: 1F8 heavy chain
| 分子 | 名称: 1F8 heavy chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 23.900818 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVQLQQSGAE LMKPGASVKI SCKATGYTFS SYWIEWVKQR PGHGLEWIGE ILPGSGRTNY DERFKGKATF TADTSSNTAY MQLSSLTSE DSAVYYCARP RIYGMDYWGQ GTSVTVSSAK TTPPSVYPLA PGSAAQTNSM VTLGCLVKGY FPEPVTVTWN S GSLSSGVH ...文字列: QVQLQQSGAE LMKPGASVKI SCKATGYTFS SYWIEWVKQR PGHGLEWIGE ILPGSGRTNY DERFKGKATF TADTSSNTAY MQLSSLTSE DSAVYYCARP RIYGMDYWGQ GTSVTVSSAK TTPPSVYPLA PGSAAQTNSM VTLGCLVKGY FPEPVTVTWN S GSLSSGVH TFPAVLQSDL YTLSSSVTVP SSPRPSETVT CNVAHPASST KVDKKIEPRG LVPR |
-分子 #7: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 7 / コピー数: 10 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: TBS |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: PLASMA CLEANING |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
| 詳細 | 1:3:3 molar ratio of P1 HA, 1F8, and P1-05 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)