+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | KSQ+AT from first module of the pikromycin synthase | |||||||||
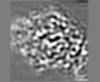
 マップデータ マップデータ | Primary map. Density fades for distal AT domains. Use AT_newest.ccp4 for these (related through twofold of PikKSAT1_dimer.cif). | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ketosynthase-like / acyltransferase / decarboxylation / BIOSYNTHETIC PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報10-deoxymethynolide synthase / narbonolide synthase / macrolide biosynthetic process / acyltransferase activity, transferring groups other than amino-acyl groups / fatty acid synthase activity / phosphopantetheine binding / 3-oxoacyl-[acyl-carrier-protein] synthase activity / fatty acid biosynthetic process 類似検索 - 分子機能 | |||||||||
| 生物種 |  Streptomyces venezuelae (バクテリア) Streptomyces venezuelae (バクテリア) | |||||||||
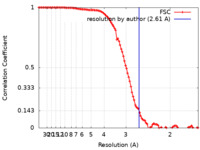
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.61 Å | |||||||||
 データ登録者 データ登録者 | Keatinge-Clay AT / Dickinson MS / Miyazawa T / McCool RS | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2022 ジャーナル: Structure / 年: 2022タイトル: Priming enzymes from the pikromycin synthase reveal how assembly-line ketosynthases catalyze carbon-carbon chemistry. 著者: Miles S Dickinson / Takeshi Miyazawa / Ryan S McCool / Adrian T Keatinge-Clay /  要旨: The first domain of modular polyketide synthases (PKSs) is most commonly a ketosynthase (KS)-like enzyme, KS, that primes polyketide synthesis. Unlike downstream KSs that fuse α-carboxyacyl groups ...The first domain of modular polyketide synthases (PKSs) is most commonly a ketosynthase (KS)-like enzyme, KS, that primes polyketide synthesis. Unlike downstream KSs that fuse α-carboxyacyl groups to growing polyketide chains, it performs an extension-decoupled decarboxylation of these groups to generate primer units. When Pik127, a model triketide synthase constructed from modules of the pikromycin synthase, was studied by cryoelectron microscopy (cryo-EM), the dimeric didomain comprised of KS and the neighboring methylmalonyl-selective acyltransferase (AT) dominated the class averages and yielded structures at 2.5- and 2.8-Å resolution, respectively. Comparisons with ketosynthases complexed with their substrates revealed the conformation of the (2S)-methylmalonyl-S-phosphopantetheinyl portion of KS and KS substrates prior to decarboxylation. Point mutants of Pik127 probed the roles of residues in the KS active site, while an AT-swapped version of Pik127 demonstrated that KS can also decarboxylate malonyl groups. Mechanisms for how KS and KS domains catalyze carbon-carbon chemistry are proposed. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26839.map.gz emd_26839.map.gz | 4.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26839-v30.xml emd-26839-v30.xml emd-26839.xml emd-26839.xml | 23.1 KB 23.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26839_fsc.xml emd_26839_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26839.png emd_26839.png | 67.7 KB | ||
| Filedesc metadata |  emd-26839.cif.gz emd-26839.cif.gz | 7.3 KB | ||
| その他 |  emd_26839_additional_1.map.gz emd_26839_additional_1.map.gz emd_26839_half_map_1.map.gz emd_26839_half_map_1.map.gz emd_26839_half_map_2.map.gz emd_26839_half_map_2.map.gz | 1 MB 95.3 MB 95.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26839 http://ftp.pdbj.org/pub/emdb/structures/EMD-26839 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26839 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26839 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26839_validation.pdf.gz emd_26839_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26839_full_validation.pdf.gz emd_26839_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_26839_validation.xml.gz emd_26839_validation.xml.gz | 16.7 KB | 表示 | |
| CIF形式データ |  emd_26839_validation.cif.gz emd_26839_validation.cif.gz | 20.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26839 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26839 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26839 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26839 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7uwrMC  8czcC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26839.map.gz / 形式: CCP4 / 大きさ: 5.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26839.map.gz / 形式: CCP4 / 大きさ: 5.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Primary map. Density fades for distal AT domains. Use AT_newest.ccp4 for these (related through twofold of PikKSAT1_dimer.cif). | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.81 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Map for AT (Chain B) obtained through local...
| ファイル | emd_26839_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
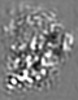
| 注釈 | Map for AT (Chain B) obtained through local refinement. Apply twofold from PikKSQAT1.cif to obtain map for AT (Chain A). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
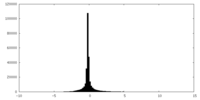
| 密度ヒストグラム |
-ハーフマップ: second half map
| ファイル | emd_26839_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
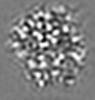
| 注釈 | second half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
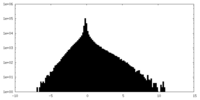
| 密度ヒストグラム |
-ハーフマップ: first half map
| ファイル | emd_26839_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | first half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
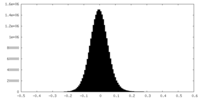
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Pik127
| 全体 | 名称: Pik127 |
|---|---|
| 要素 |
|
-超分子 #1: Pik127
| 超分子 | 名称: Pik127 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Polyketide synthase engineered from the first, second, and seventh modules of the pikromycin synthase as described in Miyazawa et al., 2021, Chem. Commun. 57:8762-8765 |
|---|---|
| 由来(天然) | 生物種:  Streptomyces venezuelae (バクテリア) / 株: 15439 Streptomyces venezuelae (バクテリア) / 株: 15439 |
| 分子量 | 理論値: 410 KDa |
-分子 #1: Narbonolide/10-deoxymethynolide synthase PikA1, modules 1 and 2
| 分子 | 名称: Narbonolide/10-deoxymethynolide synthase PikA1, modules 1 and 2 タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: 10-deoxymethynolide synthase |
|---|---|
| 由来(天然) | 生物種:  Streptomyces venezuelae (バクテリア) Streptomyces venezuelae (バクテリア) |
| 分子量 | 理論値: 91.797898 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EPVAVVGISC RVPGARDPRE FWELLAAGGQ AVTDVPADRW NAGDFYDPDR SAPGRSNSRW GGFIEDVDRF DAAFFGISPR EAAEMDPQQ RLALELGWEA LERAGIDPSS LTGTRTGVFA GAIWDDYATL KHRQGGAAIT PHTVTGLHRG IIANRLSYTL G LRGPSMVV ...文字列: EPVAVVGISC RVPGARDPRE FWELLAAGGQ AVTDVPADRW NAGDFYDPDR SAPGRSNSRW GGFIEDVDRF DAAFFGISPR EAAEMDPQQ RLALELGWEA LERAGIDPSS LTGTRTGVFA GAIWDDYATL KHRQGGAAIT PHTVTGLHRG IIANRLSYTL G LRGPSMVV DSGQSSSLVA VHLACESLRR GESELALAGG VSLNLVPDSI IGASKFGGLS PDGRAYTFDA RANGYVRGEG GG FVVLKRL SRAVADGDPV LAVIRGSAVN NGGAAQGMTT PDAQAQEAVL REAHERAGTA PADVRYVELH GTGTPVGDPI EAA ALGAAL GTGRPAGQPL LVGSVKTNIG HLEGAAGIAG LIKAVLAVRG RALPASLNYE TPNPAIPFEE LNLRVNTEYL PWEP EHDGQ RMVVGVSSFG MGGTNAHVVL EEAPGVVEGA SVVESTVGGS AVGGGVVPWV VSAKSAAALD AQIERLAAFA SRDRT DGVD AGAVDAGAVD AGAVARVLAG GRAQFEHRAV VVGSGPDDLA AALAAPEGLV RGVASGVGRV AFVFPGQGTQ WAGMGA ELL DSSAVFAAAM AECEAALSPY VDWSLEAVVR QAPGAPTLER VDVVQPVTFA VMVSLARVWQ HHGVTPQAVV GHSQGEI AA AYVAGALSLD DAARVVTLRS KSIAAHLAGK GGMLSLALSE DAVLERLAGF DGLSVAAVNG PTATVVSGDP VQIEELAR A CEADGVRARV IPVDYASHSR QVEIIESELA EVLAGLSPQA PRVPFFSTLE GAWITEPVLD GGYWYRNLRH RVGFAPAVE TLATDEGFTH FVEVSAHPVL TMALPGTVTG LATLRRDNGG QDRLVASLAE AWANGLAVDW SPLLPSATGH HSDLPTYAFQ TERHWL UniProtKB: Pikromycin polyketide synthase component PikAI |
-分子 #2: water
| 分子 | 名称: water / タイプ: ligand / ID: 2 / コピー数: 55 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: Buffer was made fresh because TCEP rapidly degrades in phosphate | |||||||||||||||
| グリッド | モデル: C-flat-2/2 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 12 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 30 sec. | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: Blotted for 3 seconds with a force of "1" before plunging.. | |||||||||||||||
| 詳細 | This sample was monodisperse. It was incubated with its substrates, methylmalonyl-CoA and NADPH, for 30 min. at 25 C before freezing. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 詳細 | Collected 4284 movies at 0 degrees tilt and 2912 movies at -30 degrees tilt |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 7196 / 平均露光時間: 5.0 sec. / 平均電子線量: 70.0 e/Å2 / 詳細: 20 frames per second |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 61728 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)