+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Nucleoplasmic pre-60S intermediate of the Nog2 containing post-rotation state from a SPB1 D52A strain | ||||||||||||
 マップデータ マップデータ | Nucleoplasmic pre-60S intermediate of the Nog2 containing post-rotation state from a SPB1 D52A strain | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | ribosome biogenesis / K-loop GTPase / GTPase / ribosome / 5S rRNP | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of ribosomal subunit export from nucleus / preribosome, large subunit precursor / ribosomal large subunit export from nucleus / large ribosomal subunit rRNA binding / GTPase activity / GTP binding / nucleolus / nucleoplasm / nucleus 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
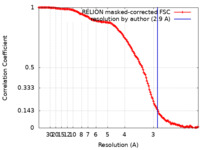
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | ||||||||||||
 データ登録者 データ登録者 | Sekulski K / Cruz VE / Weirich CS / Erzberger JP | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: rRNA methylation by Spb1 regulates the GTPase activity of Nog2 during 60S ribosomal subunit assembly. 著者: Kamil Sekulski / Victor Emmanuel Cruz / Christine S Weirich / Jan P Erzberger /  要旨: Biogenesis of the large ribosomal (60S) subunit involves the assembly of three rRNAs and 46 proteins, a process requiring approximately 70 ribosome biogenesis factors (RBFs) that bind and release the ...Biogenesis of the large ribosomal (60S) subunit involves the assembly of three rRNAs and 46 proteins, a process requiring approximately 70 ribosome biogenesis factors (RBFs) that bind and release the pre-60S at specific steps along the assembly pathway. The methyltransferase Spb1 and the K-loop GTPase Nog2 are essential RBFs that engage the rRNA A-loop during sequential steps in 60S maturation. Spb1 methylates the A-loop nucleotide G2922 and a catalytically deficient mutant strain (spb1) has a severe 60S biogenesis defect. However, the assembly function of this modification is currently unknown. Here, we present cryo-EM reconstructions that reveal that unmethylated G2922 leads to the premature activation of Nog2 GTPase activity and capture a Nog2-GDP-AlF transition state structure that implicates the direct involvement of unmodified G2922 in Nog2 GTPase activation. Genetic suppressors and in vivo imaging indicate that premature GTP hydrolysis prevents the efficient binding of Nog2 to early nucleoplasmic 60S intermediates. We propose that G2922 methylation levels regulate Nog2 recruitment to the pre-60S near the nucleolar/nucleoplasmic phase boundary, forming a kinetic checkpoint to regulate 60S production. Our approach and findings provide a template to study the GTPase cycles and regulatory factor interactions of the other K-loop GTPases involved in ribosome assembly. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26799.map.gz emd_26799.map.gz | 228.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26799-v30.xml emd-26799-v30.xml emd-26799.xml emd-26799.xml | 20.4 KB 20.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26799_fsc.xml emd_26799_fsc.xml | 14.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26799.png emd_26799.png | 126.8 KB | ||
| Filedesc metadata |  emd-26799.cif.gz emd-26799.cif.gz | 6.2 KB | ||
| その他 |  emd_26799_half_map_1.map.gz emd_26799_half_map_1.map.gz emd_26799_half_map_2.map.gz emd_26799_half_map_2.map.gz | 194.3 MB 193.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26799 http://ftp.pdbj.org/pub/emdb/structures/EMD-26799 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26799 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26799 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26799_validation.pdf.gz emd_26799_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26799_full_validation.pdf.gz emd_26799_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_26799_validation.xml.gz emd_26799_validation.xml.gz | 21.8 KB | 表示 | |
| CIF形式データ |  emd_26799_validation.cif.gz emd_26799_validation.cif.gz | 28.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26799 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26799 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26799 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26799 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26799.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26799.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Nucleoplasmic pre-60S intermediate of the Nog2 containing post-rotation state from a SPB1 D52A strain | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half-map 1 from nucleoplasmic pre-60S intermediate of the...
| ファイル | emd_26799_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
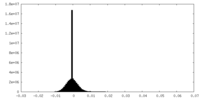
| 注釈 | Half-map 1 from nucleoplasmic pre-60S intermediate of the Nog2 containing post-rotation state from a SPB1 D52A strain | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map 2 from nucleoplasmic pre-60S intermediate of the...
| ファイル | emd_26799_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
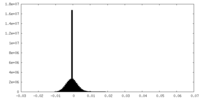
| 注釈 | Half-map 2 from nucleoplasmic pre-60S intermediate of the Nog2 containing post-rotation state from a SPB1 D52A strain | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Map of the nucleolar post-rotation pre-60S intermediate purified ...
| 全体 | 名称: Map of the nucleolar post-rotation pre-60S intermediate purified with tags on Tif6 and Nog2 from a SPB1 D52A strain. |
|---|---|
| 要素 |
|
-超分子 #1: Map of the nucleolar post-rotation pre-60S intermediate purified ...
| 超分子 | 名称: Map of the nucleolar post-rotation pre-60S intermediate purified with tags on Tif6 and Nog2 from a SPB1 D52A strain. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 3.1 MDa |
-分子 #1: Nucleolar GTP-binding protein 2
| 分子 | 名称: Nucleolar GTP-binding protein 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 55.58559 KDa |
| 配列 | 文字列: MGTGKKEKSR RIREGDTKDG NLRVKGENFY RDSKRVKFLN MYTSGKEIRN KKGNLIRAAS FQDSTIPDAR VQPDRRWFGN TRVISQDAL QHFRSALGET QKDTYQVLLR RNKLPMSLLE EKDADESPKA RILDTESYAD AFGPKAQRKR PRLAASNLED L VKATNEDI ...文字列: MGTGKKEKSR RIREGDTKDG NLRVKGENFY RDSKRVKFLN MYTSGKEIRN KKGNLIRAAS FQDSTIPDAR VQPDRRWFGN TRVISQDAL QHFRSALGET QKDTYQVLLR RNKLPMSLLE EKDADESPKA RILDTESYAD AFGPKAQRKR PRLAASNLED L VKATNEDI TKYEEKQVLD ATLGLMGNQE DKENGWTSAA KEAIFSKGQS KRIWNELYKV IDSSDVVIHV LDARDPLGTR CK SVEEYMK KETPHKHLIY VLNKCDLVPT WVAAAWVKHL SKERPTLAFH ASITNSFGKG SLIQLLRQFS QLHTDRKQIS VGF IGYPNT GKSSIINTLR KKKVCQVAPI PGETKVWQYI TLMKRIFLID CPGIVPPSSK DSEEDILFRG VVRVEHVTHP EQYI PGVLK RCQVKHLERT YEISGWKDAT EFIEILARKQ GRLLKGGEPD ESGVSKQILN DFNRGKIPWF VLPPEKEGEE KPKKK EVEK TA UniProtKB: Nucleolar GTP-binding protein 2 |
-分子 #2: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 2 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #3: POTASSIUM ION
| 分子 | 名称: POTASSIUM ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: K |
|---|---|
| 分子量 | 理論値: 39.098 Da |
-分子 #4: GUANOSINE-5'-DIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: GDP |
|---|---|
| 分子量 | 理論値: 443.201 Da |
| Chemical component information |  ChemComp-GDP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE | |||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 5434 / 平均露光時間: 0.05 sec. / 平均電子線量: 1.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 2.2 µm / 最小 デフォーカス(補正後): 0.9 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)