+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SARS-CoV-2 spike protein closed conformation | |||||||||
 マップデータ マップデータ | SARS-CoV-2 spike protein; sharpened with DeepEMhancer | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.56 Å | |||||||||
 データ登録者 データ登録者 | Kuhn CC / Basnet N / Bodakuntla S / Alvarez-Brecht P / Nichols S / Martinez-Sanchez A / Agostini L / Soh YM / Takagi J / Biertumpfel C / Mizuno N | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Direct Cryo-ET observation of platelet deformation induced by SARS-CoV-2 spike protein. 著者: Christopher Cyrus Kuhn / Nirakar Basnet / Satish Bodakuntla / Pelayo Alvarez-Brecht / Scott Nichols / Antonio Martinez-Sanchez / Lorenzo Agostini / Young-Min Soh / Junichi Takagi / Christian ...著者: Christopher Cyrus Kuhn / Nirakar Basnet / Satish Bodakuntla / Pelayo Alvarez-Brecht / Scott Nichols / Antonio Martinez-Sanchez / Lorenzo Agostini / Young-Min Soh / Junichi Takagi / Christian Biertümpfel / Naoko Mizuno /    要旨: SARS-CoV-2 is a novel coronavirus responsible for the COVID-19 pandemic. Its high pathogenicity is due to SARS-CoV-2 spike protein (S protein) contacting host-cell receptors. A critical hallmark of ...SARS-CoV-2 is a novel coronavirus responsible for the COVID-19 pandemic. Its high pathogenicity is due to SARS-CoV-2 spike protein (S protein) contacting host-cell receptors. A critical hallmark of COVID-19 is the occurrence of coagulopathies. Here, we report the direct observation of the interactions between S protein and platelets. Live imaging shows that the S protein triggers platelets to deform dynamically, in some cases, leading to their irreversible activation. Cellular cryo-electron tomography reveals dense decorations of S protein on the platelet surface, inducing filopodia formation. Hypothesizing that S protein binds to filopodia-inducing integrin receptors, we tested the binding to RGD motif-recognizing platelet integrins and find that S protein recognizes integrin αβ. Our results infer that the stochastic activation of platelets is due to weak interactions of S protein with integrin, which can attribute to the pathogenesis of COVID-19 and the occurrence of rare but severe coagulopathies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26798.map.gz emd_26798.map.gz | 331.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26798-v30.xml emd-26798-v30.xml emd-26798.xml emd-26798.xml | 22 KB 22 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26798_fsc.xml emd_26798_fsc.xml | 17.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26798.png emd_26798.png | 108.5 KB | ||
| マスクデータ |  emd_26798_msk_1.map emd_26798_msk_1.map | 371.3 MB |  マスクマップ マスクマップ | |
| その他 |  emd_26798_additional_1.map.gz emd_26798_additional_1.map.gz emd_26798_additional_2.map.gz emd_26798_additional_2.map.gz emd_26798_half_map_1.map.gz emd_26798_half_map_1.map.gz emd_26798_half_map_2.map.gz emd_26798_half_map_2.map.gz | 351.1 MB 185.4 MB 345 MB 345 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26798 http://ftp.pdbj.org/pub/emdb/structures/EMD-26798 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26798 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26798 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26798_validation.pdf.gz emd_26798_validation.pdf.gz | 770.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26798_full_validation.pdf.gz emd_26798_full_validation.pdf.gz | 769.7 KB | 表示 | |
| XML形式データ |  emd_26798_validation.xml.gz emd_26798_validation.xml.gz | 24.3 KB | 表示 | |
| CIF形式データ |  emd_26798_validation.cif.gz emd_26798_validation.cif.gz | 31.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26798 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26798 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26798 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26798 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26798.map.gz / 形式: CCP4 / 大きさ: 371.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26798.map.gz / 形式: CCP4 / 大きさ: 371.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | SARS-CoV-2 spike protein; sharpened with DeepEMhancer | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.93 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_26798_msk_1.map emd_26798_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
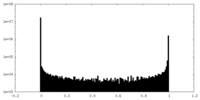
| 密度ヒストグラム |
-追加マップ: SARS-CoV-2 spike protein; sharpened cryosparc map
| ファイル | emd_26798_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | SARS-CoV-2 spike protein; sharpened cryosparc map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
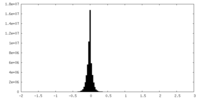
| 密度ヒストグラム |
-追加マップ: SARS-CoV-2 spike protein; unsharpened cryosparc map
| ファイル | emd_26798_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | SARS-CoV-2 spike protein; unsharpened cryosparc map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
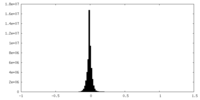
| 密度ヒストグラム |
-ハーフマップ: SARS-CoV-2 spike protein; half map A from cryosparc
| ファイル | emd_26798_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | SARS-CoV-2 spike protein; half map A from cryosparc | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
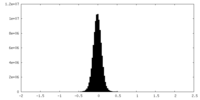
| 密度ヒストグラム |
-ハーフマップ: SARS-CoV-2 spike protein; half map B from cryosparc
| ファイル | emd_26798_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | SARS-CoV-2 spike protein; half map B from cryosparc | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Spike glycoprotein
| 全体 | 名称:  |
|---|---|
| 要素 |
|
-超分子 #1: Spike glycoprotein
| 超分子 | 名称: Spike glycoprotein / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Spike glycoprotein
| 分子 | 名称: Spike glycoprotein / タイプ: protein_or_peptide / ID: 1 詳細: SPIKE_SARS2 Spike glycoprotein(UniProt P0DTC2) RRAR furin cleavage site mutated to GSAGC-terminal Rho1D4-tag(TETSQVAPA) fused with spacer GSSG 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDGV YFASTEKSNI IRGWIFGTTL DSKTQSLLIV NNATNVVIKV CEFQFCNDPF LGVYYHKNNK SWMESEFRVY SSANNCTFEY ...文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDGV YFASTEKSNI IRGWIFGTTL DSKTQSLLIV NNATNVVIKV CEFQFCNDPF LGVYYHKNNK SWMESEFRVY SSANNCTFEY VSQPFLMDLE GKQGNFKNLR EFVFKNIDGY FKIYSKHTPI NLVRDLPQGF SALEPLVDLP IGINITRFQT LLALHRSYLT PGDSSSGWTA GAAAYYVGYL QPRTFLLKYN ENGTITDAVD CALDPLSETK CTLKSFTVEK GIYQTSNFRV QPTESIVRFP NITNLCPFGE VFNATRFASV YAWNRKRISN CVADYSVLYN SASFSTFKCY GVSPTKLNDL CFTNVYADSF VIRGDEVRQI APGQTGKIAD YNYKLPDDFT GCVIAWNSNN LDSKVGGNYN YLYRLFRKSN LKPFERDIST EIYQAGSTPC NGVEGFNCYF PLQSYGFQPT NGVGYQPYRV VVLSFELLHA PATVCGPKKS TNLVKNKCVN FNFNGLTGTG VLTESNKKFL PFQQFGRDIA DTTDAVRDPQ TLEILDITPC SFGGVSVITP GTNTSNQVAV LYQDVNCTEV PVAIHADQLT PTWRVYSTGS NVFQTRAGCL IGAEHVNNSY ECDIPIGAGI CASYQTQTNS PGSAGSVASQ SIIAYTMSLG AENSVAYSNN SIAIPTNFTI SVTTEILPVS MTKTSVDCTM YICGDSTECS NLLLQYGSFC TQLNRALTGI AVEQDKNTQE VFAQVKQIYK TPPIKDFGGF NFSQILPDPS KPSKRSFIED LLFNKVTLAD AGFIKQYGDC LGDIAARDLI CAQKFNGLTV LPPLLTDEMI AQYTSALLAG TITSGWTFGA GAALQIPFAM QMAYRFNGIG VTQNVLYENQ KLIANQFNSA IGKIQDSLSS TASALGKLQD VVNQNAQALN TLVKQLSSNF GAISSVLNDI LSRLDKVEAE VQIDRLITGR LQSLQTYVTQ QLIRAAEIRA SANLAATKMS ECVLGQSKRV DFCGKGYHLM SFPQSAPHGV VFLHVTYVPA QEKNFTTAPA ICHDGKAHFP REGVFVSNGT HWFVTQRNFY EPQIITTDNT FVSGNCDVVI GIVNNTVYDP LQPELDSFKE ELDKYFKNHT SPDVDLGDIS GINASVVNIQ KEIDRLNEVA KNLNESLIDL QELGKYEQYI KWPWYIWLGF IAGLIAIVMV TIMLCCMTSC CSCLKGCCSC GSCCKFDEDD SEPVLKGVKL HYTGSSGTET SQVAPA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 1 / 実像数: 3060 / 平均電子線量: 49.11 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 150000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)