+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM subtomogram average of IFT-A in anterograde IFT trains at 23 Angstrom resolution (Chlamydomonas reinhardtii) | |||||||||
 マップデータ マップデータ | Primary masked map | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 23.0 Å | |||||||||
 データ登録者 データ登録者 | Jordan MA / Pigino G | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Biorxiv / 年: 2022 ジャーナル: Biorxiv / 年: 2022タイトル: Integrative modeling reveals the molecular architecture of the Intraflagellar Transport A (IFT-A) complex 著者: McCafferty CL / Papoulas O / Jordan MA / Hoogerbrugge G / Nichols C / Pigino G / Taylor DW / Wallingford JB / Marcotte EM | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26791.map.gz emd_26791.map.gz | 109.8 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26791-v30.xml emd-26791-v30.xml emd-26791.xml emd-26791.xml | 15.9 KB 15.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_26791.png emd_26791.png | 66.4 KB | ||
| その他 |  emd_26791_additional_1.map.gz emd_26791_additional_1.map.gz emd_26791_additional_2.map.gz emd_26791_additional_2.map.gz | 844.3 KB 93.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26791 http://ftp.pdbj.org/pub/emdb/structures/EMD-26791 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26791 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26791 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26791_validation.pdf.gz emd_26791_validation.pdf.gz | 300.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26791_full_validation.pdf.gz emd_26791_full_validation.pdf.gz | 299.7 KB | 表示 | |
| XML形式データ |  emd_26791_validation.xml.gz emd_26791_validation.xml.gz | 5.2 KB | 表示 | |
| CIF形式データ |  emd_26791_validation.cif.gz emd_26791_validation.cif.gz | 5.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26791 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26791 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26791 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26791 | HTTPS FTP |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26791.map.gz / 形式: CCP4 / 大きさ: 2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26791.map.gz / 形式: CCP4 / 大きさ: 2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Primary masked map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 7.066 Å | ||||||||||||||||||||
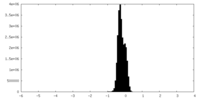
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Composite polymer map
| ファイル | emd_26791_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Composite polymer map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
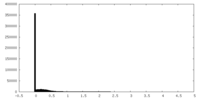
| 密度ヒストグラム |
-追加マップ: Large 300px raw map
| ファイル | emd_26791_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Large 300px raw map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
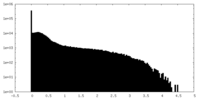
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : IFT-A complex comprised of IFT43, IFT121, IFT122, IFT139, IFT140,...
| 全体 | 名称: IFT-A complex comprised of IFT43, IFT121, IFT122, IFT139, IFT140, and IFT144 |
|---|---|
| 要素 |
|
-超分子 #1: IFT-A complex comprised of IFT43, IFT121, IFT122, IFT139, IFT140,...
| 超分子 | 名称: IFT-A complex comprised of IFT43, IFT121, IFT122, IFT139, IFT140, and IFT144 タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 詳細: Cryo-ET subtomogram averaging of an IFT-A complex, as observed in situ in the context of an anterograde intraflagellar transport (IFT) train in intact Chlamydomonas flagella |
|---|---|
| 由来(天然) | 生物種:  Organelle: cilia |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 |
|---|---|
| グリッド | モデル: Quantifoil R3.5/1 / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 291.15 K / 装置: LEICA EM GP |
| 詳細 | Fresh liquid cell cultures of 300 mL were grown for three to four days in TAP (Tris-acetate-phosphate) medium at 22 degrees C under a light-dark cycle with constant aeration. 3 microL undiluted Chlamydomonas cells were applied to the grid and mixed with 1 microL 10 nm colloidal gold particles (BBI solutions) |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV 詳細: Images were recorded with an energy filter (GIF, Gatan image filter). Digital Micrograph software (Gatan) was used to tune the GIF, and SerialEM software was employed for the automated ...詳細: Images were recorded with an energy filter (GIF, Gatan image filter). Digital Micrograph software (Gatan) was used to tune the GIF, and SerialEM software was employed for the automated acquisition of tomographic tilt series in low-dose mode. |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-10 / 撮影したグリッド数: 34 / 平均露光時間: 2.0 sec. / 平均電子線量: 1.85 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 6.0 µm / 最小 デフォーカス(公称値): 3.0 µm / 倍率(公称値): 30000 |
| 試料ステージ | 試料ホルダーモデル: GATAN 626 SINGLE TILT LIQUID NITROGEN CRYO TRANSFER HOLDER ホルダー冷却材: NITROGEN |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 23.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: PEET / 使用したサブトモグラム数: 9350 |
|---|---|
| 抽出 | トモグラム数: 96 / 使用した粒子像数: 9350 / 手法: manual picking / ソフトウェア - 名称:  IMOD IMOD詳細: bin6 tomograms were filtered with NAD (non-linear anisotropic diffusion) in IMOD to increase contrast and facilitate particle picking |
| CTF補正 | ソフトウェア - 名称:  IMOD IMOD詳細: CTF curves were estimated with CTFPLOTTER and the data were corrected by phase-flipping with CTFPHASEFLIP, both implemented in IMOD. |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: PEET |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X