+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | PARL-cleaved Skd3 (human ClpB) E455Q dodecamer bound to ATPgammaS | |||||||||||||||||||||
 マップデータ マップデータ | PARL-cleaved Skd3 (human ClpB) E455Q Nucleotide Binding Domain in the dodecameric state, half map 2 | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード | AAA+ ATPase / Chaperone / Mitochondria / Protein Folding | |||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||||||||
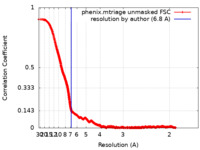
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.8 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Gupta A / Lentzsch AM / Siegel AS / Yu Z / Lu C / Chio US / Cheng Y / Shan S | |||||||||||||||||||||
| 資金援助 |  米国, 6件 米国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2023 ジャーナル: Sci Adv / 年: 2023タイトル: Dodecamer assembly of a metazoan AAA chaperone couples substrate extraction to refolding. 著者: Arpit Gupta / Alfred M Lentzsch / Alex Siegel / Zanlin Yu / Un Seng Chio / Yifan Cheng / Shu-Ou Shan /  要旨: Ring-forming AAA chaperones solubilize protein aggregates and protect organisms from proteostatic stress. In metazoans, the AAA chaperone Skd3 in the mitochondrial intermembrane space (IMS) is ...Ring-forming AAA chaperones solubilize protein aggregates and protect organisms from proteostatic stress. In metazoans, the AAA chaperone Skd3 in the mitochondrial intermembrane space (IMS) is critical for human health and efficiently refolds aggregated proteins, but its underlying mechanism is poorly understood. Here, we show that Skd3 harbors both disaggregase and protein refolding activities enabled by distinct assembly states. High-resolution structures of Skd3 hexamers in distinct conformations capture ratchet-like motions that mediate substrate extraction. Unlike previously described disaggregases, Skd3 hexamers further assemble into dodecameric cages in which solubilized substrate proteins can attain near-native states. Skd3 mutants defective in dodecamer assembly retain disaggregase activity but are impaired in client refolding, linking the disaggregase and refolding activities to the hexameric and dodecameric states of Skd3, respectively. We suggest that Skd3 is a combined disaggregase and foldase, and this property is particularly suited to meet the complex proteostatic demands in the mitochondrial IMS. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26728.map.gz emd_26728.map.gz | 70.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26728-v30.xml emd-26728-v30.xml emd-26728.xml emd-26728.xml | 16 KB 16 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26728_fsc.xml emd_26728_fsc.xml | 376.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26728.png emd_26728.png | 75.8 KB | ||
| Filedesc metadata |  emd-26728.cif.gz emd-26728.cif.gz | 5 KB | ||
| その他 |  emd_26728_half_map_1.map.gz emd_26728_half_map_1.map.gz emd_26728_half_map_2.map.gz emd_26728_half_map_2.map.gz | 71.1 MB 71.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26728 http://ftp.pdbj.org/pub/emdb/structures/EMD-26728 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26728 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26728 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26728_validation.pdf.gz emd_26728_validation.pdf.gz | 650.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26728_full_validation.pdf.gz emd_26728_full_validation.pdf.gz | 650.1 KB | 表示 | |
| XML形式データ |  emd_26728_validation.xml.gz emd_26728_validation.xml.gz | 86.3 KB | 表示 | |
| CIF形式データ |  emd_26728_validation.cif.gz emd_26728_validation.cif.gz | 172.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26728 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26728 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26728 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26728 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26728.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26728.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PARL-cleaved Skd3 (human ClpB) E455Q Nucleotide Binding Domain in the dodecameric state, half map 2 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.3904 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: PARL-cleaved Skd3 (human ClpB) E455Q Nucleotide Binding Domain...
| ファイル | emd_26728_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PARL-cleaved Skd3 (human ClpB) E455Q Nucleotide Binding Domain in the dodecameric state, half map 1 | ||||||||||||
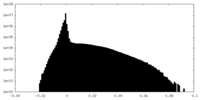
| 投影像・断面図 |
| ||||||||||||
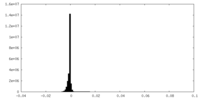
| 密度ヒストグラム |
-ハーフマップ: PARL-cleaved Skd3 (human ClpB) E455Q Nucleotide Binding Domain...
| ファイル | emd_26728_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PARL-cleaved Skd3 (human ClpB) E455Q Nucleotide Binding Domain in the dodecameric state, half map 1 | ||||||||||||
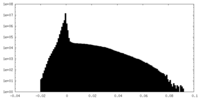
| 投影像・断面図 |
| ||||||||||||
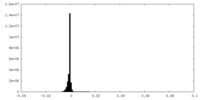
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : PARL-cleaved Skd3
| 全体 | 名称: PARL-cleaved Skd3 |
|---|---|
| 要素 |
|
-超分子 #1: PARL-cleaved Skd3
| 超分子 | 名称: PARL-cleaved Skd3 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 792 KDa |
-分子 #1: PARL-cleaved Skd3
| 分子 | 名称: PARL-cleaved Skd3 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: YSKSPSNKDA ALLEAARANN MQEVSRLLSE GADVNAKHRL GWTALMVAAI NRNNSVVQVL LAAGADPNLG DDFSSVYKTA KEQGIHSLED GGQDGASRHI TNQWTSALEF RRWLGLPAGV LITREDDFNN RLNNRASFKG CTALHYAVLA DDYRTVKELL DGGANPLQRN ...文字列: YSKSPSNKDA ALLEAARANN MQEVSRLLSE GADVNAKHRL GWTALMVAAI NRNNSVVQVL LAAGADPNLG DDFSSVYKTA KEQGIHSLED GGQDGASRHI TNQWTSALEF RRWLGLPAGV LITREDDFNN RLNNRASFKG CTALHYAVLA DDYRTVKELL DGGANPLQRN EMGHTPLDYA REGEVMKLLR TSEAKYQEKQ RKREAEERRR FPLEQRLKEH IIGQESAIAT VGAAIRRKEN GWYDEEHPLV FLFLGSSGIG KTELAKQTAK YMHKDAKKGF IRLDMSEFQE RHEVAKFIGS PPGYVGHEEG GQLTKKLKQC PNAVVLFDQV DKAHPDVLTI MLQLFDEGRL TDGKGKTIDC KDAIFIMTSN VASDEIAQHA LQLRQEALEM SRNRIAENLG DVQISDKITI SKNFKENVIR PILKAHFRRD EFLGRINEIV YFLPFCHSEL IQLVNKELNF WAKRAKQRHN ITLLWDREVA DVLVDGYNVH YGARSIKHEV ERRVVNQLAA AYEQDLLPGG CTLRITVEDS DKQLLKSPEL PSPQAEKRLP KLRLEIIDKD SKTRRLDIRA PLHPEKVCNT I |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): -2.0 µm / 最小 デフォーカス(公称値): -1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)