+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SARS-CoV-2 Omicron-BA.1 2-RBD up Spike Protein Trimer without the P986-P987 stabilizing mutations (S-GSAS-Omicron-BA.1) | |||||||||
 マップデータ マップデータ | Sharp map from homogeneous C1 refinement in cryosparc | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Omicron Spike protein / SARS-CoV-2 / variant of concern / 1-down / VIRAL PROTEIN / BA.1 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.96 Å | |||||||||
 データ登録者 データ登録者 | Stalls V / Acharya P | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: bioRxiv / 年: 2022 ジャーナル: bioRxiv / 年: 2022タイトル: Structural diversity of the SARS-CoV-2 Omicron spike. 著者: Sophie M-C Gobeil / Rory Henderson / Victoria Stalls / Katarzyna Janowska / Xiao Huang / Aaron May / Micah Speakman / Esther Beaudoin / Kartik Manne / Dapeng Li / Rob Parks / Maggie Barr / ...著者: Sophie M-C Gobeil / Rory Henderson / Victoria Stalls / Katarzyna Janowska / Xiao Huang / Aaron May / Micah Speakman / Esther Beaudoin / Kartik Manne / Dapeng Li / Rob Parks / Maggie Barr / Margaret Deyton / Mitchell Martin / Katayoun Mansouri / Robert J Edwards / Gregory D Sempowski / Kevin O Saunders / Kevin Wiehe / Wilton Williams / Bette Korber / Barton F Haynes / Priyamvada Acharya 要旨: Aided by extensive spike protein mutation, the SARS-CoV-2 Omicron variant overtook the previously dominant Delta variant. Spike conformation plays an essential role in SARS-CoV-2 evolution via ...Aided by extensive spike protein mutation, the SARS-CoV-2 Omicron variant overtook the previously dominant Delta variant. Spike conformation plays an essential role in SARS-CoV-2 evolution via changes in receptor binding domain (RBD) and neutralizing antibody epitope presentation affecting virus transmissibility and immune evasion. Here, we determine cryo-EM structures of the Omicron and Delta spikes to understand the conformational impacts of mutations in each. The Omicron spike structure revealed an unusually tightly packed RBD organization with long range impacts that were not observed in the Delta spike. Binding and crystallography revealed increased flexibility at the functionally critical fusion peptide site in the Omicron spike. These results reveal a highly evolved Omicron spike architecture with possible impacts on its high levels of immune evasion and transmissibility. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26600.map.gz emd_26600.map.gz | 96.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26600-v30.xml emd-26600-v30.xml emd-26600.xml emd-26600.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_26600.png emd_26600.png | 79.9 KB | ||
| Filedesc metadata |  emd-26600.cif.gz emd-26600.cif.gz | 5.6 KB | ||
| その他 |  emd_26600_half_map_1.map.gz emd_26600_half_map_1.map.gz emd_26600_half_map_2.map.gz emd_26600_half_map_2.map.gz | 95.4 MB 95.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26600 http://ftp.pdbj.org/pub/emdb/structures/EMD-26600 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26600 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26600 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26600_validation.pdf.gz emd_26600_validation.pdf.gz | 777.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26600_full_validation.pdf.gz emd_26600_full_validation.pdf.gz | 777.4 KB | 表示 | |
| XML形式データ |  emd_26600_validation.xml.gz emd_26600_validation.xml.gz | 13.5 KB | 表示 | |
| CIF形式データ |  emd_26600_validation.cif.gz emd_26600_validation.cif.gz | 15.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26600 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26600 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26600 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26600 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26600.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26600.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharp map from homogeneous C1 refinement in cryosparc | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half A map from homogeneous C1 refinement in cryosparc
| ファイル | emd_26600_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half A map from homogeneous C1 refinement in cryosparc | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
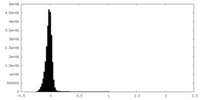
| 密度ヒストグラム |
-ハーフマップ: Half B map from homogeneous C1 refinement in cryosparc
| ファイル | emd_26600_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half B map from homogeneous C1 refinement in cryosparc | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
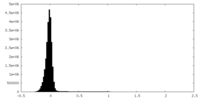
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV-2 S-GSAS-Omicron-BA.1 Spike Ectodomain
| 全体 | 名称: SARS-CoV-2 S-GSAS-Omicron-BA.1 Spike Ectodomain |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 S-GSAS-Omicron-BA.1 Spike Ectodomain
| 超分子 | 名称: SARS-CoV-2 S-GSAS-Omicron-BA.1 Spike Ectodomain / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 427 KDa |
-分子 #1: Spike glycoprotein
| 分子 | 名称: Spike glycoprotein / タイプ: other / ID: 1 / 分類: other |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHVISG TNGTKRFDNP VLPFNDGVYF ASIEKSNIIR GWIFGTTLDS KTQSLLIVNN ATNVVIKVCE FQFCNDPFLD HKNNKSWMES EFRVYSSANN CTFEYVSQPF ...文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHVISG TNGTKRFDNP VLPFNDGVYF ASIEKSNIIR GWIFGTTLDS KTQSLLIVNN ATNVVIKVCE FQFCNDPFLD HKNNKSWMES EFRVYSSANN CTFEYVSQPF LMDLEGKQGN FKNLREFVFK NIDGYFKIYS KHTPIIVREP EDLPQGFSAL EPLVDLPIGI NITRFQTLLA LHRSYLTPGD SSSGWTAGAA AYYVGYLQPR TFLLKYNENG TITDAVDCAL DPLSETKCTL KSFTVEKGIY QTSNFRVQPT ESIVRFPNIT NLCPFDEVFN ATRFASVYAW NRKRISNCVA DYSVLYNLAP FFTFKCYGVS PTKLNDLCFT NVYADSFVIR GDEVRQIAPG QTGNIADYNY KLPDDFTGCV IAWNSNKLDS KVSGNYNYLY RLFRKSNLKP FERDISTEIY QAGNKPCNGV AGFNCYFPLR SYSFRPTYGV GHQPYRVVVL SFELLHAPAT VCGPKKSTNL VKNKCVNFNF NGLKGTGVLT ESNKKFLPFQ QFGRDIADTT DAVRDPQTLE ILDITPCSFG GVSVITPGTN TSNQVAVLYQ GVNCTEVPVA IHADQLTPTW RVYSTGSNVF QTRAGCLIGA EYVNNSYECD IPIGAGICAS YQTQTKSHGS ASSVASQSII AYTMSLGAEN SVAYSNNSIA IPTNFTISVT TEILPVSMTK TSVDCTMYIC GDSTECSNLL LQYGSFCTQL KRALTGIAVE QDKNTQEVFA QVKQIYKTPP IKYFGGFNFS QILPDPSKPS KRSFIEDLLF NKVTLADAGF IKQYGDCLGD IAARDLICAQ KFKGLTVLPP LLTDEMIAQY TSALLAGTIT SGWTFGAGAA LQIPFAMQMA YRFNGIGVTQ NVLYENQKLI ANQFNSAIGK IQDSLSSTAS ALGKLQDVVN HNAQALNTLV KQLSSKFGAI SSVLNDIFSR LDKVEAEVQI DRLITGRLQS LQTYVTQQLI RAAEIRASAN LAATKMSECV LGQSKRVDFC GKGYHLMSFP QSAPHGVVFL HVTYVPAQEK NFTTAPAICH DGKAHFPREG VFVSNGTHWF VTQRNFYEPQ IITTDNTFVS GNCDVVIGIV NNTVYDPLQP ELDS |
| 組換発現 | 生物種: Mammalia (両生類) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: NITROGEN |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.2 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.96 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 101363 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)