+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of full-length dimeric ClbP | |||||||||
 マップデータ マップデータ | Final map for ClbP | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | colibactin peptidase / S12 peptidase / inner-membrane hydrolase / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報antibiotic catabolic process / beta-lactamase activity / beta-lactamase / outer membrane-bounded periplasmic space / response to antibiotic / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
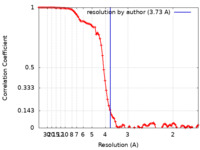
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.73 Å | |||||||||
 データ登録者 データ登録者 | Velilla JA / Walsh Jr RM / Gaudet R | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Chem Biol / 年: 2023 ジャーナル: Nat Chem Biol / 年: 2023タイトル: Structural basis of colibactin activation by the ClbP peptidase. 著者: José A Velilla / Matthew R Volpe / Grace E Kenney / Richard M Walsh / Emily P Balskus / Rachelle Gaudet /  要旨: Colibactin, a DNA cross-linking agent produced by gut bacteria, is implicated in colorectal cancer. Its biosynthesis uses a prodrug resistance mechanism: a non-toxic precursor assembled in the ...Colibactin, a DNA cross-linking agent produced by gut bacteria, is implicated in colorectal cancer. Its biosynthesis uses a prodrug resistance mechanism: a non-toxic precursor assembled in the cytoplasm is activated after export to the periplasm. This activation is mediated by ClbP, an inner-membrane peptidase with an N-terminal periplasmic catalytic domain and a C-terminal three-helix transmembrane domain. Although the transmembrane domain is required for colibactin activation, its role in catalysis is unclear. Our structure of full-length ClbP bound to a product analog reveals an interdomain interface important for substrate binding and enzyme stability and interactions that explain the selectivity of ClbP for the N-acyl-D-asparagine prodrug motif. Based on structural and biochemical evidence, we propose that ClbP dimerizes to form an extended substrate-binding site that can accommodate a pseudodimeric precolibactin with its two terminal prodrug motifs in the two ClbP active sites, thus enabling the coordinated activation of both electrophilic warheads. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26593.map.gz emd_26593.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26593-v30.xml emd-26593-v30.xml emd-26593.xml emd-26593.xml | 18.6 KB 18.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26593_fsc.xml emd_26593_fsc.xml | 8.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26593.png emd_26593.png | 190 KB | ||
| Filedesc metadata |  emd-26593.cif.gz emd-26593.cif.gz | 6.6 KB | ||
| その他 |  emd_26593_half_map_1.map.gz emd_26593_half_map_1.map.gz emd_26593_half_map_2.map.gz emd_26593_half_map_2.map.gz | 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26593 http://ftp.pdbj.org/pub/emdb/structures/EMD-26593 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26593 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26593 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26593_validation.pdf.gz emd_26593_validation.pdf.gz | 888.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26593_full_validation.pdf.gz emd_26593_full_validation.pdf.gz | 888.2 KB | 表示 | |
| XML形式データ |  emd_26593_validation.xml.gz emd_26593_validation.xml.gz | 16.4 KB | 表示 | |
| CIF形式データ |  emd_26593_validation.cif.gz emd_26593_validation.cif.gz | 21.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26593 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26593 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26593 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26593 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ul6MC  7mdeC  7mdfC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26593.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26593.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final map for ClbP | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.825 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map A for ClbP
| ファイル | emd_26593_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A for ClbP | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B for ClbP
| ファイル | emd_26593_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B for ClbP | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ClbP
| 全体 | 名称: ClbP |
|---|---|
| 要素 |
|
-超分子 #1: ClbP
| 超分子 | 名称: ClbP / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Full-length ClbP |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 110 KDa |
-分子 #1: Beta-lactamase
| 分子 | 名称: Beta-lactamase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: beta-lactamase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 53.523309 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QEHEPIGAQD ERLSTLIHQR MQEAKVPALS VSVTIKGVRQ RFVYGVADVA SQKANTLDTV YELGSMSKAF TGLVVQILIQ EGRLRQGDD IITYLPEMRL NYQGKPASLT VADFLYHTSG LPFSTLARLE NPMPGSAVAQ QLRNENLLFA PGAKFSYASA N YDVLGAVI ...文字列: QEHEPIGAQD ERLSTLIHQR MQEAKVPALS VSVTIKGVRQ RFVYGVADVA SQKANTLDTV YELGSMSKAF TGLVVQILIQ EGRLRQGDD IITYLPEMRL NYQGKPASLT VADFLYHTSG LPFSTLARLE NPMPGSAVAQ QLRNENLLFA PGAKFSYASA N YDVLGAVI ENVTGKTFTE VIAERLTQPL GMSATVAVKG DEIIVNKASG YKLGFGKPVL FHAPLARNHV PAAYIHSTLP DM EIWIDAW LHRKALPATL REAMSNSWRG NSDVPLAADN RILYASGWFI DQNQGPYISH GGQNPNFSSC IALRPDQQIG IVA LANMNS NLILQLCADI DNYLRIGKYA DGAGDAITAT DTLFVYLTLL LCFWGAVVVV RGAFRVYRAT AHGPGKQQRL RLRV RDYII ALAVPGLVAA MLYVAPGILS PGLDWRFILV WGPSSVLAIP FGIILLAFVL TLNHQIKRIL LHNKEWDDEH HHHHH HHHH UniProtKB: Beta-lactamase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.3 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa / 詳細: 30 s glow discharge at 15mA | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV 詳細: Three uL of sample were deposited onto 400 mesh Quantifoil Cu 1.2/1.3 grids that had been glow discharged in a PELCO easiGLOW (Ted Pella) at 0.39 mBar, 15 mA for 30 s. Samples were vitrified ...詳細: Three uL of sample were deposited onto 400 mesh Quantifoil Cu 1.2/1.3 grids that had been glow discharged in a PELCO easiGLOW (Ted Pella) at 0.39 mBar, 15 mA for 30 s. Samples were vitrified in 100% liquid ethane using a Vitrobot Mark IV (Thermo Fisher Scientific), with a wait time of 30 s, blot time of 5 s and a blot force of 16 at 100% humidity.. | ||||||||||||
| 詳細 | Protein was purified by Ni affinity chromatography followed by SEC on an S200 10/300 column equilibrated with 10 mM HEPES pH 7.3, 200 mM NaCl, 0.06% GDN. Sample used for preparing grids came from the peak fraction and was not concentrated. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 3888 / 平均露光時間: 2.5 sec. / 平均電子線量: 76.191 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 60606 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)