[日本語] English
 万見
万見- EMDB-26574: KS-AT di-domain of mycobacterial Pks13 with endogenous KS ligand bound -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | KS-AT di-domain of mycobacterial Pks13 with endogenous KS ligand bound | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | mycolic acid synthesis /  ketosynthase / ketosynthase /  acyltransferase (アシルトランスフェラーゼ) / acyltransferase (アシルトランスフェラーゼ) /  multi-domain assembly (タンパク質ドメイン) / multi-domain assembly (タンパク質ドメイン) /  BIOSYNTHETIC PROTEIN (生合成) BIOSYNTHETIC PROTEIN (生合成) | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報6-deoxyerythronolide-B synthase /  erythronolide synthase activity / lipid biosynthetic process / erythronolide synthase activity / lipid biosynthetic process /  phosphopantetheine binding phosphopantetheine binding類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) | ||||||||||||
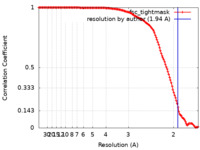
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 1.94 Å クライオ電子顕微鏡法 / 解像度: 1.94 Å | ||||||||||||
 データ登録者 データ登録者 | Kim SK / Dickinson MS / Finer-Moore JS / Rosenberg OS / Stroud RM | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Structure and dynamics of the essential endogenous mycobacterial polyketide synthase Pks13. 著者: Sun Kyung Kim / Miles Sasha Dickinson / Janet Finer-Moore / Ziqiang Guan / Robyn M Kaake / Ignacia Echeverria / Jen Chen / Ernst H Pulido / Andrej Sali / Nevan J Krogan / Oren S Rosenberg / Robert M Stroud /  要旨: The mycolic acid layer of the Mycobacterium tuberculosis cell wall is essential for viability and virulence, and the enzymes responsible for its synthesis are targets for antimycobacterial drug ...The mycolic acid layer of the Mycobacterium tuberculosis cell wall is essential for viability and virulence, and the enzymes responsible for its synthesis are targets for antimycobacterial drug development. Polyketide synthase 13 (Pks13) is a module encoding several enzymatic and transport functions that carries out the condensation of two different long-chain fatty acids to produce mycolic acids. We determined structures by cryogenic-electron microscopy of dimeric multi-enzyme Pks13 purified from mycobacteria under normal growth conditions, captured with native substrates. Structures define the ketosynthase (KS), linker and acyl transferase (AT) domains at 1.8 Å resolution and two alternative locations of the N-terminal acyl carrier protein. These structures suggest intermediate states on the pathway for substrate delivery to the KS domain. Other domains, visible at lower resolution, are flexible relative to the KS-AT core. The chemical structures of three bound endogenous long-chain fatty acid substrates were determined by electrospray ionization mass spectrometry. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26574.map.gz emd_26574.map.gz | 108.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26574-v30.xml emd-26574-v30.xml emd-26574.xml emd-26574.xml | 27.3 KB 27.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26574_fsc.xml emd_26574_fsc.xml | 14.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26574.png emd_26574.png | 74.6 KB | ||
| その他 |  emd_26574_additional_1.map.gz emd_26574_additional_1.map.gz emd_26574_additional_2.map.gz emd_26574_additional_2.map.gz emd_26574_half_map_1.map.gz emd_26574_half_map_1.map.gz emd_26574_half_map_2.map.gz emd_26574_half_map_2.map.gz | 14 MB 185.7 MB 202.8 MB 202.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26574 http://ftp.pdbj.org/pub/emdb/structures/EMD-26574 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26574 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26574 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26574.map.gz / 形式: CCP4 / 大きさ: 219.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26574.map.gz / 形式: CCP4 / 大きさ: 219.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.835 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: density-modified, sharpened map
| ファイル | emd_26574_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | density-modified, sharpened map | ||||||||||||
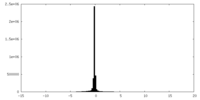
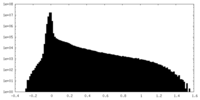
| 投影像・断面図 |
| ||||||||||||
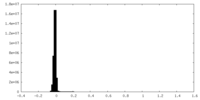
| 密度ヒストグラム |
-追加マップ: DeepEMhancer sharpened map contour at 0.268
| ファイル | emd_26574_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DeepEMhancer sharpened map contour at 0.268 | ||||||||||||
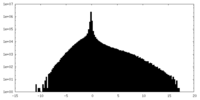
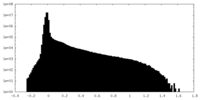
| 投影像・断面図 |
| ||||||||||||
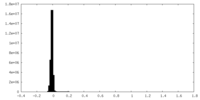
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_26574_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
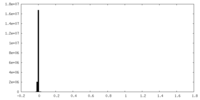
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_26574_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
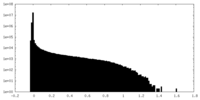
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Mycobacterial polyketide synthase 13
| 全体 | 名称: Mycobacterial polyketide synthase 13 |
|---|---|
| 要素 |
|
-超分子 #1: Mycobacterial polyketide synthase 13
| 超分子 | 名称: Mycobacterial polyketide synthase 13 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: The gene for Mycobacterium smegmatis polyketide synthase 13 (Pks13) was tagged with TEV-cleavable GFP at its C-terminus and purified from its natural source with anti-GFP nanobody beads. GFP ...詳細: The gene for Mycobacterium smegmatis polyketide synthase 13 (Pks13) was tagged with TEV-cleavable GFP at its C-terminus and purified from its natural source with anti-GFP nanobody beads. GFP was cleaved to yield the full-length Pks13. |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
-超分子 #2: Ketosynthase domain
| 超分子 | 名称: Ketosynthase domain / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 / 詳細: Linked to acyl transferase domain at the C-terminus |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
-超分子 #3: Acyl transferase domain
| 超分子 | 名称: Acyl transferase domain / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #1 / 詳細: linked to ketosynthase domain at the N-terminus |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
-分子 #1: Polyketide synthase PKS13
| 分子 | 名称: Polyketide synthase PKS13 / タイプ: protein_or_peptide / ID: 1 詳細: The gene for M. smegmatis polyketide synthase 13 (Pks13) was tagged with TEV-cleavable GFP at its C-terminus and purified from its natural source with anti-GFP nanobody beads. GFP was cleaved ...詳細: The gene for M. smegmatis polyketide synthase 13 (Pks13) was tagged with TEV-cleavable GFP at its C-terminus and purified from its natural source with anti-GFP nanobody beads. GFP was cleaved to yield the full-length Pks13. コピー数: 2 / 光学異性体: LEVO / EC番号: 6-deoxyerythronolide-B synthase |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア)株: ATCC 700084 / mc(2)155 |
| 分子量 | 理論値: 198.633359 KDa |
| 配列 | 文字列: MDENHSDSNL PEGTEPAGRP APRTDMTVNE MREWLRNWVA NATGQSADAI DESTPMVELG LSSRDAVAMA SDIEDLTGVT LTATVAFRH PTIESLATVI IEGEPEPEPY DEDEDWSRTR DVEDIAIVGV ATRFPGDLNT PDEMWEALLE GKDCVTDLPE D RWTEFLDE ...文字列: MDENHSDSNL PEGTEPAGRP APRTDMTVNE MREWLRNWVA NATGQSADAI DESTPMVELG LSSRDAVAMA SDIEDLTGVT LTATVAFRH PTIESLATVI IEGEPEPEPY DEDEDWSRTR DVEDIAIVGV ATRFPGDLNT PDEMWEALLE GKDCVTDLPE D RWTEFLDE PRIAERVKKA RTRGGYLTDI KGFDSEFFAL SKMEADNIDP QQRMALELTW EALEHARIPA SSLRGESVGV YI GSSTNDY SFLAMSDPSI AHPYAITGTA SSIIANRVSY FYDFRGPSVA VDTACSSSLV ATHQGVQALR AGEADVAIVG GVN ALVTPL VTVGFDEVGG VLAPDGRIKS FSSDADGYAR SEGGGMLVLK RISDARRDGD QILAVIAGSA VNHDGRSNGL LAPN PDAQA EVLRKAYKDA GINPRDVDYI EAHGTGTILG DPIEADALGR IVGKGRPADK PALLGAVKSN LGHLESAAGA ASLAK MTLA LANDKLPPSI NYAGPNPYID FEKERLKVND TVSDWPRYSG KAIAGVSGFG FGGANAHVVM REVLAGDLVE PEPEPE PEA KPEKSEADAV YVGGVRMDEY GEFIDEDEPA EGGDAYPSYD EDSYELPGIT EAAQRLLEQA REELEAKEAE EPTKQLV PL AVSAFLTSRK RQAAAELADW IDSPEGRASS LESIGRSLSR RNHGRSRAVV LAHDHDEAIK GLRALAEGKQ HPSVLSAD G PVTNGPVWVL AGFGAQHRKM GKSLYLRNEV FAEWINKVDA LIQDERGYSI LELILDDNVD YTDATCEYPI EVVQLVIFA IQIALGELLR HHGAKPAAVV GQSLGEAAAS YFAGGLSLAD ATRTICSRSH LMGEGEAMLF GEYIRLMALV EYSADEIKTV FSDYPDLEV CVYAAPTQTV IGGPPDQVDA IIARAESEGK FARKFQTKGA SHTQQMDPLL GELAAELQGI EPKPLTTGYF S TVHEGTFI RPGSAPIHDV DYWKKGLRHS VYFTQGIRNA VDNGHTTFLE LAPNPVALMQ VGLTTASAGL HDAQLIATLA RK QDEVESM ISAMAQLYVH GHDLDFRTLF PRRSKGLAGA LDFANIPPTR FKRKEHWLPA HFTGDSSAVM PGNHVATPDG RHV WEFVPR GKTDLAALVK AAAAQVLPDA KLAAFEQRAV PADNARLVTT LTRHPGGATV QVHARVEESF TLVYDAIVAR ANGA GVTAL PVAVGAGVAV SGDVAGEGAG ASVIEDDEPD AEILQDNLTA GAGMGADFQK WDPNSGETIG QRLGTIVGAA MGYEP EDLP WEVPLIELGL DSLMAVRIKN RVEYDFDLPP IQLTAVRDAN LYNVEELIRY AIEHRDEVEQ IAESQKGKTA EEIAAE QSE LLGGASTVAE LEAKLAEAGH PLAAKDSEDS ENSEDNAAGA AAAAEASAVE GLEIPPPPTD PTGPGGAPIP PPPSDPS GP AQAASATDAP AGTVNKATAA AAAAKVLTQE AVTEALGADV PPRDAAERVT FATWAIVTGK SPGGIFNELP TVSEETAK K MAERLSERAE GTITVEDVLG AKTIEGLATI VREQLEEGVV DGFVRTLRPP KEGSNAVPLF VFHPAGGSTV VYEPLMKRL PADVPVYGLE RVEGSIEERA AEYVPKLLEM HKGPFVLAGW SLGGALAYAC AIGLKQSGAD VRFVGLIDTV LPGEPIDQSK EGMRARWDR YARFAERTFN VEIPAIPYEE LEKLDDEGQV KYVLEIVKES GVQIPGGIIE HQRTSYLDNR ALDTVDIKPY D GHVTLYMA DRYHDDAIVF EPAYATRKPD GGWGSFVSDL EVVHIGGEHI QAIDEPYIAK VGAHMSEALN RIEAQASKED GA KSKSTSE NLYFQ |
-分子 #2: UNKNOWN LIGAND
| 分子 | 名称: UNKNOWN LIGAND / タイプ: ligand / ID: 2 詳細: The fatty acid ligand designated as UNL (unknown ligand) is a divided population of fatty acids of formula C55H106O2 and C40H78O2 as confirmed by mass spectrometry. コピー数: 2 / 式: UNL |
|---|---|
| 分子量 | 理論値: 312.53 Da |
| Chemical component information | 
ChemComp-UNL: |
-分子 #3: water
| 分子 | 名称: water / タイプ: ligand / ID: 3 / コピー数: 390 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283.15 K / 装置: FEI VITROBOT MARK IV 詳細: Full length pks13 was concentrated to 1.5 mg mL-1 for cryo-EM grid preparation. 4.5 uL of sample was applied to freshly glow discharged holey carbon on gold R1.2/1.3 300 mesh Quantifoil grids ...詳細: Full length pks13 was concentrated to 1.5 mg mL-1 for cryo-EM grid preparation. 4.5 uL of sample was applied to freshly glow discharged holey carbon on gold R1.2/1.3 300 mesh Quantifoil grids and blotted for 9 s with Whatman 1 filter paper at max humidity and 10oC in a FEI Mark IV Vitrobot, before vitrification in liquid nitrogen-cooled liquid ethane.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm最小 デフォーカス(公称値): 0.7000000000000001 µm 倍率(公称値): 105000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 7567 / 平均露光時間: 5.9 sec. / 平均電子線量: 45.8 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7uk4: |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X