+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Bacteriophage Lambda Red-Beta N-terminal domain helical assembly in complex with dsDNA | |||||||||
 マップデータ マップデータ | Symmetrised, sharpened map of the RedBeta177 helical assembly bound to dsDNA | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Annealase / Synaptase / SSAP / Single-strand annealing protein / DNA annealing intermediate / Recombinase / Two-component recombinase / Viral / DNA-binding / RECOMBINATION-DNA complex | |||||||||
| 機能・相同性 | Bacteriophage lambda, Recombination protein bet / RecT family / RecT family / DNA recombination / DNA binding / Recombination protein bet 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Escherichia virus Lambda (ウイルス) / Escherichia virus Lambda (ウイルス) /  | |||||||||
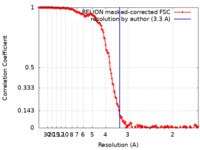
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Newing TP / Tolun G | |||||||||
| 資金援助 |  オーストラリア, 1件 オーストラリア, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Redβ annealase structure reveals details of oligomerization and λ Red-mediated homologous DNA recombination. 著者: Timothy P Newing / Jodi L Brewster / Lucy J Fitschen / James C Bouwer / Nikolas P Johnston / Haibo Yu / Gökhan Tolun /  要旨: The Redβ protein of the bacteriophage λ red recombination system is a model annealase which catalyzes single-strand annealing homologous DNA recombination. Here we present the structure of a ...The Redβ protein of the bacteriophage λ red recombination system is a model annealase which catalyzes single-strand annealing homologous DNA recombination. Here we present the structure of a helical oligomeric annealing intermediate of Redβ, consisting of N-terminal residues 1-177 bound to two complementary 27mer oligonucleotides, determined via cryogenic electron microscopy (cryo-EM) to a final resolution of 3.3 Å. The structure reveals a continuous binding groove which positions and stabilizes complementary DNA strands in a planar orientation to facilitate base pairing via a network of hydrogen bonding. Definition of the inter-subunit interface provides a structural basis for the propensity of Redβ to oligomerize into functionally significant long helical filaments, a trait shared by most annealases. Our cryo-EM structure and molecular dynamics simulations suggest that residues 133-138 form a flexible loop which modulates access to the binding groove. More than half a century after its discovery, this combination of structural and computational observations has allowed us to propose molecular mechanisms for the actions of the model annealase Redβ, a defining member of the Redβ/RecT protein family. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26566.map.gz emd_26566.map.gz | 106.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26566-v30.xml emd-26566-v30.xml emd-26566.xml emd-26566.xml | 21.6 KB 21.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26566_fsc.xml emd_26566_fsc.xml | 15.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26566.png emd_26566.png | 175.3 KB | ||
| マスクデータ |  emd_26566_msk_1.map emd_26566_msk_1.map | 343 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-26566.cif.gz emd-26566.cif.gz | 6.8 KB | ||
| その他 |  emd_26566_half_map_1.map.gz emd_26566_half_map_1.map.gz emd_26566_half_map_2.map.gz emd_26566_half_map_2.map.gz | 318.4 MB 318.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26566 http://ftp.pdbj.org/pub/emdb/structures/EMD-26566 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26566 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26566 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26566_validation.pdf.gz emd_26566_validation.pdf.gz | 1.3 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26566_full_validation.pdf.gz emd_26566_full_validation.pdf.gz | 1.3 MB | 表示 | |
| XML形式データ |  emd_26566_validation.xml.gz emd_26566_validation.xml.gz | 23.9 KB | 表示 | |
| CIF形式データ |  emd_26566_validation.cif.gz emd_26566_validation.cif.gz | 31.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26566 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26566 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26566 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26566 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ujlMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26566.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26566.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Symmetrised, sharpened map of the RedBeta177 helical assembly bound to dsDNA | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.84 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_26566_msk_1.map emd_26566_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unfiltered half map B of the RedBeta177 helical...
| ファイル | emd_26566_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered half map B of the RedBeta177 helical assembly bound to dsDNA | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unfiltered half map A of the RedBeta177 helical...
| ファイル | emd_26566_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered half map A of the RedBeta177 helical assembly bound to dsDNA | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RedBeta177 oligomeric helical assembly bound to two complementary...
| 全体 | 名称: RedBeta177 oligomeric helical assembly bound to two complementary 27mer ssDNA oligonucleotides |
|---|---|
| 要素 |
|
-超分子 #1: RedBeta177 oligomeric helical assembly bound to two complementary...
| 超分子 | 名称: RedBeta177 oligomeric helical assembly bound to two complementary 27mer ssDNA oligonucleotides タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Helical complex assembled through sequential addition of complementary oligonucleotides in a controlled environment |
|---|---|
| 分子量 | 理論値: 102.26 kDa/nm |
-超分子 #2: Red-beta annealase N-terminal domain
| 超分子 | 名称: Red-beta annealase N-terminal domain / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Escherichia virus Lambda (ウイルス) Escherichia virus Lambda (ウイルス) |
-超分子 #3: Template ssDNA
| 超分子 | 名称: Template ssDNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 / 詳細: 27mer ssDNA oligonucleotide |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #4: Complementary ssDNA
| 超分子 | 名称: Complementary ssDNA / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #3 / 詳細: 27mer ssDNA oligonucleotide |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Recombination protein bet
| 分子 | 名称: Recombination protein bet / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Escherichia virus Lambda (ウイルス) Escherichia virus Lambda (ウイルス) |
| 分子量 | 理論値: 21.275008 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSTALATLAG KLAERVGMDS VDPQELITTL RQTAFKGDAS DAQFIALLIV ANQYGLNPWT KEIYAFPDKQ NGIVPVVGVD GWSRIINEN QQFDGMDFEQ DNESCTCRIY RKDRNHPICV TEWMDECRRE PFKTREGREI TGPWQSHPKR MLRHKAMIQC A RLAFGFAG IYDKDEAERS SHHHHHH UniProtKB: Recombination protein bet |
-分子 #2: Template DNA
| 分子 | 名称: Template DNA / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 8.244295 KDa |
| 配列 | 文字列: (DT)(DG)(DC)(DA)(DG)(DC)(DA)(DG)(DC)(DT) (DT)(DT)(DA)(DC)(DC)(DA)(DT)(DC)(DT)(DG) (DC)(DC)(DG)(DC)(DT)(DG)(DG) |
-分子 #3: Complementary DNA
| 分子 | 名称: Complementary DNA / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 8.351386 KDa |
| 配列 | 文字列: (DC)(DC)(DA)(DG)(DC)(DG)(DG)(DC)(DA)(DG) (DA)(DT)(DG)(DG)(DT)(DA)(DA)(DA)(DG)(DC) (DT)(DG)(DC)(DT)(DG)(DC)(DA) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 濃度 | 2.6 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 50 / 前処理 - タイプ: GLOW DISCHARGE | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV 詳細: Sample loading volume ranged between 2 and 3 microlitres. Samples were blotted for 5 seconds prior to vitrification.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / 詳細: Installed but not used |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-50 / 撮影したグリッド数: 1 / 実像数: 4710 / 平均露光時間: 9.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 59500 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-7ujl: |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)