+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | ClpAP complex bound to ClpS N-terminal extension, class IIc | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | AAA+ protease / Adaptor / protein complex / CHAPERONE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報endopeptidase Clp / endopeptidase Clp complex / ATP-dependent peptidase activity / protein quality control for misfolded or incompletely synthesized proteins / protein unfolding / protein catabolic process / peptidase activity / cellular response to heat / ATPase binding / serine-type endopeptidase activity ...endopeptidase Clp / endopeptidase Clp complex / ATP-dependent peptidase activity / protein quality control for misfolded or incompletely synthesized proteins / protein unfolding / protein catabolic process / peptidase activity / cellular response to heat / ATPase binding / serine-type endopeptidase activity / ATP hydrolysis activity / proteolysis / ATP binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.24 Å | |||||||||
 データ登録者 データ登録者 | Kim S / Fei X / Sauer RT / Baker TA | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2022 ジャーナル: Nat Struct Mol Biol / 年: 2022タイトル: AAA+ protease-adaptor structures reveal altered conformations and ring specialization. 著者: Sora Kim / Xue Fei / Robert T Sauer / Tania A Baker /  要旨: ClpAP, a two-ring AAA+ protease, degrades N-end-rule proteins bound by the ClpS adaptor. Here we present high-resolution cryo-EM structures of Escherichia coli ClpAPS complexes, showing how ClpA pore ...ClpAP, a two-ring AAA+ protease, degrades N-end-rule proteins bound by the ClpS adaptor. Here we present high-resolution cryo-EM structures of Escherichia coli ClpAPS complexes, showing how ClpA pore loops interact with the ClpS N-terminal extension (NTE), which is normally intrinsically disordered. In two classes, the NTE is bound by a spiral of pore-1 and pore-2 loops in a manner similar to substrate-polypeptide binding by many AAA+ unfoldases. Kinetic studies reveal that pore-2 loops of the ClpA D1 ring catalyze the protein remodeling required for substrate delivery by ClpS. In a third class, D2 pore-1 loops are rotated, tucked away from the channel and do not bind the NTE, demonstrating asymmetry in engagement by the D1 and D2 rings. These studies show additional structures and functions for key AAA+ elements. Pore-loop tucking may be used broadly by AAA+ unfoldases, for example, during enzyme pausing/unloading. #1:  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: ClpAP AAA+ Protease Adaptor Structures Reveal Pore-Loop Specialization 著者: Kim S / Fei X / Sauer RT / Baker TA | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26558.map.gz emd_26558.map.gz | 22.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26558-v30.xml emd-26558-v30.xml emd-26558.xml emd-26558.xml | 16.7 KB 16.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_26558.png emd_26558.png | 80.9 KB | ||
| Filedesc metadata |  emd-26558.cif.gz emd-26558.cif.gz | 6.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26558 http://ftp.pdbj.org/pub/emdb/structures/EMD-26558 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26558 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26558 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26558.map.gz / 形式: CCP4 / 大きさ: 24.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26558.map.gz / 形式: CCP4 / 大きさ: 24.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.87 Å | ||||||||||||||||||||||||||||||||||||
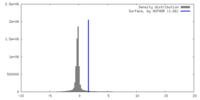
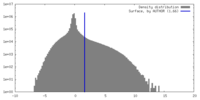
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : AAA+ protease complex ClpAP bound to adaptor protein ClpS and GFP
| 全体 | 名称: AAA+ protease complex ClpAP bound to adaptor protein ClpS and GFP |
|---|---|
| 要素 |
|
-超分子 #1: AAA+ protease complex ClpAP bound to adaptor protein ClpS and GFP
| 超分子 | 名称: AAA+ protease complex ClpAP bound to adaptor protein ClpS and GFP タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: ATP-dependent Clp protease ATP-binding subunit ClpA
| 分子 | 名称: ATP-dependent Clp protease ATP-binding subunit ClpA / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 84.291758 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLNQELELSL NMAFARAREH RHEFMTVEHL LLALLSNPSA REALEACSVD LVALRQELEA FIEQTTPVLP ASEEERDTQP TLSFQRVLQ RAVFHVQSSG RNEVTGANVL VAIFSEQESQ AAYLLRKHEV SRLDVVNFIS HGTRKDEPTQ SSDPGSQPNS E EQAGGEER ...文字列: MLNQELELSL NMAFARAREH RHEFMTVEHL LLALLSNPSA REALEACSVD LVALRQELEA FIEQTTPVLP ASEEERDTQP TLSFQRVLQ RAVFHVQSSG RNEVTGANVL VAIFSEQESQ AAYLLRKHEV SRLDVVNFIS HGTRKDEPTQ SSDPGSQPNS E EQAGGEER TENFTTNLNQ LARVGGIDPL IGREKELERA IQVLCRRRKN NPLLVGESGV GKTAIAEGLA WRIVQGDVPE VM ADCTIYS LDIGSLLAGT KYRGDFEKRF KALLKQLEQD TNSILFIDEI HTIIGAGAAS GGQVDAANLI KPLLSSGKIR VIG STTYQE FSNIFEKDRA LARRFQKIDI TEPSIEETVQ IINGLKPKYE AHHDVRYTAK AVRAAVELAV KYINDRHLPD KAID VIDEA GARARLMPVS KRKKTVNVAD IESVVARIAR IPEKSVSQSD RDTLKNLGDR LKMLVFGQDK AIEALTEAIK MARAG LGHE HKPVGSFLFA GPTGVGKTEV TVQLSKALGI ELLRFDMSEY MERHTVSRLI GAPPGYVGFD QGGLLTDAVI KHPHAV LLL DEIEKAHPDV FNILLQVMDN GTLTDNNGRK ADFRNVVLVM TTNAGVRETE RKSIGLIHQD NSTDAMEEIK KIFTPEF RN RLDNIIWFDH LSTDVIHQVV DKFIVELQVQ LDQKGVSLEV SQEARNWLAE KGYDRAMGAR PMARVIQDNL KKPLANEL L FGSLVDGGQV TVALDKEKNE LTYGFQSAQK HKAEAAH UniProtKB: ATP-dependent Clp protease ATP-binding subunit ClpA |
-分子 #2: ATP-dependent Clp protease adapter protein ClpS
| 分子 | 名称: ATP-dependent Clp protease adapter protein ClpS / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 12.193038 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGKTNDWLDF DQLAEEKVRD ALKPPSMYKV ILVNDDYTPM EFVIDVLQKF FSYDVERATQ LMLAVHYQGK AICGVFTAEV AETKVAMVN KYARENEHPL LCTLEKA UniProtKB: ATP-dependent Clp protease adapter protein ClpS |
-分子 #3: ATP-dependent Clp protease proteolytic subunit
| 分子 | 名称: ATP-dependent Clp protease proteolytic subunit / タイプ: protein_or_peptide / ID: 3 / コピー数: 7 / 光学異性体: LEVO / EC番号: endopeptidase Clp |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 22.660018 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ALVPMVIEQT SRGERSFDIY SRLLKERVIF LTGQVEDHMA NLIVAQMLFL EAENPEKDIY LYINSPGGVI TAGMSIYDTM QFIKPDVST ICMGQAASMG AFLLTAGAKG KRFCLPNSRV MIHQPLGGYQ GQATDIEIHA REILKVKGRM NELMALHTGQ S LEQIERDT ...文字列: ALVPMVIEQT SRGERSFDIY SRLLKERVIF LTGQVEDHMA NLIVAQMLFL EAENPEKDIY LYINSPGGVI TAGMSIYDTM QFIKPDVST ICMGQAASMG AFLLTAGAKG KRFCLPNSRV MIHQPLGGYQ GQATDIEIHA REILKVKGRM NELMALHTGQ S LEQIERDT ERDRFLSAPE AVEYGLVDSI LTHRNRSHHH HHH UniProtKB: ATP-dependent Clp protease proteolytic subunit |
-分子 #4: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 4 / コピー数: 9 / 式: AGS |
|---|---|
| 分子量 | 理論値: 523.247 Da |
| Chemical component information |  ChemComp-AGS: |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 10 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #6: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 6 / コピー数: 3 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 34.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): -2.5 µm / 最小 デフォーカス(公称値): -0.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-7uiz: |
 ムービー
ムービー コントローラー
コントローラー


















 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)