+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SARS-CoV-2 spike in complex with multivalent miniprotein inhibitor FUS31-G10 (2RBDs open) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.57 Å | |||||||||
 データ登録者 データ登録者 | Park YJ / Seattle Structural Genomics Center for Infectious Disease (SSGCID) / Veesler D | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Transl Med / 年: 2022 ジャーナル: Sci Transl Med / 年: 2022タイトル: Multivalent designed proteins neutralize SARS-CoV-2 variants of concern and confer protection against infection in mice. 著者: Andrew C Hunt / James Brett Case / Young-Jun Park / Longxing Cao / Kejia Wu / Alexandra C Walls / Zhuoming Liu / John E Bowen / Hsien-Wei Yeh / Shally Saini / Louisa Helms / Yan Ting Zhao / ...著者: Andrew C Hunt / James Brett Case / Young-Jun Park / Longxing Cao / Kejia Wu / Alexandra C Walls / Zhuoming Liu / John E Bowen / Hsien-Wei Yeh / Shally Saini / Louisa Helms / Yan Ting Zhao / Tien-Ying Hsiang / Tyler N Starr / Inna Goreshnik / Lisa Kozodoy / Lauren Carter / Rashmi Ravichandran / Lydia B Green / Wadim L Matochko / Christy A Thomson / Bastian Vögeli / Antje Krüger / Laura A VanBlargan / Rita E Chen / Baoling Ying / Adam L Bailey / Natasha M Kafai / Scott E Boyken / Ajasja Ljubetič / Natasha Edman / George Ueda / Cameron M Chow / Max Johnson / Amin Addetia / Mary-Jane Navarro / Nuttada Panpradist / Michael Gale / Benjamin S Freedman / Jesse D Bloom / Hannele Ruohola-Baker / Sean P J Whelan / Lance Stewart / Michael S Diamond / David Veesler / Michael C Jewett / David Baker /    要旨: New variants of severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) continue to arise and prolong the coronavirus disease 2019 (COVID-19) pandemic. Here, we used a cell-free expression ...New variants of severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) continue to arise and prolong the coronavirus disease 2019 (COVID-19) pandemic. Here, we used a cell-free expression workflow to rapidly screen and optimize constructs containing multiple computationally designed miniprotein inhibitors of SARS-CoV-2. We found the broadest efficacy was achieved with a homotrimeric version of the 75-residue angiotensin-converting enzyme 2 (ACE2) mimic AHB2 (TRI2-2) designed to geometrically match the trimeric spike architecture. Consistent with the design model, in the cryo-electron microscopy structure TRI2-2 forms a tripod at the apex of the spike protein that engaged all three receptor binding domains simultaneously. TRI2-2 neutralized Omicron (B.1.1.529), Delta (B.1.617.2), and all other variants tested with greater potency than the monoclonal antibodies used clinically for the treatment of COVID-19. TRI2-2 also conferred prophylactic and therapeutic protection against SARS-CoV-2 challenge when administered intranasally in mice. Designed miniprotein receptor mimics geometrically arrayed to match pathogen receptor binding sites could be a widely applicable antiviral therapeutic strategy with advantages over antibodies in greater resistance to viral escape and antigenic drift, and advantages over native receptor traps in lower chances of autoimmune responses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26509.map.gz emd_26509.map.gz | 230 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26509-v30.xml emd-26509-v30.xml emd-26509.xml emd-26509.xml | 22.3 KB 22.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_26509.png emd_26509.png | 45.1 KB | ||
| その他 |  emd_26509_additional_1.map.gz emd_26509_additional_1.map.gz emd_26509_half_map_1.map.gz emd_26509_half_map_1.map.gz emd_26509_half_map_2.map.gz emd_26509_half_map_2.map.gz | 121.6 MB 226.3 MB 226.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26509 http://ftp.pdbj.org/pub/emdb/structures/EMD-26509 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26509 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26509 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26509_validation.pdf.gz emd_26509_validation.pdf.gz | 1017.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26509_full_validation.pdf.gz emd_26509_full_validation.pdf.gz | 1017.4 KB | 表示 | |
| XML形式データ |  emd_26509_validation.xml.gz emd_26509_validation.xml.gz | 15.7 KB | 表示 | |
| CIF形式データ |  emd_26509_validation.cif.gz emd_26509_validation.cif.gz | 18.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26509 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26509 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26509 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26509 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26509.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26509.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_26509_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
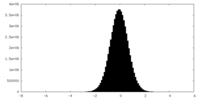
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_26509_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
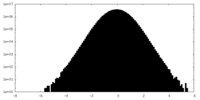
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_26509_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV-2 spike in complex with multivalent miniprotein inhibito...
| 全体 | 名称: SARS-CoV-2 spike in complex with multivalent miniprotein inhibitor FUS31-G10 (2RBDs open) |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 spike in complex with multivalent miniprotein inhibito...
| 超分子 | 名称: SARS-CoV-2 spike in complex with multivalent miniprotein inhibitor FUS31-G10 (2RBDs open) タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #2: SARS-CoV-2 spike
| 超分子 | 名称: SARS-CoV-2 spike / タイプ: complex / キメラ: Yes / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: Multivalent miniprotein inhibitor FUS31-G10
| 超分子 | 名称: Multivalent miniprotein inhibitor FUS31-G10 / タイプ: complex / キメラ: Yes / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種: Synthetic construct (人工物) |
| 組換発現 | 生物種:  |
-分子 #1: SARS-CoV-2 Spike
| 分子 | 名称: SARS-CoV-2 Spike / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDG VYFASTEKSN IIRGWIFGTT LDSKTQSLLI VNNATNVVIK VCEFQFCNDP FLGVYYHKNN KSWMESEFRV Y SSANNCTF ...文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDG VYFASTEKSN IIRGWIFGTT LDSKTQSLLI VNNATNVVIK VCEFQFCNDP FLGVYYHKNN KSWMESEFRV Y SSANNCTF EYVSQPFLMD LEGKQGNFKN LREFVFKNID GYFKIYSKHT PINLVRDLPQ GFSALEPLVD LPIGINITRF QT LLALHRS YLTPGDSSSG WTAGAAAYYV GYLQPRTFLL KYNENGTITD AVDCALDPLS ETKCTLKSFT VEKGIYQTSN FRV QPTESI VRFPNITNLC PFGEVFNATR FASVYAWNRK RISNCVADYS VLYNSASFST FKCYGVSPTK LNDLCFTNVY ADSF VIRGD EVRQIAPGQT GKIADYNYKL PDDFTGCVIA WNSNNLDSKV GGNYNYLYRL FRKSNLKPFE RDISTEIYQA GSTPC NGVE GFNCYFPLQS YGFQPTNGVG YQPYRVVVLS FELLHAPATV CGPKKSTNLV KNKCVNFNFN GLTGTGVLTE SNKKFL PFQ QFGRDIADTT DAVRDPQTLE ILDITPCSFG GVSVITPGTN TSNQVAVLYQ DVNCTEVPVA IHADQLTPTW RVYSTGS NV FQTRAGCLIG AEHVNNSYEC DIPIGAGICA SYQTQTNSPG SASSVASQSI IAYTMSLGAE NSVAYSNNSI AIPTNFTI S VTTEILPVSM TKTSVDCTMY ICGDSTECSN LLLQYGSFCT QLNRALTGIA VEQDKNTQEV FAQVKQIYKT PPIKDFGGF NFSQILPDPS KPSKRSPIED LLFNKVTLAD AGFIKQYGDC LGDIAARDLI CAQKFNGLTV LPPLLTDEMI AQYTSALLAG TITSGWTFG AGPALQIPFP MQMAYRFNGI GVTQNVLYEN QKLIANQFNS AIGKIQDSLS STPSALGKLQ DVVNQNAQAL N TLVKQLSS NFGAISSVLN DILSRLDPPE AEVQIDRLIT GRLQSLQTYV TQQLIRAAEI RASANLAATK MSECVLGQSK RV DFCGKGY HLMSFPQSAP HGVVFLHVTY VPAQEKNFTT APAICHDGKA HFPREGVFVS NGTHWFVTQR NFYEPQIITT DNT FVSGNC DVVIGIVNNT VYDPLQPELD SFKEELDKYF KNHTSPDVDL GDISGINASV VNIQKEIDRL NEVAKNLNES LIDL QELGK YEQGSGYIPE APRDGQAYVR KDGEWVLLST FLGRSLEVLF QGPGHHHHHH HHSAWSHPQF EKGGGSGGGG SGGSA WSHP QFEK |
-分子 #2: Multivalent miniprotein inhibitor FUS31-G10
| 分子 | 名称: Multivalent miniprotein inhibitor FUS31-G10 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: Synthetic construct (人工物) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEKKI SAWS HPQFEK GGG SGGGSGGSAW SHPQFEKGGS GSSG NLDEL HMQMTDLVYE ALHFAKDEEF QKHVFQLFEK ATKAYKNKDR QKLEKVVEEL KELLERLLS GGGSGGGSGG DKENVLQKI YEIMKELERL GHAEASMQVS DLIYEFMKTK DENLLEEAER LLEEVKR |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 70.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER / 詳細: cryoSPARC ab initio |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.57 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC / 使用した粒子像数: 9733 |
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)