+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the DU422 SOSIP.664 trimer in complex with neutralizing antibody Fab fragments 10-1074 and BG24 | ||||||||||||
 マップデータ マップデータ | Sharpened map | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | broadly neutralizing antibody / bNAb / HIV-1 / CD4 binding site / VH1-2 / VRC01-class / antiviral protein / VIRAL PROTEIN-IMMUNE SYSTEM complex | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated perturbation of host defense response / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell ...symbiont-mediated perturbation of host defense response / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | ||||||||||||
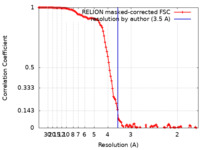
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | ||||||||||||
 データ登録者 データ登録者 | Barnes CO / Bjorkman PJ | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: A naturally arising broad and potent CD4-binding site antibody with low somatic mutation. 著者: Christopher O Barnes / Till Schoofs / Priyanthi N P Gnanapragasam / Jovana Golijanin / Kathryn E Huey-Tubman / Henning Gruell / Philipp Schommers / Nina Suh-Toma / Yu Erica Lee / Julio C ...著者: Christopher O Barnes / Till Schoofs / Priyanthi N P Gnanapragasam / Jovana Golijanin / Kathryn E Huey-Tubman / Henning Gruell / Philipp Schommers / Nina Suh-Toma / Yu Erica Lee / Julio C Cetrulo Lorenzi / Alicja Piechocka-Trocha / Johannes F Scheid / Anthony P West / Bruce D Walker / Michael S Seaman / Florian Klein / Michel C Nussenzweig / Pamela J Bjorkman /   要旨: The induction of broadly neutralizing antibodies (bNAbs) is a potential strategy for a vaccine against HIV-1. However, most bNAbs exhibit features such as unusually high somatic hypermutation, ...The induction of broadly neutralizing antibodies (bNAbs) is a potential strategy for a vaccine against HIV-1. However, most bNAbs exhibit features such as unusually high somatic hypermutation, including insertions and deletions, which make their induction challenging. VRC01-class bNAbs not only exhibit extraordinary breadth and potency but also rank among the most highly somatically mutated bNAbs. Here, we describe a VRC01-class antibody isolated from a viremic controller, BG24, that is much less mutated than most relatives of its class while achieving comparable breadth and potency. A 3.8-Å x-ray crystal structure of a BG24-BG505 Env trimer complex revealed conserved contacts at the gp120 interface characteristic of the VRC01-class Abs, despite lacking common CDR3 sequence motifs. The existence of moderately mutated CD4-binding site (CD4bs) bNAbs such as BG24 provides a simpler blueprint for CD4bs antibody induction by a vaccine, raising the prospect that such an induction might be feasible with a germline-targeting approach. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26443.map.gz emd_26443.map.gz | 15.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26443-v30.xml emd-26443-v30.xml emd-26443.xml emd-26443.xml | 25.4 KB 25.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26443_fsc.xml emd_26443_fsc.xml | 12 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26443.png emd_26443.png | 61 KB | ||
| Filedesc metadata |  emd-26443.cif.gz emd-26443.cif.gz | 7.7 KB | ||
| その他 |  emd_26443_half_map_1.map.gz emd_26443_half_map_1.map.gz emd_26443_half_map_2.map.gz emd_26443_half_map_2.map.gz | 114.2 MB 114.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26443 http://ftp.pdbj.org/pub/emdb/structures/EMD-26443 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26443 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26443 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26443_validation.pdf.gz emd_26443_validation.pdf.gz | 838.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26443_full_validation.pdf.gz emd_26443_full_validation.pdf.gz | 838.2 KB | 表示 | |
| XML形式データ |  emd_26443_validation.xml.gz emd_26443_validation.xml.gz | 19.4 KB | 表示 | |
| CIF形式データ |  emd_26443_validation.cif.gz emd_26443_validation.cif.gz | 25.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26443 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26443 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26443 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26443 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ucgMC  7uceC  7ucfC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26443.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26443.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.869 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_26443_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
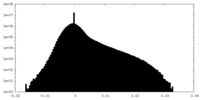
| 投影像・断面図 |
| ||||||||||||
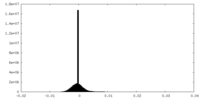
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_26443_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
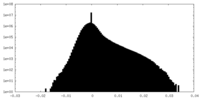
| 投影像・断面図 |
| ||||||||||||
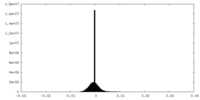
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of DU422 SOSIP trimer bound to 10-1074 and BG24 Fab fragments
| 全体 | 名称: Complex of DU422 SOSIP trimer bound to 10-1074 and BG24 Fab fragments |
|---|---|
| 要素 |
|
-超分子 #1: Complex of DU422 SOSIP trimer bound to 10-1074 and BG24 Fab fragments
| 超分子 | 名称: Complex of DU422 SOSIP trimer bound to 10-1074 and BG24 Fab fragments タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 650 KDa |
-分子 #1: Envelope glycoprotein gp41
| 分子 | 名称: Envelope glycoprotein gp41 / タイプ: protein_or_peptide / ID: 1 / 詳細: Env mimic DU422 SOSIP.664 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 17.104527 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AVGLGAVLLG FLGAAGSTMG AASMTLTVQA RQLLSGIVQQ QSNLLRAPEA QQHLLQLTVW GIKQLQTRVL AIERYLKDQQ LLGLWGCSG KLICCTAVPW NSSWSNKSLG DIWDNMTWMQ WDREISNYTN TIFRLLEESQ NQQEKNEKDL LALD UniProtKB: Envelope glycoprotein gp160 |
-分子 #2: BG24 CDRH2-v2 Fab heavy chain
| 分子 | 名称: BG24 CDRH2-v2 Fab heavy chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.49859 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLVQSRAE VKKPGASVKV SCEASGYNFV DHYIHWVRQA PGQRPQWVGW MNPQWGQVNY ARTFQGRVTM TRDTSIDTAY MQLNRLTSG DTAVYYCATQ VKLDSSAGYP FDIWGQGTMV TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA ...文字列: QVQLVQSRAE VKKPGASVKV SCEASGYNFV DHYIHWVRQA PGQRPQWVGW MNPQWGQVNY ARTFQGRVTM TRDTSIDTAY MQLNRLTSG DTAVYYCATQ VKLDSSAGYP FDIWGQGTMV TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA LTSGVHTFPA VLQSSGLYSL SSVVTVPSSS LGTQTYICNV NHKPSNTKVD KKVEPKSCDK HHHHHH |
-分子 #3: BG24 light chain
| 分子 | 名称: BG24 light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 21.925209 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QSALTQPRSV SGSPGQSVNI SCTGAYSGLG WYQQHPGRAP KLIIYEVNRR PSGVSDRFSG SKSGNTASLT ISGLRTEDEA DYFCSAFEY FGEGTKLTVL SQPKAAPSVT LFPPSSEELQ ANKATLVCLI SDFYPGAVTV AWKADSSPVK AGVETTTPSK Q SNNKYAAS ...文字列: QSALTQPRSV SGSPGQSVNI SCTGAYSGLG WYQQHPGRAP KLIIYEVNRR PSGVSDRFSG SKSGNTASLT ISGLRTEDEA DYFCSAFEY FGEGTKLTVL SQPKAAPSVT LFPPSSEELQ ANKATLVCLI SDFYPGAVTV AWKADSSPVK AGVETTTPSK Q SNNKYAAS SYLSLTPEQW KSHRSYSCQV THEGSTVEKT VAPTECS |
-分子 #4: Envelope glycoprotein gp160
| 分子 | 名称: Envelope glycoprotein gp160 / タイプ: protein_or_peptide / ID: 4 / 詳細: Env mimic DU422 SOSIP.664 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 56.99998 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGAENLDLW VTVYYGVPVW KEAKTTLFCA SDAKAYDKEV RNVWATHACV PTDPNPQEI VLENVTENFN MWKNDMVDQM HEDIISLWDQ SLKPCVKLTP LCVTLNCKNV NISANANATA TLNSSMNGEI K NCSFNTTT ...文字列: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGAENLDLW VTVYYGVPVW KEAKTTLFCA SDAKAYDKEV RNVWATHACV PTDPNPQEI VLENVTENFN MWKNDMVDQM HEDIISLWDQ SLKPCVKLTP LCVTLNCKNV NISANANATA TLNSSMNGEI K NCSFNTTT ELRDKKQKVY ALFYKPDVVP LNGGEHNETG EYILINCNSS TITQACPKVS FDPIPIHYCA PAGYAILKCN NK TFNGTGP CNNVSTVQCT HGIKPVVSTQ LLLNGSLAEE EIIVRSENLT NNIKTIIVHL NKSVEINCTR PNNNTRKSVR IGP GQWFYA TGEIIGDIRE AHCNISRETW NSTLIQVKEK LREHYNKTIK FEPSSGGDLE VTTHSFNCRG EFFYCNTTKL FNET KLFNE SEYVDNKTII LPCRIKQIIN MWQEVGRAMY APPIEGNITC KSNITGLLLT WDGGENSTEG VFRPGGGNMK DNWRS ELYK YKVVEIKPLG VAPTKCKRKV VGR UniProtKB: Envelope glycoprotein gp160 |
-分子 #5: 10-1074 Fab heavy chain
| 分子 | 名称: 10-1074 Fab heavy chain / タイプ: protein_or_peptide / ID: 5 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 26.389465 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLQESGPG LVKPSETLSV TCSVSGDSMN NYYWTWIRQS PGKGLEWIGY ISDRESATYN PSLNSRVVIS RDTSKNQLSL KLNSVTPAD TAVYYCATAR RGQRIYGVVS FGEFFYYYSM DVWGKGTTVT VSSASTKGPS VFPLAPSSKS TSGGTAALGC L VKDYFPEP ...文字列: QVQLQESGPG LVKPSETLSV TCSVSGDSMN NYYWTWIRQS PGKGLEWIGY ISDRESATYN PSLNSRVVIS RDTSKNQLSL KLNSVTPAD TAVYYCATAR RGQRIYGVVS FGEFFYYYSM DVWGKGTTVT VSSASTKGPS VFPLAPSSKS TSGGTAALGC L VKDYFPEP VTVSWNSGAL TSGVHTFPAV LQSSGLYSLS SVVTVPSSSL GTQTYICNVN HKPSNTKVDK RVEPKSCDKH HH HHH |
-分子 #6: 10-1074 light chain
| 分子 | 名称: 10-1074 light chain / タイプ: protein_or_peptide / ID: 6 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.708293 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: SYVRPLSVAL GETARISCGR QALGSRAVQW YQHRPGQAPI LLIYNNQDRP SGIPERFSGT PDINFGTRAT LTISGVEAGD EADYYCHMW DSRSGFSWSF GGATRLTVLG QPKAAPSVFI FPPSDEQLKS GTASVVCLLN NFYPREAKVQ WKVDNALQSG N SQESVTEQ ...文字列: SYVRPLSVAL GETARISCGR QALGSRAVQW YQHRPGQAPI LLIYNNQDRP SGIPERFSGT PDINFGTRAT LTISGVEAGD EADYYCHMW DSRSGFSWSF GGATRLTVLG QPKAAPSVFI FPPSDEQLKS GTASVVCLLN NFYPREAKVQ WKVDNALQSG N SQESVTEQ DSKDSTYSLS STLTLSKADY EKHKVYACEV THQGLSSPVT KSFNRGEC |
-分子 #10: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 10 / コピー数: 21 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 1989 / 平均露光時間: 3.5 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)