+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Rabies virus glycoprotein pre-fusion trimer in complex with neutralizing antibody RVA122 | ||||||||||||
 マップデータ マップデータ | Rabies virus glycoprotein pre-fusion trimer in complex with neutralizing antibody RVA122 | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Rabies virus glycoprotein / antibodies / viral fusion protein / VIRAL PROTEIN / VIRAL PROTEIN-IMMUNE SYSTEM complex | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral membrane / entry receptor-mediated virion attachment to host cell / fusion of virus membrane with host endosome membrane / viral envelope / virion membrane / membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Rabies virus (ウイルス) / Rabies virus (ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
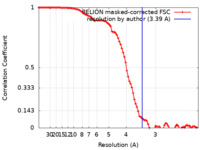
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.39 Å | ||||||||||||
 データ登録者 データ登録者 | Callaway HM / Zyla D / Larrous F / Dias de Melo G / Hastie KM / Avalos RD / Agarwal A / Bouhry H / Corti D / Saphire EO | ||||||||||||
| 資金援助 |  米国, 米国,  スイス, 3件 スイス, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Structure of the rabies virus glycoprotein trimer bound to a prefusion-specific neutralizing antibody. 著者: Heather M Callaway / Dawid Zyla / Florence Larrous / Guilherme Dias de Melo / Kathryn M Hastie / Ruben Diaz Avalos / Alyssa Agarwal / Davide Corti / Hervé Bourhy / Erica Ollmann Saphire /    要旨: Rabies infection is nearly 100% lethal if untreated and kills more than 50,000 people annually, many of them children. Existing rabies vaccines target the rabies virus glycoprotein (RABV-G) but ...Rabies infection is nearly 100% lethal if untreated and kills more than 50,000 people annually, many of them children. Existing rabies vaccines target the rabies virus glycoprotein (RABV-G) but generate short-lived immune responses, likely because the protein is heterogeneous under physiological conditions. Here, we report the 3.39 Å cryo-electron microscopy structure of trimeric, prefusion RABV-G complexed with RVA122, a potently neutralizing human antibody. RVA122 binds to a quaternary epitope at the top of RABV-G, bridging domains and stabilizing RABV-G protomers in a prefusion state. RABV-G trimerization involves side-to-side interactions between the central α helix and adjacent loops, rather than contacts between central helices, and interactions among the fusion loops at the glycoprotein base. These results provide a basis from which to develop improved rabies vaccines based on RABV-G stabilized in the prefusion conformation. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26397.map.gz emd_26397.map.gz | 48.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26397-v30.xml emd-26397-v30.xml emd-26397.xml emd-26397.xml | 20.6 KB 20.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26397_fsc.xml emd_26397_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26397.png emd_26397.png | 166.6 KB | ||
| マスクデータ |  emd_26397_msk_1.map emd_26397_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-26397.cif.gz emd-26397.cif.gz | 6.5 KB | ||
| その他 |  emd_26397_half_map_1.map.gz emd_26397_half_map_1.map.gz emd_26397_half_map_2.map.gz emd_26397_half_map_2.map.gz | 49.4 MB 49.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26397 http://ftp.pdbj.org/pub/emdb/structures/EMD-26397 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26397 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26397 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26397_validation.pdf.gz emd_26397_validation.pdf.gz | 801.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26397_full_validation.pdf.gz emd_26397_full_validation.pdf.gz | 800.7 KB | 表示 | |
| XML形式データ |  emd_26397_validation.xml.gz emd_26397_validation.xml.gz | 16.1 KB | 表示 | |
| CIF形式データ |  emd_26397_validation.cif.gz emd_26397_validation.cif.gz | 21.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26397 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26397 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26397 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26397 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26397.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26397.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Rabies virus glycoprotein pre-fusion trimer in complex with neutralizing antibody RVA122 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_26397_msk_1.map emd_26397_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1 for rabies virus glycoprotein pre-fusion...
| ファイル | emd_26397_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 for rabies virus glycoprotein pre-fusion trimer in complex with neutralizing antibody RVA122 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2 for rabies virus glycoprotein pre-fusion...
| ファイル | emd_26397_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 for rabies virus glycoprotein pre-fusion trimer in complex with neutralizing antibody RVA122 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Rabies virus glycoprotein pre-fusion trimer in complex with neutr...
| 全体 | 名称: Rabies virus glycoprotein pre-fusion trimer in complex with neutralizing antibody RVA122 |
|---|---|
| 要素 |
|
-超分子 #1: Rabies virus glycoprotein pre-fusion trimer in complex with neutr...
| 超分子 | 名称: Rabies virus glycoprotein pre-fusion trimer in complex with neutralizing antibody RVA122 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|
-分子 #1: Glycoprotein
| 分子 | 名称: Glycoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rabies virus (ウイルス) / 株: Pasteur vaccins / PV Rabies virus (ウイルス) / 株: Pasteur vaccins / PV |
| 分子量 | 理論値: 49.273547 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MVPQALLFVP LLVFPLCFGK FPIYTIPDKL GPWSPIDIHH LSCPNNLVVE DEGCTNLSGF SYMELKVGYI SAIKMNGFTC TGVVTEAET YTNFVGYVTT TFKRKHFRPT PDACRAAYNW KMAGDPRYEE SLHNPYPDYH WLRTVKTTKE SLVIISPSVA D LDPYDRSL ...文字列: MVPQALLFVP LLVFPLCFGK FPIYTIPDKL GPWSPIDIHH LSCPNNLVVE DEGCTNLSGF SYMELKVGYI SAIKMNGFTC TGVVTEAET YTNFVGYVTT TFKRKHFRPT PDACRAAYNW KMAGDPRYEE SLHNPYPDYH WLRTVKTTKE SLVIISPSVA D LDPYDRSL HSRVFPGGNC SGVAVSSTYC STNHDYTIWM PENPRLGMSC DIFTNSRGKR ASKGSETCGF VDERGLYKSL KG ACKLKLC GVLGLRLMDG TWVAMQTSNE TKWCPPGQLV NLHDFRSDEI EHLVVEELVK KREECLDALE SIMTTKSVSF RRL SHLRKL VPGFGKAYTI FNKTLMEADA HYKSVRTWNE IIPSKGCLRV GGRCHPHVNG VFFNGIILGP DGNVLIPEMQ SSLL QQHME LLVSSVIPLM HPLADPSTVF KNGDEAEDFV EVHL UniProtKB: Glycoprotein |
-分子 #2: RVA122 Fab Light Chain
| 分子 | 名称: RVA122 Fab Light Chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.44983 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QSVLTQSPSA SDTPGQRVTI SCSGSSSNIG SNYVYWYQQF PGTAPKLLIY KSDKRPSGVP DRFSGSTSGT SASLAISGLR SEDEADYYC AAWDNRLSGW LFGGGTKLTV LGTVAAPSVF IFPPSDEQLK SGTASVVCLL NNFYPREAKV QWKVDNALQS G NSQESVTE ...文字列: QSVLTQSPSA SDTPGQRVTI SCSGSSSNIG SNYVYWYQQF PGTAPKLLIY KSDKRPSGVP DRFSGSTSGT SASLAISGLR SEDEADYYC AAWDNRLSGW LFGGGTKLTV LGTVAAPSVF IFPPSDEQLK SGTASVVCLL NNFYPREAKV QWKVDNALQS G NSQESVTE QDSKDSTYSL SSTLTLSKAD YEKHKVYACE VTHQGLRSPV TKSFNRGEC |
-分子 #3: RVA122 Fab Heavy Chain
| 分子 | 名称: RVA122 Fab Heavy Chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 28.196096 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVHLQESGPG LVKPSETLSL TCTVSGDSMN NFYWGWIRQP AGKGLEWIGY IYYSGTTNYN PSLKSRVTIS IDTSKNQFSL KVNSVTAAD TAVYYCARDS GDYVSYYYYG MDVWGPGTTV TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA ...文字列: QVHLQESGPG LVKPSETLSL TCTVSGDSMN NFYWGWIRQP AGKGLEWIGY IYYSGTTNYN PSLKSRVTIS IDTSKNQFSL KVNSVTAAD TAVYYCARDS GDYVSYYYYG MDVWGPGTTV TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA LTSGVHTFPA VLQSSGLYSL SSVVTVPSSS LGTQTYICNV NHKPSNTKVD KKVEPKSCDL EVDDDDKAGW SH PQFEKGG GSGGGSGGGS WSHPQFEK |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 9 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: PBS pH 7.4 with 0.09mM LMNG | ||||||||||||||||||
| グリッド | モデル: C-flat-2/1 | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 85 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)