+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Alpha1/BetaB Heteromeric Glycine Receptor in Glycine-Bound State | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Glycine / Ion Channel / Ligand-Gated / Receptor / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Neurotransmitter receptors and postsynaptic signal transmission / extracellularly glycine-gated ion channel activity / extracellularly glycine-gated chloride channel activity / transmitter-gated monoatomic ion channel activity / regulation of neuron differentiation / ligand-gated monoatomic ion channel activity / glycine binding / cellular response to zinc ion / cellular response to ethanol / response to amino acid ...Neurotransmitter receptors and postsynaptic signal transmission / extracellularly glycine-gated ion channel activity / extracellularly glycine-gated chloride channel activity / transmitter-gated monoatomic ion channel activity / regulation of neuron differentiation / ligand-gated monoatomic ion channel activity / glycine binding / cellular response to zinc ion / cellular response to ethanol / response to amino acid / chloride channel complex / monoatomic ion transport / chloride transmembrane transport / central nervous system development / cellular response to amino acid stimulus / transmembrane signaling receptor activity / intracellular protein localization / perikaryon / postsynaptic membrane / dendrite / zinc ion binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
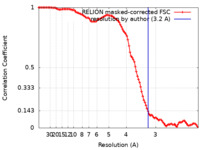
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Gibbs E / Chakrapani S / Kumar A | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Conformational transitions and allosteric modulation in a heteromeric glycine receptor 著者: Gibbs E / Klemm E / Seiferth D / Kumar A / Ilca SL / Biggin PC / Chakrapani S | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26141.map.gz emd_26141.map.gz | 52.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26141-v30.xml emd-26141-v30.xml emd-26141.xml emd-26141.xml | 23.3 KB 23.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26141_fsc.xml emd_26141_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26141.png emd_26141.png | 80.6 KB | ||
| マスクデータ |  emd_26141_msk_1.map emd_26141_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-26141.cif.gz emd-26141.cif.gz | 7 KB | ||
| その他 |  emd_26141_half_map_1.map.gz emd_26141_half_map_1.map.gz emd_26141_half_map_2.map.gz emd_26141_half_map_2.map.gz | 95.5 MB 95.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26141 http://ftp.pdbj.org/pub/emdb/structures/EMD-26141 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26141 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26141 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26141_validation.pdf.gz emd_26141_validation.pdf.gz | 836.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26141_full_validation.pdf.gz emd_26141_full_validation.pdf.gz | 835.7 KB | 表示 | |
| XML形式データ |  emd_26141_validation.xml.gz emd_26141_validation.xml.gz | 17.3 KB | 表示 | |
| CIF形式データ |  emd_26141_validation.cif.gz emd_26141_validation.cif.gz | 22.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26141 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26141 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26141 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26141 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7tviMC  7tu9C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26141.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26141.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_26141_msk_1.map emd_26141_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_26141_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_26141_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Zebrafish Alpha1 BetaB Heteromeric Glycine Receptor
| 全体 | 名称: Zebrafish Alpha1 BetaB Heteromeric Glycine Receptor |
|---|---|
| 要素 |
|
-超分子 #1: Zebrafish Alpha1 BetaB Heteromeric Glycine Receptor
| 超分子 | 名称: Zebrafish Alpha1 BetaB Heteromeric Glycine Receptor / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 250 KDa |
-分子 #1: Glycine receptor subunit alphaZ1
| 分子 | 名称: Glycine receptor subunit alphaZ1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 52.537598 KDa |
| 組換発現 | 生物種:  Spodoptera aff. frugiperda 1 BOLD-2017 (蝶・蛾) Spodoptera aff. frugiperda 1 BOLD-2017 (蝶・蛾) |
| 配列 | 文字列: MFALGIYLWE TIVFFSLAAS QQAAARKAAS PMPPSEFLDK LMGKVSGYDA RIRPNFKGPP VNVTCNIFIN SFGSIAETTM DYRVNIFLR QQWNDPRLAY SEYPDDSLDL DPSMLDSIWK PDLFFANEKG ANFHEVTTDN KLLRISKNGN VLYSIRITLV L ACPMDLKN ...文字列: MFALGIYLWE TIVFFSLAAS QQAAARKAAS PMPPSEFLDK LMGKVSGYDA RIRPNFKGPP VNVTCNIFIN SFGSIAETTM DYRVNIFLR QQWNDPRLAY SEYPDDSLDL DPSMLDSIWK PDLFFANEKG ANFHEVTTDN KLLRISKNGN VLYSIRITLV L ACPMDLKN FPMDVQTCIM QLESFGYTMN DLIFEWDEKG AVQVADGLTL PQFILKEEKD LRYCTKHYNT GKFTCIEARF HL ERQMGYY LIQMYIPSLL IVILSWVSFW INMDAAPARV GLGITTVLTM TTQSSGSRAS LPKVSYVKAI DIWMAVCLLF VFS ALLEYA AVNFIARQHK ELLRFQRRRR HLKEDEAGDG RFSFAAYGMG PACLQAKDGM AIKGNNNNAP TSTNPPEKTV EEMR KLFIS RAKRIDTVSR VAFPLVFLIF NIFYWITYKI IRSEDIHKQL VPRGSHHHHH HHH UniProtKB: Glycine receptor subunit alphaZ1 |
-分子 #2: Glycine receptor beta subunit 2
| 分子 | 名称: Glycine receptor beta subunit 2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 65.820281 KDa |
| 組換発現 | 生物種:  Spodoptera aff. frugiperda 1 BOLD-2017 (蝶・蛾) Spodoptera aff. frugiperda 1 BOLD-2017 (蝶・蛾) |
| 配列 | 文字列: MKALKVIFML LIICLWMEGG FTKEKSAKKW SHPQFEKGGG SGGGSGGGSW SHPQFEKGGG SGGGSGGGSW SHPQFEKGGG SGGGSGGGS WSHPQFEKEN LYFQGEKSAK KGKKKGKQVY CPSQLSSEDL ARVPANSTSN ILNKLLITYD PRIRPNFKGI P VEDRVNIF ...文字列: MKALKVIFML LIICLWMEGG FTKEKSAKKW SHPQFEKGGG SGGGSGGGSW SHPQFEKGGG SGGGSGGGSW SHPQFEKGGG SGGGSGGGS WSHPQFEKEN LYFQGEKSAK KGKKKGKQVY CPSQLSSEDL ARVPANSTSN ILNKLLITYD PRIRPNFKGI P VEDRVNIF INSFGSIQET TMDYRVNIFL RQRWNDPRLR LPQDFKSDSL TVDPKMFKCL WKPDLFFANE KSANFHDVTQ EN ILLFIFR NGDVLISMRL SVTLSCPLDL TLFPMDTQRC KMQLESFGYT TDDLQFMWQS GDPVQMDEIA LPQFDIKQED IEY GNCTKY YAGTGYYTCV EVIFTLRRQV GFYMMGVYAP TLLIVVLSWL SFWINPDASA ARVPLGILSV LSLSSECTSL ASEL PKVSY VKAIDIWLIA CLLFGFASLV EYAVVQVMLN SPKLLEAERA KIATKEKAEG KTPAKNTING MGSTPIHVST LQVTE TRCK KVCTSKSDLR TNDFSIVGSL PRDFELSNFD CYGKPIEVGS AFSKSQAKNN KKPPPPKPVI PSAAKRIDLY ARALFP FSF LFFNVIYWSV YLENLYFQGT ETSQVAPA UniProtKB: Glycine receptor subunit beta |
-分子 #3: GLYCINE
| 分子 | 名称: GLYCINE / タイプ: ligand / ID: 3 / コピー数: 5 / 式: GLY |
|---|---|
| 分子量 | 理論値: 75.067 Da |
| Chemical component information |  ChemComp-GLY: |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 6 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 5 sec. | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV | |||||||||||||||
| 詳細 | Single Particle |
- 電子顕微鏡法 #1
電子顕微鏡法 #1
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 撮影 | Image recording ID: 1 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.8 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 電子顕微鏡法 #1~
電子顕微鏡法 #1~
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 撮影 | Image recording ID: 2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.8 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)