+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | LH2-LH3 antenna in parallel configuration embedded in a nanodisc | ||||||||||||||||||
 マップデータ マップデータ | LH2-LH3 Antenna in parallel configuration | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | antenna / membrane protein / nanodisc / bacteriochlorophyll / PHOTOSYNTHESIS | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報organelle inner membrane / plasma membrane light-harvesting complex / bacteriochlorophyll binding / photosynthesis, light reaction / electron transporter, transferring electrons within the cyclic electron transport pathway of photosynthesis activity / metal ion binding / plasma membrane 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  Magnetospirillum molischianum (走磁性) Magnetospirillum molischianum (走磁性) | ||||||||||||||||||
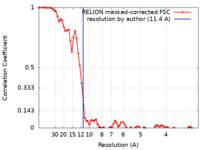
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 11.4 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Toporik H / Harris D | ||||||||||||||||||
| 資金援助 |  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: Elucidating interprotein energy transfer dynamics within the antenna network from purple bacteria. 著者: Dihao Wang / Olivia C Fiebig / Dvir Harris / Hila Toporik / Yi Ji / Chern Chuang / Muath Nairat / Ashley L Tong / John I Ogren / Stephanie M Hart / Jianshu Cao / James N Sturgis / Yuval Mazor ...著者: Dihao Wang / Olivia C Fiebig / Dvir Harris / Hila Toporik / Yi Ji / Chern Chuang / Muath Nairat / Ashley L Tong / John I Ogren / Stephanie M Hart / Jianshu Cao / James N Sturgis / Yuval Mazor / Gabriela S Schlau-Cohen /   要旨: In photosynthesis, absorbed light energy transfers through a network of antenna proteins with near-unity quantum efficiency to reach the reaction center, which initiates the downstream biochemical ...In photosynthesis, absorbed light energy transfers through a network of antenna proteins with near-unity quantum efficiency to reach the reaction center, which initiates the downstream biochemical reactions. While the energy transfer dynamics within individual antenna proteins have been extensively studied over the past decades, the dynamics between the proteins are poorly understood due to the heterogeneous organization of the network. Previously reported timescales averaged over such heterogeneity, obscuring individual interprotein energy transfer steps. Here, we isolated and interrogated interprotein energy transfer by embedding two variants of the primary antenna protein from purple bacteria, light-harvesting complex 2 (LH2), together into a near-native membrane disc, known as a nanodisc. We integrated ultrafast transient absorption spectroscopy, quantum dynamics simulations, and cryogenic electron microscopy to determine interprotein energy transfer timescales. By varying the diameter of the nanodiscs, we replicated a range of distances between the proteins. The closest distance possible between neighboring LH2, which is the most common in native membranes, is 25 Å and resulted in a timescale of 5.7 ps. Larger distances of 28 to 31 Å resulted in timescales of 10 to 14 ps. Corresponding simulations showed that the fast energy transfer steps between closely spaced LH2 increase transport distances by ∼15%. Overall, our results introduce a framework for well-controlled studies of interprotein energy transfer dynamics and suggest that protein pairs serve as the primary pathway for the efficient transport of solar energy. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26138.map.gz emd_26138.map.gz | 4.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26138-v30.xml emd-26138-v30.xml emd-26138.xml emd-26138.xml | 15 KB 15 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26138_fsc.xml emd_26138_fsc.xml | 7.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26138.png emd_26138.png | 18.7 KB | ||
| Filedesc metadata |  emd-26138.cif.gz emd-26138.cif.gz | 5.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26138 http://ftp.pdbj.org/pub/emdb/structures/EMD-26138 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26138 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26138 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26138_validation.pdf.gz emd_26138_validation.pdf.gz | 386.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26138_full_validation.pdf.gz emd_26138_full_validation.pdf.gz | 386.5 KB | 表示 | |
| XML形式データ |  emd_26138_validation.xml.gz emd_26138_validation.xml.gz | 9.4 KB | 表示 | |
| CIF形式データ |  emd_26138_validation.cif.gz emd_26138_validation.cif.gz | 12.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26138 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26138 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26138 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26138 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26138.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26138.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | LH2-LH3 Antenna in parallel configuration | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.6 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : LH2 and LH3 antennae
| 全体 | 名称: LH2 and LH3 antennae |
|---|---|
| 要素 |
|
-超分子 #1: LH2 and LH3 antennae
| 超分子 | 名称: LH2 and LH3 antennae / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  Magnetospirillum molischianum (走磁性) Magnetospirillum molischianum (走磁性) |
-分子 #1: Light-harvesting protein B-800/850 alpha chain
| 分子 | 名称: Light-harvesting protein B-800/850 alpha chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Magnetospirillum molischianum (走磁性) Magnetospirillum molischianum (走磁性) |
| 分子量 | 理論値: 6.076175 KDa |
| 配列 | 文字列: MSNPKDDYKI WLVINPSTWL PVIWIVATVV AIAVHAAVLA APGFNWIALG AAKSAAK UniProtKB: Light-harvesting protein B-800/850 alpha chain |
-分子 #2: Light-harvesting protein B-800/850 beta 1 chain
| 分子 | 名称: Light-harvesting protein B-800/850 beta 1 chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Magnetospirillum molischianum (走磁性) Magnetospirillum molischianum (走磁性) |
| 分子量 | 理論値: 4.920682 KDa |
| 配列 | 文字列: RSLSGLTEEE AIAVHDQFKT TFSAFIILAA VAHVLVWVWK PWF UniProtKB: Light-harvesting protein B-800/850 beta 1 chain |
-分子 #3: Light-harvesting protein B800-820 alpha chain
| 分子 | 名称: Light-harvesting protein B800-820 alpha chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Magnetospirillum molischianum (走磁性) Magnetospirillum molischianum (走磁性) |
| 分子量 | 理論値: 6.219315 KDa |
| 配列 | 文字列: MSNPKDDYKI WLVINPSTWL PVIWIVALLT AIAVHSFVLS VPGYNFLASA AAKTAAK UniProtKB: Light-harvesting protein B800-820 alpha chain |
-分子 #4: Light-harvesting protein B800-820 beta chain
| 分子 | 名称: Light-harvesting protein B800-820 beta chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Magnetospirillum molischianum (走磁性) Magnetospirillum molischianum (走磁性) |
| 分子量 | 理論値: 5.252069 KDa |
| 配列 | 文字列: MAERSLSGLT EEEAVAVHDQ FKTTFSAFIL LAAVAHVLVW IWKPWF UniProtKB: Light-harvesting protein B800-820 beta chain |
-分子 #5: BACTERIOCHLOROPHYLL A
| 分子 | 名称: BACTERIOCHLOROPHYLL A / タイプ: ligand / ID: 5 / コピー数: 48 / 式: BCL |
|---|---|
| 分子量 | 理論値: 911.504 Da |
| Chemical component information |  ChemComp-BCL: |
-分子 #6: LYCOPENE
| 分子 | 名称: LYCOPENE / タイプ: ligand / ID: 6 / コピー数: 16 / 式: LYC |
|---|---|
| 分子量 | 理論値: 536.873 Da |
| Chemical component information |  ChemComp-LYC: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 54.56 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.4 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)