+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of GSK682753A-bound EBI2/GPR183 | ||||||||||||
 マップデータ マップデータ | GSK682753A-bound EBI2/GPR183 | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | EBI2 / GPR183 / oxysterol / GPCR / signaling / immunity / immunocyte migration / IMMUNE SYSTEM | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of astrocyte chemotaxis / B cell activation involved in immune response / dendritic cell homeostasis / mature B cell differentiation involved in immune response / T follicular helper cell differentiation / T cell chemotaxis / oxysterol binding / Class A/1 (Rhodopsin-like receptors) / leukocyte chemotaxis / dendritic cell chemotaxis ...regulation of astrocyte chemotaxis / B cell activation involved in immune response / dendritic cell homeostasis / mature B cell differentiation involved in immune response / T follicular helper cell differentiation / T cell chemotaxis / oxysterol binding / Class A/1 (Rhodopsin-like receptors) / leukocyte chemotaxis / dendritic cell chemotaxis / humoral immune response / positive regulation of B cell proliferation / osteoclast differentiation / G protein-coupled receptor activity / G alpha (i) signalling events / adaptive immune response / positive regulation of ERK1 and ERK2 cascade / immune response / G protein-coupled receptor signaling pathway / nucleoplasm / plasma membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.98 Å | ||||||||||||
 データ登録者 データ登録者 | Chen H / Huang W | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2022 ジャーナル: Structure / 年: 2022タイトル: Structures of oxysterol sensor EBI2/GPR183, a key regulator of the immune response. 著者: Hongwen Chen / Weijiao Huang / Xiaochun Li /  要旨: Oxysterols induce the migration of B-lymphocytes and dendritic cells to interfollicular regions of lymphoid tissues through binding the EBI2 (GPR183) to stimulate effective adaptive immunity and ...Oxysterols induce the migration of B-lymphocytes and dendritic cells to interfollicular regions of lymphoid tissues through binding the EBI2 (GPR183) to stimulate effective adaptive immunity and antibody production during infection. Aberrant EBI2 signaling is implicated in inflammatory bowel disease, sclerosis, and infectious disease. Here, we report the cryo-EM structures of an EBI2-G signaling complex with its endogenous agonist 7α,25-OHC and that of an inactive EBI2 bound to the inverse agonist GSK682753A. These structures reveal an agonist binding site for the oxysterol and a potential ligand entrance site exposed to the lipid bilayer. Mutations within the oxysterol binding site and the Gα interface attenuate G protein signaling and abolish oxysterol-mediated cell migration indicating that G protein signaling directly involves in the oxysterol-EBI2 pathway. Together, these findings provide new insight into how EBI2 is activated by an oxysterol ligand and will facilitate the development of therapeutic approaches that target EBI2-linked diseases. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26135.map.gz emd_26135.map.gz | 53.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26135-v30.xml emd-26135-v30.xml emd-26135.xml emd-26135.xml | 19.4 KB 19.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_26135.png emd_26135.png | 70 KB | ||
| Filedesc metadata |  emd-26135.cif.gz emd-26135.cif.gz | 6.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26135 http://ftp.pdbj.org/pub/emdb/structures/EMD-26135 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26135 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26135 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7tuyMC  7tuzC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26135.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26135.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GSK682753A-bound EBI2/GPR183 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||
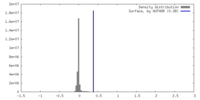
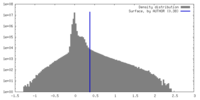
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : complex of GSK682753A-bound EBI2/GPR183-BRIL chimera with anti-BR...
| 全体 | 名称: complex of GSK682753A-bound EBI2/GPR183-BRIL chimera with anti-BRIL Fab and anti-Fab nanobody |
|---|---|
| 要素 |
|
-超分子 #1: complex of GSK682753A-bound EBI2/GPR183-BRIL chimera with anti-BR...
| 超分子 | 名称: complex of GSK682753A-bound EBI2/GPR183-BRIL chimera with anti-BRIL Fab and anti-Fab nanobody タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|
-超分子 #2: anti-BRIL Fab Heavy chain, anti-BRIL Fab Light chain
| 超分子 | 名称: anti-BRIL Fab Heavy chain, anti-BRIL Fab Light chain タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1, #3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: anti-Fab Nanobody
| 超分子 | 名称: anti-Fab Nanobody / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-超分子 #4: G-protein coupled receptor 183,Soluble cytochrome b562 fusion
| 超分子 | 名称: G-protein coupled receptor 183,Soluble cytochrome b562 fusion タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #4 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: anti-BRIL Fab Heavy chain
| 分子 | 名称: anti-BRIL Fab Heavy chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.321084 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF NVVDFSLHWV RQAPGKGLEW VAYISSSSGS TSYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC ARWGYWPGEP WWKAFDYWGQ GTLVTVSSAS TKGPSVFPLA PSSKSTSGGT AALGCLVKDY F PEPVTVSW ...文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF NVVDFSLHWV RQAPGKGLEW VAYISSSSGS TSYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC ARWGYWPGEP WWKAFDYWGQ GTLVTVSSAS TKGPSVFPLA PSSKSTSGGT AALGCLVKDY F PEPVTVSW NSGALTSGVH TFPAVLQSSG LYSLSSVVTV PSSSLGTQTY ICNVNHKPSN TKVDKKVEPK S |
-分子 #2: anti-Fab Nanobody
| 分子 | 名称: anti-Fab Nanobody / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 15.071431 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: HHHHHHGENL YFQGSQVQLQ ESGGGLVQPG GSLRLSCAAS GRTISRYAMS WFRQAPGKER EFVAVARRSG DGAFYADSVQ GRFTVSRDD AKNTVYLQMN SLKPEDTAVY YCAIDSDTFY SGSYDYWGQG TQVTVSS |
-分子 #3: anti-BRIL Fab Light chain
| 分子 | 名称: anti-BRIL Fab Light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.483062 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQYLYYSLVT FGQGTKVEIK RTVAAPSVFI FPPSDSQLKS GTASVVCLLN NFYPREAKVQ WKVDNALQSG N SQESVTEQ ...文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQYLYYSLVT FGQGTKVEIK RTVAAPSVFI FPPSDSQLKS GTASVVCLLN NFYPREAKVQ WKVDNALQSG N SQESVTEQ DSKDSTYSLS STLTLSKADY EKHKVYACEV THQGLSSPVT KSFNRGE |
-分子 #4: G-protein coupled receptor 183,Soluble cytochrome b562 fusion
| 分子 | 名称: G-protein coupled receptor 183,Soluble cytochrome b562 fusion タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 54.364539 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDIQMANNFT PPSATPQGND CDLYAHHSTA RIVMPLHYSL VFIIGLVGNL LALVVIVQNR KKINSTTLYS TNLVISDILF TTALPTRIA YYAMGFDWRI GDALCRITAL VFYINTYAGV NFMTCLSIDR FIAVVHPLRY NKIKRIEHAK GVCIFVWILV F AQTLPLLI ...文字列: MDIQMANNFT PPSATPQGND CDLYAHHSTA RIVMPLHYSL VFIIGLVGNL LALVVIVQNR KKINSTTLYS TNLVISDILF TTALPTRIA YYAMGFDWRI GDALCRITAL VFYINTYAGV NFMTCLSIDR FIAVVHPLRY NKIKRIEHAK GVCIFVWILV F AQTLPLLI NPMSKQEAER ITCMEYPNFE ETKSLPWILL GACFIGYVLP LIIILICYSQ ICCKLFRARR QLADLEDNWE TL NDNLKVI EKADNAAQVK DALTKMRAAA LDAQKATPPK LEDKSPDSPE MKDFRHGFDI LVGQIDDALK LANEGKVKEA QAA AEQLKT TRNAYIQKYL ERARSTLSGV NKKALNTIIL IIVVFVLCFT PYHVAIIQHM IKKLRFSNFL ECSQRHSFQI SLHF TVCLM NFNCCMDPFI YFFACKGYKR KVMRMLKRQV SVSISSAVKS APEENSREMT ETQMMIHSKS SNGKDYKDDD DK UniProtKB: G-protein coupled receptor 183, G-protein coupled receptor 183 |
-分子 #5: 8-[(2E)-3-(4-chlorophenyl)prop-2-enoyl]-3-[(3,4-dichlorophenyl)me...
| 分子 | 名称: 8-[(2E)-3-(4-chlorophenyl)prop-2-enoyl]-3-[(3,4-dichlorophenyl)methyl]-1-oxa-3,8-diazaspiro[4.5]decan-2-one タイプ: ligand / ID: 5 / コピー数: 1 / 式: KKF |
|---|---|
| 分子量 | 理論値: 479.783 Da |
| Chemical component information | 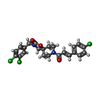 ChemComp-KKF: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DIFFRACTION / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)