+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | C2-symmetric single-particle cryo-EM map of T. vaginalis FDPF3 | |||||||||
 マップデータ マップデータ | C2-symmetric, sharpened, masked map of TvFDPF3 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | oxygen scavenging / fmn / nadh / flavodiiron protein / OXIDOREDUCTASE | |||||||||
| 生物種 |  Trichomonas vaginalis (ちつほねまくむし) Trichomonas vaginalis (ちつほねまくむし) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.8 Å | |||||||||
 データ登録者 データ登録者 | Bell TA / Chao LH | |||||||||
| 資金援助 | 2件
| |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2022 ジャーナル: J Biol Chem / 年: 2022タイトル: A natural fusion of flavodiiron, rubredoxin, and rubredoxin oxidoreductase domains is a self-sufficient water-forming oxidase of Trichomonas vaginalis. 著者: Evana N Abdulaziz / Tristan A Bell / Bazlur Rashid / Mina L Heacock / Tarik Begic / Owen S Skinner / Mohammad A Yaseen / Luke H Chao / Vamsi K Mootha / Antonio J Pierik / Valentin Cracan /   要旨: Microaerophilic pathogens such as Giardia lamblia, Entamoeba histolytica, and Trichomonas vaginalis have robust oxygen consumption systems to detoxify oxygen and maintain intracellular redox balance. ...Microaerophilic pathogens such as Giardia lamblia, Entamoeba histolytica, and Trichomonas vaginalis have robust oxygen consumption systems to detoxify oxygen and maintain intracellular redox balance. This oxygen consumption results from HO-forming NADH oxidase (NOX) activity of two distinct flavin-containing systems: HO-forming NOXes and multicomponent flavodiiron proteins (FDPs). Neither system is membrane bound, and both recycle NADH into oxidized NAD while simultaneously removing O from the local environment. However, little is known about the specific contributions of these systems in T. vaginalis. In this study, we use bioinformatics and biochemical analyses to show that T. vaginalis lacks a NOX-like enzyme and instead harbors three paralogous genes (FDPF1-3), each encoding a natural fusion product between the N-terminal FDP, central rubredoxin (Rb), and C-terminal NADH:Rb oxidoreductase domains. Unlike a "stand-alone" FDP that lacks Rb and oxidoreductase domains, this natural fusion protein with fully populated flavin redox centers directly accepts reducing equivalents of NADH to catalyze the four-electron reduction of oxygen to water within a single polypeptide with an extremely high turnover. Furthermore, using single-particle cryo-EM, we present structural insights into the spatial organization of the FDP core within this multidomain fusion protein. Together, these results contribute to our understanding of systems that allow protozoan parasites to maintain optimal redox balance and survive transient exposure to oxic conditions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25787.map.gz emd_25787.map.gz | 3.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25787-v30.xml emd-25787-v30.xml emd-25787.xml emd-25787.xml | 19.1 KB 19.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25787_fsc.xml emd_25787_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25787.png emd_25787.png | 83.3 KB | ||
| Filedesc metadata |  emd-25787.cif.gz emd-25787.cif.gz | 5 KB | ||
| その他 |  emd_25787_additional_1.map.gz emd_25787_additional_1.map.gz emd_25787_half_map_1.map.gz emd_25787_half_map_1.map.gz emd_25787_half_map_2.map.gz emd_25787_half_map_2.map.gz | 59.6 MB 48.5 MB 48.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25787 http://ftp.pdbj.org/pub/emdb/structures/EMD-25787 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25787 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25787 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25787_validation.pdf.gz emd_25787_validation.pdf.gz | 684.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25787_full_validation.pdf.gz emd_25787_full_validation.pdf.gz | 684.4 KB | 表示 | |
| XML形式データ |  emd_25787_validation.xml.gz emd_25787_validation.xml.gz | 16.1 KB | 表示 | |
| CIF形式データ |  emd_25787_validation.cif.gz emd_25787_validation.cif.gz | 21.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25787 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25787 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25787 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25787 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 電子顕微鏡画像生データ |  EMPIAR-10895 (タイトル: C2-symmetric single-particle cryo-EM map of T. vaginalis FDPF3 - unaligned multi-frame micrographs EMPIAR-10895 (タイトル: C2-symmetric single-particle cryo-EM map of T. vaginalis FDPF3 - unaligned multi-frame micrographsData size: 1.4 TB Data #1: Unaligned multi-frame micrographs of recombinantly expressed and purified T. vaginalis FDPF3 [picked particles - multiframe - unprocessed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25787.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25787.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | C2-symmetric, sharpened, masked map of TvFDPF3 | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.825 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: C2-symmetric, sharpened, unmasked map of TvFDPF3
| ファイル | emd_25787_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | C2-symmetric, sharpened, unmasked map of TvFDPF3 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
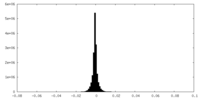
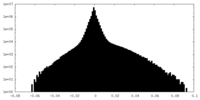
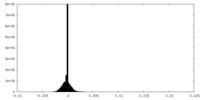
| 密度ヒストグラム |
-ハーフマップ: Half-map 1 from masked refinement of C2-symmetric map...
| ファイル | emd_25787_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 1 from masked refinement of C2-symmetric map prior to sharpening | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
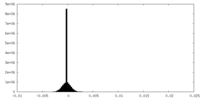
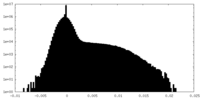
| 密度ヒストグラム |
-ハーフマップ: Half-map 2 from masked refinement of C2-symmetric map...
| ファイル | emd_25787_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 2 from masked refinement of C2-symmetric map prior to sharpening | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
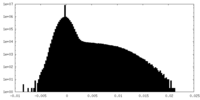
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Dimeric assembly of TvFDPF3 N-terminal FDP-like domains
| 全体 | 名称: Dimeric assembly of TvFDPF3 N-terminal FDP-like domains |
|---|---|
| 要素 |
|
-超分子 #1: Dimeric assembly of TvFDPF3 N-terminal FDP-like domains
| 超分子 | 名称: Dimeric assembly of TvFDPF3 N-terminal FDP-like domains タイプ: complex / ID: 1 / 親要素: 0 / 詳細: Resolved portion of full-lengh TvFDPF3 |
|---|---|
| 由来(天然) | 生物種:  Trichomonas vaginalis (ちつほねまくむし) / 株: C-1:NIH Trichomonas vaginalis (ちつほねまくむし) / 株: C-1:NIH |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 12 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 3.9 kPa 詳細: Grid was washed twice with chloroform and air dried prior to glow discharge and sample application. | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV / 詳細: Blotting condition: 5 s at +15 blotting force.. | |||||||||
| 詳細 | Monodisperse sample |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 7398 / 平均電子線量: 54.5 e/Å2 / 詳細: Images were collected as 51-frame movies. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.3 µm / 倍率(公称値): 105000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X