+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | BG505.MD39TS Env trimer in complex with Fab from antibody C05 | |||||||||
 マップデータ マップデータ | BG505.MD39TS Env trimer in complex with Fab from antibody C05 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | antibody / Env / envelope / Viral Protein-Immune System complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / identical protein binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  | |||||||||
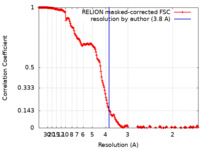
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Moore A / Du J | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Induction of tier-2 neutralizing antibodies in mice with a DNA-encoded HIV envelope native like trimer. 著者: Ziyang Xu / Susanne Walker / Megan C Wise / Neethu Chokkalingam / Mansi Purwar / Alan Moore / Edgar Tello-Ruiz / Yuanhan Wu / Sonali Majumdar / Kylie M Konrath / Abhijeet Kulkarni / Nicholas ...著者: Ziyang Xu / Susanne Walker / Megan C Wise / Neethu Chokkalingam / Mansi Purwar / Alan Moore / Edgar Tello-Ruiz / Yuanhan Wu / Sonali Majumdar / Kylie M Konrath / Abhijeet Kulkarni / Nicholas J Tursi / Faraz I Zaidi / Emma L Reuschel / Ishaan Patel / April Obeirne / Jianqiu Du / Katherine Schultheis / Lauren Gites / Trevor Smith / Janess Mendoza / Kate E Broderick / Laurent Humeau / Jesper Pallesen / David B Weiner / Daniel W Kulp /  要旨: HIV Envelope (Env) is the main vaccine target for induction of neutralizing antibodies. Stabilizing Env into native-like trimer (NLT) conformations is required for recombinant protein immunogens to ...HIV Envelope (Env) is the main vaccine target for induction of neutralizing antibodies. Stabilizing Env into native-like trimer (NLT) conformations is required for recombinant protein immunogens to induce autologous neutralizing antibodies(nAbs) against difficult to neutralize HIV strains (tier-2) in rabbits and non-human primates. Immunizations of mice with NLTs have generally failed to induce tier-2 nAbs. Here, we show that DNA-encoded NLTs fold properly in vivo and induce autologous tier-2 nAbs in mice. DNA-encoded NLTs also uniquely induce both CD4 + and CD8 + T-cell responses as compared to corresponding protein immunizations. Murine neutralizing antibodies are identified with an advanced sequencing technology. The structure of an Env-Ab (C05) complex, as determined by cryo-EM, identifies a previously undescribed neutralizing Env C3/V5 epitope. Beyond potential functional immunity gains, DNA vaccines permit in vivo folding of structured antigens and provide significant cost and speed advantages for enabling rapid evaluation of new HIV vaccines. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25376.map.gz emd_25376.map.gz | 479.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25376-v30.xml emd-25376-v30.xml emd-25376.xml emd-25376.xml | 22.3 KB 22.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25376_fsc.xml emd_25376_fsc.xml | 18.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25376.png emd_25376.png | 98.3 KB | ||
| Filedesc metadata |  emd-25376.cif.gz emd-25376.cif.gz | 6.6 KB | ||
| その他 |  emd_25376_additional_1.map.gz emd_25376_additional_1.map.gz emd_25376_half_map_1.map.gz emd_25376_half_map_1.map.gz emd_25376_half_map_2.map.gz emd_25376_half_map_2.map.gz | 410.6 MB 412.6 MB 410.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25376 http://ftp.pdbj.org/pub/emdb/structures/EMD-25376 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25376 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25376 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25376_validation.pdf.gz emd_25376_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25376_full_validation.pdf.gz emd_25376_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_25376_validation.xml.gz emd_25376_validation.xml.gz | 25.6 KB | 表示 | |
| CIF形式データ |  emd_25376_validation.cif.gz emd_25376_validation.cif.gz | 33.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25376 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25376 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25376 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25376 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7sq1MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25376.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25376.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | BG505.MD39TS Env trimer in complex with Fab from antibody C05 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.84 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unsharpened map
| ファイル | emd_25376_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map 1
| ファイル | emd_25376_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map 2
| ファイル | emd_25376_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : BG505.MD39TS Env trimer in complex with Fab from antibody C05
| 全体 | 名称: BG505.MD39TS Env trimer in complex with Fab from antibody C05 |
|---|---|
| 要素 |
|
-超分子 #1: BG505.MD39TS Env trimer in complex with Fab from antibody C05
| 超分子 | 名称: BG505.MD39TS Env trimer in complex with Fab from antibody C05 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 分子量 | 理論値: 410 KDa |
-分子 #1: Transmembrane protein gp41
| 分子 | 名称: Transmembrane protein gp41 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 17.134324 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AVGIGAVSLG FLGAAGSTMG AASMTLTVQA RNLLSGIVQQ QSNLLRAPEP QQHLLKDTHW GIKQLQARVL AVEHYLRDQQ LLGIWGCSG KLICCTNVPW NSSWSNRNLS EIWDNMTWLQ WDKEISNYTQ IIYGLLEESQ NQQEKNEQDL LALD UniProtKB: Envelope glycoprotein gp160 |
-分子 #2: Envelope glycoprotein gp160
| 分子 | 名称: Envelope glycoprotein gp160 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 52.917855 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: ENLWVTVYYG VPVWKDAETT LFCASDAKAY ETEKHNVWAT HACVPTDPNP QEIHLENVTE EFNMWKNNMV EQMHEDIISL WDQSLKPCV KLTPLCVTLQ CTNVTNNITD DMRGELKNCS FNMTTELRDK KQKVYSLFYR LDVVQINENQ GNRSNNSNKE Y RLINCNTS ...文字列: ENLWVTVYYG VPVWKDAETT LFCASDAKAY ETEKHNVWAT HACVPTDPNP QEIHLENVTE EFNMWKNNMV EQMHEDIISL WDQSLKPCV KLTPLCVTLQ CTNVTNNITD DMRGELKNCS FNMTTELRDK KQKVYSLFYR LDVVQINENQ GNRSNNSNKE Y RLINCNTS AITQACPKVS FEPIPIHYCA PAGFAILKCK DKKFNGTGPC PSVSTVQCTH GIKPVVSTQL LLNGSLAEEE VI IRSENIT NNAKNILVQL NTPVQINCTR PNNNTVKSIR IGPGQAFYYT GDIIGDIRQA HCNVSKATWN ETLGKVVKQL RKH FGNNTI IRFAQSSGGD LEVTTHSFNC GGEFFYCNTS GLFNSTWISN TSVQGSNSTG SNDSITLPCR IKQIINMWQR IGQA MYAPP IQGVIRCVSN ITGLILTRDG GSTNSTTETF RPGGGDMRDN WRSELYKYKV VKIEPLGVAP TRCKRRV UniProtKB: Envelope glycoprotein gp160 |
-分子 #3: C05 Fab Light chain
| 分子 | 名称: C05 Fab Light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 12.031465 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIVLTQSPAS LAVSPGQRAT ISCRPSESVD NYGISFMNWF QQKPGQPPKL LIYAASNRGS GVPARFTGSG SGTDFSLNIH PMEEDDIAM YFCQQSKEVP YTFGGGTKLE IK |
-分子 #4: C05 Fab heavy chain
| 分子 | 名称: C05 Fab heavy chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.727245 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EVKLEESGGG LVQAGGSMKL SCVASGFSLS NYWMNWVRQS PEKGLEWVAE IRLKPQNYAT HYAESVKGRF SISRDDSRST VYLQMNNLR AEDTGIYYCT RPGYYGHYAM DYWGQGTSVT VSS |
-分子 #9: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 9 / コピー数: 41 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.05 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 6000 pixel / デジタル化 - サイズ - 縦: 4000 pixel / 撮影したグリッド数: 1 / 実像数: 5485 / 平均露光時間: 2.59 sec. / 平均電子線量: 60.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER |
|---|---|
| 得られたモデル |  PDB-7sq1: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)