+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-25210 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of human SARS-CoV-2 spike glycoprotein trimer bound by neutralizing antibody C1C-A3 Fab (variable region) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | COVID-19 / SARS-CoV-2 / neutralizing antibody / neutralization escape / IMMUNE SYSTEM / VIRAL PROTEIN-IMMUNE SYSTEM complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated disruption of host tissue / Maturation of spike protein / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / viral translation / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion ...symbiont-mediated disruption of host tissue / Maturation of spike protein / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / viral translation / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell / membrane fusion / Attachment and Entry / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / host cell surface receptor binding / symbiont-mediated suppression of host innate immune response / receptor ligand activity / endocytosis involved in viral entry into host cell / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / symbiont entry into host cell / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
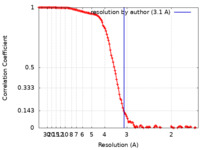
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Pan J / Abraham J | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2022 ジャーナル: Science / 年: 2022タイトル: Structural basis for continued antibody evasion by the SARS-CoV-2 receptor binding domain. 著者: Katherine G Nabel / Sarah A Clark / Sundaresh Shankar / Junhua Pan / Lars E Clark / Pan Yang / Adrian Coscia / Lindsay G A McKay / Haley H Varnum / Vesna Brusic / Nicole V Tolan / Guohai Zhou ...著者: Katherine G Nabel / Sarah A Clark / Sundaresh Shankar / Junhua Pan / Lars E Clark / Pan Yang / Adrian Coscia / Lindsay G A McKay / Haley H Varnum / Vesna Brusic / Nicole V Tolan / Guohai Zhou / Michaël Desjardins / Sarah E Turbett / Sanjat Kanjilal / Amy C Sherman / Anand Dighe / Regina C LaRocque / Edward T Ryan / Casey Tylek / Joel F Cohen-Solal / Anhdao T Darcy / Davide Tavella / Anca Clabbers / Yao Fan / Anthony Griffiths / Ivan R Correia / Jane Seagal / Lindsey R Baden / Richelle C Charles / Jonathan Abraham /   要旨: Many studies have examined the impact of severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) variants on neutralizing antibody activity after they have become dominant strains. Here, we ...Many studies have examined the impact of severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) variants on neutralizing antibody activity after they have become dominant strains. Here, we evaluate the consequences of further viral evolution. We demonstrate mechanisms through which the SARS-CoV-2 receptor binding domain (RBD) can tolerate large numbers of simultaneous antibody escape mutations and show that pseudotypes containing up to seven mutations, as opposed to the one to three found in previously studied variants of concern, are more resistant to neutralization by therapeutic antibodies and serum from vaccine recipients. We identify an antibody that binds the RBD core to neutralize pseudotypes for all tested variants but show that the RBD can acquire an N-linked glycan to escape neutralization. Our findings portend continued emergence of escape variants as SARS-CoV-2 adapts to humans. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25210.map.gz emd_25210.map.gz | 167.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25210-v30.xml emd-25210-v30.xml emd-25210.xml emd-25210.xml | 23.2 KB 23.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25210_fsc.xml emd_25210_fsc.xml | 12.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25210.png emd_25210.png | 144 KB | ||
| Filedesc metadata |  emd-25210.cif.gz emd-25210.cif.gz | 8.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25210 http://ftp.pdbj.org/pub/emdb/structures/EMD-25210 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25210 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25210 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25210_validation.pdf.gz emd_25210_validation.pdf.gz | 570.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25210_full_validation.pdf.gz emd_25210_full_validation.pdf.gz | 570 KB | 表示 | |
| XML形式データ |  emd_25210_validation.xml.gz emd_25210_validation.xml.gz | 13.1 KB | 表示 | |
| CIF形式データ |  emd_25210_validation.cif.gz emd_25210_validation.cif.gz | 17.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25210 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25210 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25210 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25210 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25210.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25210.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.825 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV-2 spike protein trimer in complex with human patient ant...
| 全体 | 名称: SARS-CoV-2 spike protein trimer in complex with human patient antibody C1C-A3 Fab |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 spike protein trimer in complex with human patient ant...
| 超分子 | 名称: SARS-CoV-2 spike protein trimer in complex with human patient antibody C1C-A3 Fab タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 詳細: Concentration of the SARS-CoV-2 spike protein (trimeric) and human patient antibody C1C-A3 Fab is 0.6 and 0.3 mg/mL in the final complex sample in buffer consisting of 150 mM NaCl and 25 mM Tris-HCl, pH 7.5 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 50 KDa |
-超分子 #2: SARS-CoV-2 spike protein
| 超分子 | 名称: SARS-CoV-2 spike protein / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 詳細: the SARS-Cov-2 spike protein is a trimeric protein of a calculated amino acid molecular weight of 418176 Da, with 23 N-linked glycans. Total molecular weight is 556176 Da assuming each glycan is 2000 Da. |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: C1C-A3 Fab (variable region)
| 超分子 | 名称: C1C-A3 Fab (variable region) / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 詳細: Fab of C1C-A3 antibody recombinantly expressed using sequence sequenced from single B cells from human patient peripheral blood sample |
|---|
-分子 #1: Spike glycoprotein
| 分子 | 名称: Spike glycoprotein / タイプ: protein_or_peptide / ID: 1 詳細: amino acids 1-15 are the endogenous signal peptide, 16-1208 are SARS-CoV-2 spike protein residues 16-1208, 1209-1210, 1237-1239, and 1255-1258 are linkers, 1211-1236 are T4 fibritinn foldon, ...詳細: amino acids 1-15 are the endogenous signal peptide, 16-1208 are SARS-CoV-2 spike protein residues 16-1208, 1209-1210, 1237-1239, and 1255-1258 are linkers, 1211-1236 are T4 fibritinn foldon, 1240-1254 are the avi-tag for biotinylation, 1259-1265 are the TEV protease cleavage site, 1266-1273 are the 8xHis tag; mutations include R682G, R683S, R685S, F817P, A892P, A899P, A942P, K986P, V987P コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 141.192203 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDG VYFASTEKSN IIRGWIFGTT LDSKTQSLLI VNNATNVVIK VCEFQFCNDP FLGVYYHKNN KSWMESEFRV Y SSANNCTF ...文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDG VYFASTEKSN IIRGWIFGTT LDSKTQSLLI VNNATNVVIK VCEFQFCNDP FLGVYYHKNN KSWMESEFRV Y SSANNCTF EYVSQPFLMD LEGKQGNFKN LREFVFKNID GYFKIYSKHT PINLVRDLPQ GFSALEPLVD LPIGINITRF QT LLALHRS YLTPGDSSSG WTAGAAAYYV GYLQPRTFLL KYNENGTITD AVDCALDPLS ETKCTLKSFT VEKGIYQTSN FRV QPTESI VRFPNITNLC PFGEVFNATR FASVYAWNRK RISNCVADYS VLYNSASFST FKCYGVSPTK LNDLCFTNVY ADSF VIRGD EVRQIAPGQT GKIADYNYKL PDDFTGCVIA WNSNNLDSKV GGNYNYLYRL FRKSNLKPFE RDISTEIYQA GSTPC NGVE GFNCYFPLQS YGFQPTNGVG YQPYRVVVLS FELLHAPATV CGPKKSTNLV KNKCVNFNFN GLTGTGVLTE SNKKFL PFQ QFGRDIADTT DAVRDPQTLE ILDITPCSFG GVSVITPGTN TSNQVAVLYQ DVNCTEVPVA IHADQLTPTW RVYSTGS NV FQTRAGCLIG AEHVNNSYEC DIPIGAGICA SYQTQTNSPG SASSVASQSI IAYTMSLGAE NSVAYSNNSI AIPTNFTI S VTTEILPVSM TKTSVDCTMY ICGDSTECSN LLLQYGSFCT QLNRALTGIA VEQDKNTQEV FAQVKQIYKT PPIKDFGGF NFSQILPDPS KPSKRSPIED LLFNKVTLAD AGFIKQYGDC LGDIAARDLI CAQKFNGLTV LPPLLTDEMI AQYTSALLAG TITSGWTFG AGPALQIPFP MQMAYRFNGI GVTQNVLYEN QKLIANQFNS AIGKIQDSLS STPSALGKLQ DVVNQNAQAL N TLVKQLSS NFGAISSVLN DILSRLDPPE AEVQIDRLIT GRLQSLQTYV TQQLIRAAEI RASANLAATK MSECVLGQSK RV DFCGKGY HLMSFPQSAP HGVVFLHVTY VPAQEKNFTT APAICHDGKA HFPREGVFVS NGTHWFVTQR NFYEPQIITT DNT FVSGNC DVVIGIVNNT VYDPLQPELD SFKEELDKYF KNHTSPDVDL GDISGINASV VNIQKEIDRL NEVAKNLNES LIDL QELGK YEQSGYIPEA PRDGQAYVRK DGEWVLLSTF LSAGGLNDIF EAQKIEWHEG ASGENLYFQG HHHHHHHH UniProtKB: Spike glycoprotein |
-分子 #2: neutralizing antibody C1C-A3 heavy chain variable region
| 分子 | 名称: neutralizing antibody C1C-A3 heavy chain variable region タイプ: protein_or_peptide / ID: 2 詳細: amino acids 1-20 are the human tPA signal peptide, 21-22 are a linker, 23-250 are the C1C-A3 Fab heavy chain residues 1-213 (note insertions in CDR loops; numbering should be according to the PDB file). コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 27.022607 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKRGLCCVLL LCGAVFVSPS ASQVQLVESG GGVVQPGRSL RLSCAASGFT FSSYGMHWVR QAPGKGLEWV AVIWYDGTNT YYADSVKGR FTISRDNSKN TLYLQMNSLR AEDTAVYYCA RDLAYRDYVW RYFDLWGRGT LVTVSGASTK GPSVFPLAPS S KSTSGGTA ...文字列: MKRGLCCVLL LCGAVFVSPS ASQVQLVESG GGVVQPGRSL RLSCAASGFT FSSYGMHWVR QAPGKGLEWV AVIWYDGTNT YYADSVKGR FTISRDNSKN TLYLQMNSLR AEDTAVYYCA RDLAYRDYVW RYFDLWGRGT LVTVSGASTK GPSVFPLAPS S KSTSGGTA ALGCLVKDYF PEPVTVSWNS GALTSGVHTF PAVLQSSGLY SLSSVVTVPS SSLGTQTYIC NVNHKPSNTK VD KRVEPKS CDR |
-分子 #3: neutralizing antibody C1C-A3 light chain variable region
| 分子 | 名称: neutralizing antibody C1C-A3 light chain variable region タイプ: protein_or_peptide / ID: 3 詳細: amino acids 1-20 are the human tPA signal peptide, 21-22 are a linker, 23-238 are the C1C-A3 Fab light chain residues 1-212 (note insertions in CDR loops; numbering should be according to the PDB file). コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.896107 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKRGLCCVLL LCGAVFVSPS ASEIVLTQSP ATLSLSPGER ATLSCRASQS VSTYLAWYQQ KFGQAPRLLI YDASNRATGI PARFSGSGS GTDFTLTISS LEPEDFAVYY CQCRSNWPPG ITFGQGTRLE IKRTVAAPSV FIFPPSDEQL KSGTASVVCL L NNFYPREA ...文字列: MKRGLCCVLL LCGAVFVSPS ASEIVLTQSP ATLSLSPGER ATLSCRASQS VSTYLAWYQQ KFGQAPRLLI YDASNRATGI PARFSGSGS GTDFTLTISS LEPEDFAVYY CQCRSNWPPG ITFGQGTRLE IKRTVAAPSV FIFPPSDEQL KSGTASVVCL L NNFYPREA KVQWKVDNAL QSGNSQESVT EQDSKDSTYS LSSTLTLSKA DYEKHKVYAC EVTHQGLSSP VTKSFNRGEC |
-分子 #5: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 5 / コピー数: 45 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.9 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 2 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa / 詳細: glow discharge done at 20 mA in Pelco easiGlow | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV / 詳細: blotting time 4-6 seconds. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 77.0 K / 最高: 77.0 K |
| 詳細 | Cs-corrected, energy filtered |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 4583 / 平均露光時間: 0.038 sec. / 平均電子線量: 56.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 60606 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER / 温度因子: 77.28 / 当てはまり具合の基準: correlation coefficient |
|---|---|
| 得られたモデル |  PDB-7sn3: |
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)