+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2505 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Septin-Gic1 complex in a single-tilt-axis subtomogram. | |||||||||
 マップデータ マップデータ | Cryo-Tomogram of Septin/Gic1 Complex. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | cell division / bud neck filaments / cytokinesis / cytoskeleton / electron tomography / septin / gic | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報septin ring organization / Cdc42 protein signal transduction / incipient cellular bud site / cellular bud tip / regulation of exit from mitosis / cellular bud neck / mating projection tip / establishment of cell polarity / small GTPase binding / regulation of cell shape ...septin ring organization / Cdc42 protein signal transduction / incipient cellular bud site / cellular bud tip / regulation of exit from mitosis / cellular bud neck / mating projection tip / establishment of cell polarity / small GTPase binding / regulation of cell shape / cell cortex / cytoskeleton / nucleus / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 電子線トモグラフィー法 / クライオ電子顕微鏡法 / 解像度: 60.0 Å | |||||||||
 データ登録者 データ登録者 | Sadian Y / Gatsogiannis C / Patasi C / Hofnagel O / Goody RS / Farkasovsky M / Raunser S | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2013 ジャーナル: Elife / 年: 2013タイトル: The role of Cdc42 and Gic1 in the regulation of septin filament formation and dissociation. 著者: Yashar Sadian / Christos Gatsogiannis / Csilla Patasi / Oliver Hofnagel / Roger S Goody / Marian Farkasovský / Stefan Raunser /  要旨: Septins are guanine nucleotide-binding proteins that polymerize into filamentous and higher-order structures. Cdc42 and its effector Gic1 are involved in septin recruitment, ring formation and ...Septins are guanine nucleotide-binding proteins that polymerize into filamentous and higher-order structures. Cdc42 and its effector Gic1 are involved in septin recruitment, ring formation and dissociation. The regulatory mechanisms behind these processes are not well understood. Here, we have used electron microscopy and cryo electron tomography to elucidate the structural basis of the Gic1-septin and Gic1-Cdc42-septin interaction. We show that Gic1 acts as a scaffolding protein for septin filaments forming long and flexible filament cables. Cdc42 in its GTP-form binds to Gic1, which ultimately leads to the dissociation of Gic1 from the filament cables. Surprisingly, Cdc42-GDP is not inactive, but in the absence of Gic1 directly interacts with septin filaments resulting in their disassembly. We suggest that this unanticipated dual function of Cdc42 is crucial for the cell cycle. Based on our results we propose a novel regulatory mechanism for septin filament formation and dissociation. DOI: http://dx.doi.org/10.7554/eLife.01085.001. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2505.map.gz emd_2505.map.gz | 6.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2505-v30.xml emd-2505-v30.xml emd-2505.xml emd-2505.xml | 9.9 KB 9.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2505.jpg emd_2505.jpg | 1.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2505 http://ftp.pdbj.org/pub/emdb/structures/EMD-2505 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2505 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2505 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  2504C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2505.map.gz / 形式: CCP4 / 大きさ: 6.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2505.map.gz / 形式: CCP4 / 大きさ: 6.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
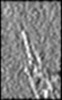
| 注釈 | Cryo-Tomogram of Septin/Gic1 Complex. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 7.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
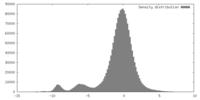
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Septin-Gic1 complex
| 全体 | 名称: Septin-Gic1 complex |
|---|---|
| 要素 |
|
-超分子 #1000: Septin-Gic1 complex
| 超分子 | 名称: Septin-Gic1 complex / タイプ: sample / ID: 1000 / Number unique components: 2 |
|---|
-分子 #1: Septin
| 分子 | 名称: Septin / タイプ: protein_or_peptide / ID: 1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | InterPro: Septin |
-分子 #2: Gic1
| 分子 | 名称: Gic1 / タイプ: protein_or_peptide / ID: 2 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | UniProtKB: GTPase-interacting component 1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 詳細: 100 mM NaCl, 20 mM mM Tris-HCl, 1 mM DTT |
|---|---|
| グリッド | 詳細: 400 Mesh 2/1 C-flat holey carbon grids |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 100 K / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | OTHER |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: in-column Omega filte エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 15.0 eV |
| 日付 | 2011年10月28日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F816 (8k x 8k) 実像数: 63 / 平均電子線量: 65 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 85470 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 4.1 mm / 倍率(公称値): 40000 |
| 試料ステージ | 試料ホルダーモデル: JEOL 3200FSC CRYOHOLDER / Tilt series - Axis1 - Min angle: -62 ° / Tilt series - Axis1 - Max angle: 62 ° / Tilt series - Axis1 - Angle increment: 2 ° |
- 画像解析
画像解析
| 詳細 | Standard eTOMO procedures for single axis tilt tomograms |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 60.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称:  Imod / 使用した粒子像数: 63 Imod / 使用した粒子像数: 63 |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)