+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of type I-D Cascade bound to a dsDNA target | |||||||||
 マップデータ マップデータ | Sharpened Map of CRISPR-Cas type I-D complex bound to a dsDNA target. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR / Complex / Ribonucleoprotein complex / type I-D / type ID / type I / cyanobacteria / synechocystis / RNA binding protein / DNA binding protein / DNA BINDING PROTEIN-DNA-RNA complex | |||||||||
| 機能・相同性 | CRISPR-associated protein Csc1 / CRISPR associated protein Csc3 / CRISPR-associated protein Csc2 / Csc2 Crispr / Slr7013 protein / Slr7012 protein / CRISPR-associated protein Csc3 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
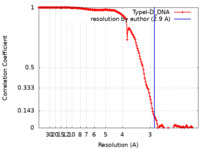
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Schwartz EA / Taylor DW | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structural rearrangements allow nucleic acid discrimination by type I-D Cascade. 著者: Evan A Schwartz / Tess M McBride / Jack P K Bravo / Daniel Wrapp / Peter C Fineran / Robert D Fagerlund / David W Taylor /   要旨: CRISPR-Cas systems are adaptive immune systems that protect prokaryotes from foreign nucleic acids, such as bacteriophages. Two of the most prevalent CRISPR-Cas systems include type I and type III. ...CRISPR-Cas systems are adaptive immune systems that protect prokaryotes from foreign nucleic acids, such as bacteriophages. Two of the most prevalent CRISPR-Cas systems include type I and type III. Interestingly, the type I-D interference proteins contain characteristic features of both type I and type III systems. Here, we present the structures of type I-D Cascade bound to both a double-stranded (ds)DNA and a single-stranded (ss)RNA target at 2.9 and 3.1 Å, respectively. We show that type I-D Cascade is capable of specifically binding ssRNA and reveal how PAM recognition of dsDNA targets initiates long-range structural rearrangements that likely primes Cas10d for Cas3' binding and subsequent non-target strand DNA cleavage. These structures allow us to model how binding of the anti-CRISPR protein AcrID1 likely blocks target dsDNA binding via competitive inhibition of the DNA substrate engagement with the Cas10d active site. This work elucidates the unique mechanisms used by type I-D Cascade for discrimination of single-stranded and double stranded targets. Thus, our data supports a model for the hybrid nature of this complex with features of type III and type I systems. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24974.map.gz emd_24974.map.gz | 290.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24974-v30.xml emd-24974-v30.xml emd-24974.xml emd-24974.xml | 19.9 KB 19.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_24974_fsc.xml emd_24974_fsc.xml | 15.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24974.png emd_24974.png | 52.4 KB | ||
| Filedesc metadata |  emd-24974.cif.gz emd-24974.cif.gz | 7.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24974 http://ftp.pdbj.org/pub/emdb/structures/EMD-24974 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24974 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24974 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24974_validation.pdf.gz emd_24974_validation.pdf.gz | 365.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24974_full_validation.pdf.gz emd_24974_full_validation.pdf.gz | 365.2 KB | 表示 | |
| XML形式データ |  emd_24974_validation.xml.gz emd_24974_validation.xml.gz | 14.5 KB | 表示 | |
| CIF形式データ |  emd_24974_validation.cif.gz emd_24974_validation.cif.gz | 19.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24974 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24974 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24974 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24974 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7sbaMC  7sbbC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24974.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24974.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened Map of CRISPR-Cas type I-D complex bound to a dsDNA target. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.045 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Type I-D Cascade bound to a dsDNA target
| 全体 | 名称: Type I-D Cascade bound to a dsDNA target |
|---|---|
| 要素 |
|
-超分子 #1: Type I-D Cascade bound to a dsDNA target
| 超分子 | 名称: Type I-D Cascade bound to a dsDNA target / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 理論値: 480 KDa |
-分子 #1: Cas7d
| 分子 | 名称: Cas7d / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 36.527109 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLDSLKSQFQ PSFPRLASGH YVHFLMLRHS QSFPVFQTDG VLNTTRTQAG LLEKTDQLSR LVMFKRKQTT PERLAGRELL RNLGLTSAD KSAKNLCEYN GEGSCKQCPD CILYGFAIGD SGSERSKVYS DSAFSLGAYE QSHRSFTFNA PFEGGTMSEA G VMRSAINE ...文字列: MLDSLKSQFQ PSFPRLASGH YVHFLMLRHS QSFPVFQTDG VLNTTRTQAG LLEKTDQLSR LVMFKRKQTT PERLAGRELL RNLGLTSAD KSAKNLCEYN GEGSCKQCPD CILYGFAIGD SGSERSKVYS DSAFSLGAYE QSHRSFTFNA PFEGGTMSEA G VMRSAINE LDHILPEVTF PTVESLRDAT YEGFIYVLGN LLRTKRYGAQ ESRTGTMKNH LVGIVFADGE IFSNLHLTQA LY DQMGGEL NKPISELCET AATVAQDLLN KEPVRKSELI FGAHLDTLLQ EVNDIYQNDA ELTKLLGSLY QQTQDYATEF GAL SGGKKK AKS UniProtKB: Slr7012 protein |
-分子 #2: Cas5d
| 分子 | 名称: Cas5d / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 28.942748 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTKIYRCKLT LHDNVFFASR EMGILYETEK YFHNWALSYA FFKGTIIPHP YGLVGQNAQT PAYLDRDREQ NLLHLNDSGI YVFPAQPIH WSYQINTFKA AQSAYYGRSV QFGGKGATKN YPINYGRAKE LAVGSEFLTY IVSQKELDLP VWIRLGKWSS K IRVEVEAI ...文字列: MTKIYRCKLT LHDNVFFASR EMGILYETEK YFHNWALSYA FFKGTIIPHP YGLVGQNAQT PAYLDRDREQ NLLHLNDSGI YVFPAQPIH WSYQINTFKA AQSAYYGRSV QFGGKGATKN YPINYGRAKE LAVGSEFLTY IVSQKELDLP VWIRLGKWSS K IRVEVEAI APDQIKTASG VYVCNHPLNP LDCPANQQIL LYNRVVMPPS SLFSQSQLQG DYWQIDRNTF LPQGFHYGAT TA IAQDSPQ LSLLDTN UniProtKB: Slr7013 protein |
-分子 #3: Cas10d
| 分子 | 名称: Cas10d / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 112.067508 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTTLLQTLLI RTLSEQKDYI LLEYFQTILP ALEEHFGNTS GLGGSFISHQ KHFGTQGYDT EKAKKMAQGF AKKGDQTLAA HILNALLTT WNVMQELEFP LNDIERRLLC LGITLHDYDK HCHAQDMAAP EPDNIQEIIN ICLELGKRLN FDEFWADWRD Y IAEISYLA ...文字列: MTTLLQTLLI RTLSEQKDYI LLEYFQTILP ALEEHFGNTS GLGGSFISHQ KHFGTQGYDT EKAKKMAQGF AKKGDQTLAA HILNALLTT WNVMQELEFP LNDIERRLLC LGITLHDYDK HCHAQDMAAP EPDNIQEIIN ICLELGKRLN FDEFWADWRD Y IAEISYLA QNTHGKQHTN LISSNWSNAG YPFTIKERKL DHPLRHLLTF GDVAVHLSSP HDLVSSTMGD RLRDLLNRLG IE KRFVYHH LRDTTGILSN AIHNVILRTV QKLDWKPLLF FAQGVIYFAP QDTEIPERNE IKQIVWQGIS QELGKKMSAG DVG FKRDGK GLKVSPQTSE LLAAADIVRI LPQVISVKVN NAKSPATPKR LEKLELGDAE REKLYEVADL RCDRLAELLG LVQK EIFLL PEPFIEWVLK DLELTSVIMP EETQVQSGGV NYGWYRVAAH YVANHATWDL EEFQEFLQGF GDRLATWAEE EGYFA EHQS PTRQIFEDYL DRYLEIQGWE SDHQAFIQEL ENYVNAKTKK SKQPICSLSS GEFPSEDQMD SVVLFKPQQY SNKNPL GGG QIKRGISKIW SLEMLLRQAF WSVPSGKFED QQPIFIYLYP AYVYAPQVVE AIRELVYGIA SVNLWDVRKH WVNNKMD LT SLKSLPWLNE EVEAGTNAQL KYTKEDLPFL ATVYTTTREK TDTDAWVKPA FLALLLPYLL GVKAIATRSM VPLYRSDQ D FRESIHLDGV AGFWSLLGIP TDLRVEDITP ALNKLLAIYT LHLAARSSPP KARWQDLPKT VQEVMTDVLN VFALAEQGL RREKRDRPYE SEVTEYWQFA ELFSQGNIVM TEKLKLTKRL VEEYRRFYQV ELSKKPSTHA ILLPLSKALE QILSVPDDWD EEELILQGS GQLQAALDRQ EVYTRPIIKD KSVAYETRQL QELEAIQIFM TTCVRDLFGE MCKGDRAILQ EQRNRIKSGA E FAYRLLAL EAQQNQN UniProtKB: CRISPR-associated protein Csc3 |
-分子 #4: Cas11d
| 分子 | 名称: Cas11d / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 17.024494 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTEKLKLTKR LVEEYRRFYQ VELSKKPSTH AILLPLSKAL EQILSVPDDW DEEELILQGS GQLQAALDRQ EVYTRPIIKD KSVAYETRQ LQELEAIQIF MTTCVRDLFG EMCKGDRAIL QEQRNRIKSG AEFAYRLLAL EAQQNQN UniProtKB: CRISPR-associated protein Csc3 |
-分子 #5: DNA target strand
| 分子 | 名称: DNA target strand / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 4.884222 KDa |
| 配列 | 文字列: (DA)(DC)(DA)(DA)(DC)(DA)(DA)(DT)(DC)(DA) (DA)(DC)(DG)(DT)(DG)(DA) |
-分子 #6: DNA non-target strand
| 分子 | 名称: DNA non-target strand / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 3.947581 KDa |
| 配列 | 文字列: (DC)(DA)(DC)(DG)(DT)(DT)(DG)(DA)(DT)(DT) (DT)(DT)(DT) |
-分子 #7: crRNA
| 分子 | 名称: crRNA / タイプ: rna / ID: 7 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.699081 KDa |
| 配列 | 文字列: ACUGAAACGA UUGUUGUGCC CCUGGCGGUC GCUUUCAAUG CCU GENBANK: GENBANK: CP073020.1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.125 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| グリッド | モデル: C-flat / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 30 sec. | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: Blot force 0, blot time 4.5s, no wait time between sample application and blotting.. | |||||||||||||||
| 詳細 | Monodisperse sample of elongated particles. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 平均露光時間: 3.0 sec. / 平均電子線量: 41.2 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Model was built de novo using coot then flexibly refined using ISOLDE. |
|---|---|
| 得られたモデル |  PDB-7sba: |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)