+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | DrmAB:ADP | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DISARM / Helicase / IMMUNE SYSTEM | |||||||||
| 生物種 |  Serratia (バクテリア) Serratia (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Bravo JPK / Taylor DW / Brouns SJJ / Aparicio-Maldonado C | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structural basis for broad anti-phage immunity by DISARM. 著者: Jack P K Bravo / Cristian Aparicio-Maldonado / Franklin L Nobrega / Stan J J Brouns / David W Taylor /    要旨: In the evolutionary arms race against phage, bacteria have assembled a diverse arsenal of antiviral immune strategies. While the recently discovered DISARM (Defense Island System Associated with ...In the evolutionary arms race against phage, bacteria have assembled a diverse arsenal of antiviral immune strategies. While the recently discovered DISARM (Defense Island System Associated with Restriction-Modification) systems can provide protection against a wide range of phage, the molecular mechanisms that underpin broad antiviral targeting but avoiding autoimmunity remain enigmatic. Here, we report cryo-EM structures of the core DISARM complex, DrmAB, both alone and in complex with an unmethylated phage DNA mimetic. These structures reveal that DrmAB core complex is autoinhibited by a trigger loop (TL) within DrmA and binding to DNA substrates containing a 5' overhang dislodges the TL, initiating a long-range structural rearrangement for DrmAB activation. Together with structure-guided in vivo studies, our work provides insights into the mechanism of phage DNA recognition and specific activation of this widespread antiviral defense system. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24938.map.gz emd_24938.map.gz | 8.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24938-v30.xml emd-24938-v30.xml emd-24938.xml emd-24938.xml | 13.7 KB 13.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_24938.png emd_24938.png | 77.9 KB | ||
| Filedesc metadata |  emd-24938.cif.gz emd-24938.cif.gz | 6.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24938 http://ftp.pdbj.org/pub/emdb/structures/EMD-24938 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24938 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24938 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24938_validation.pdf.gz emd_24938_validation.pdf.gz | 349.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24938_full_validation.pdf.gz emd_24938_full_validation.pdf.gz | 348.9 KB | 表示 | |
| XML形式データ |  emd_24938_validation.xml.gz emd_24938_validation.xml.gz | 6.4 KB | 表示 | |
| CIF形式データ |  emd_24938_validation.cif.gz emd_24938_validation.cif.gz | 7.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24938 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24938 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24938 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24938 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24938.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24938.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : DrmAB:ADP
| 全体 | 名称: DrmAB:ADP |
|---|---|
| 要素 |
|
-超分子 #1: DrmAB:ADP
| 超分子 | 名称: DrmAB:ADP / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Serratia (バクテリア) Serratia (バクテリア) |
-分子 #1: DrmA
| 分子 | 名称: DrmA / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Serratia (バクテリア) Serratia (バクテリア) |
| 分子量 | 理論値: 147.980766 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTDNNKSSKT PNAWIAIDHN FSRAQVLTYY TLQLKGEHSL HQAISDNDWI LVLDTTGNIT RVGRILRIRS DLETTTIFFD RMLQVKSVV SIGITPFKFP PNDRAGRIQW TDFIETLPKE LHITIADIPK IEDQTYIREL LQLAVMDDLL GPAGGPNELI V DMGVRDRY ...文字列: MTDNNKSSKT PNAWIAIDHN FSRAQVLTYY TLQLKGEHSL HQAISDNDWI LVLDTTGNIT RVGRILRIRS DLETTTIFFD RMLQVKSVV SIGITPFKFP PNDRAGRIQW TDFIETLPKE LHITIADIPK IEDQTYIREL LQLAVMDDLL GPAGGPNELI V DMGVRDRY LVGKLAPREA AERGQEFPID AEDIEDEEPD LIVKAKTAKV NSPSVLGSGE TDTAEEIDAA SNQSLVPSSL GM TFCVDGD VDRVEIEARW GRYERVPNDE HQFFKSNGQK AKVWKRIPCG GKIVLPLIEG SISHNAPDST SPEVRVQGSI RAK NDNGDR LITLFLVNAQ EEPDTNRDTA WVFQPELIVR AAKDAAKPAI FRRRPVLDAD GMDPEREALE MIYRDRVEFA VGHG VAVHA EIADDVTLAT EVRTTVMPQY EVQATETPGL ELSDRPAMRE MVSSGLLDMQ RLATLDIDPL VDALSVLTND YATWI DEQN LNVSSKAKGF DTQAQTAINR CQEIHTRLQE GINTLKSNEN ALAAFRFANQ AMATQRIRSL YALAMRRGED VTLDKF DVL KNRSWRPFQL AFLLLSIPSL ADPCHPDRVK PIEAYADLLW FPTGGGKTEA YLGVAAFTMA IRRMQGNLGG YDSSRGL TV IMRYTLRLLT LQQFQRATAL ICAMEVLRRE ALNKGDKSLG TEPFTIGLWV GNKVTPGTTE DSHNAIEKTR NPGSYNAG T ASPVQLTSCP WCGTEIVPGQ DVEVKKDKAG GRTFVYCGDK KGRCEFSKGK SSTQPHPGIP VLVVDEEIYH RPPTMMIAT VDKFAMMAWR GQVRTLFGRV EKECERHGLL WPGANCTGNH QAFKGQPSAK VKAIPPIRPP DLIIQDEFHL ISGPLGTMVG LYETAVDEL CSWTLNGKTV KPKIIASTAT VRKAKEQVNN VFMRQVSVFP PHGLDVEDNF FSVQRHIKDK FGRRYLGVCS P GSSRPAML IRVYTAFLTA AQELFDHFGE PADPYMTMVG YFNSLRELGG MKRLAEDDVQ TRSYRVQMSM VERPALAQRS VN NIRELTS RVSSQDIPKY LDNLEVKFKA EFDSSAGKYV TKWQEGDTRA IDVVLATNML SVGVDVNRLG LMAVNGQPKG TAE YIQATS RVGRSFPGLV CTVLTWARPR DLSHYETFEH YHATFYKHVE AQSVTPFSPR AMDRGLTGSL LSLMRLKNNE FSPN EGAGK LDMSNQSELA HAIEVLATRA GNVAEDNARK LLAENELKER ADEWAKEASK GGRILGYEKR GPDKDKTVAL IKSPG LQAW DNWTVPMSMR EVESGVRLIM DTKFIKDDHD WKPRPATKDE D |
-分子 #2: DrmB
| 分子 | 名称: DrmB / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Serratia (バクテリア) Serratia (バクテリア) |
| 分子量 | 理論値: 68.667039 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MIINNKTPVG EVRPSQLLWT YGPGALIDLP SLSVVTLGID RWERERCQPI QEARLLAAVR KVLGPQVENL RMPPFQKSEL VDPWSAEAN IGVPVRPFPR WMRCVKCGLL SPFDDGLLEI KEDRFRAERT RFVHKGCTGS KGNLPAKDAD AVPARFLLAC R DGHLDDFP ...文字列: MIINNKTPVG EVRPSQLLWT YGPGALIDLP SLSVVTLGID RWERERCQPI QEARLLAAVR KVLGPQVENL RMPPFQKSEL VDPWSAEAN IGVPVRPFPR WMRCVKCGLL SPFDDGLLEI KEDRFRAERT RFVHKGCTGS KGNLPAKDAD AVPARFLLAC R DGHLDDFP WHYFVHGGNS TCKGTLRFFE SGASLQTENL WVRCDSCEAS RSMAHAFGKA GKENLPACRG RHPHLDQFDI DC GEEPRAV LLGATNSWFP ITLSALAIPQ SKNPLSQLIQ DGWPLFEAIT AEVMVPIVVQ TLKLTGGLPG IDKYSVSDIW SAI EMHRSG GDSEFVGEAD IKGPEWEVLT EANPPTDYPH FMSKKIGTPA QFIPYISRVL LLERLREVNA LLGFTRVEAP EGSG EINER PQMASLARNK PEWVPANQVH GEGIFIQFNE KTLVAWESLD AVKQVDEMLR GGHTGWRNSR NLDPNEDYPG IRYAM LHTL SHLLIRELAL ECGYNAASIR ERIYADTSNG SPQAGILIYT AAADSDGTLG GLVDLGKPEN LGRLLVQALN RSKICS SDP LCSEHNPEKD RSLHAAACHA CTLVAETSCE QGNRYLDRSL LIPTLERIHA AFFKGF |
-分子 #3: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 80.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



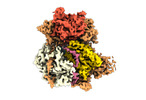



 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)




















