+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24848 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Native-form oocyte/egg Alpha-2-Macroglobulin (A2Moo) tetramer | |||||||||
 マップデータ マップデータ | Native-form oocyte/egg Alpha-2-Macroglobulin (A2Moo) tetramer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Xenopus egg extract / Protease inhibitor / PROTEIN BINDING | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 | ||||||||||
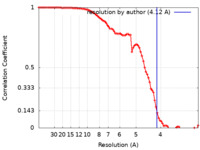
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.12 Å | |||||||||
 データ登録者 データ登録者 | Arimura Y / Funabiki H | |||||||||
| 資金援助 |  日本, 2件 日本, 2件
| |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2022 ジャーナル: J Mol Biol / 年: 2022タイトル: Structural Mechanics of the Alpha-2-Macroglobulin Transformation. 著者: Yasuhiro Arimura / Hironori Funabiki /  要旨: Alpha-2-Macroglobulin (A2M) is the critical pan-protease inhibitor of the innate immune system. When proteases cleave the A2M bait region, global structural transformation of the A2M tetramer is ...Alpha-2-Macroglobulin (A2M) is the critical pan-protease inhibitor of the innate immune system. When proteases cleave the A2M bait region, global structural transformation of the A2M tetramer is triggered to entrap the protease. The structural basis behind the cleavage-induced transformation and the protease entrapment remains unclear. Here, we report cryo-EM structures of native- and intermediate-forms of the Xenopus laevis egg A2M homolog (A2Moo or ovomacroglobulin) tetramer at 3.7-4.1 Å and 6.4 Å resolution, respectively. In the native A2Moo tetramer, two pairs of dimers arrange into a cross-like configuration with four 60 Å-wide bait-exposing grooves. Each bait in the native form threads into an aperture formed by three macroglobulin domains (MG2, MG3, MG6). The bait is released from the narrowed aperture in the induced protomer of the intermediate form. We propose that the intact bait region works as a "latch-lock" to block futile A2M transformation until its protease-mediated cleavage. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24848.map.gz emd_24848.map.gz | 56.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24848-v30.xml emd-24848-v30.xml emd-24848.xml emd-24848.xml | 21 KB 21 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_24848_fsc.xml emd_24848_fsc.xml | 8.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24848.png emd_24848.png | 154.8 KB | ||
| Filedesc metadata |  emd-24848.cif.gz emd-24848.cif.gz | 6.2 KB | ||
| その他 |  emd_24848_additional_1.map.gz emd_24848_additional_1.map.gz emd_24848_half_map_1.map.gz emd_24848_half_map_1.map.gz emd_24848_half_map_2.map.gz emd_24848_half_map_2.map.gz | 59.4 MB 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24848 http://ftp.pdbj.org/pub/emdb/structures/EMD-24848 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24848 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24848 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24848_validation.pdf.gz emd_24848_validation.pdf.gz | 900.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24848_full_validation.pdf.gz emd_24848_full_validation.pdf.gz | 899.9 KB | 表示 | |
| XML形式データ |  emd_24848_validation.xml.gz emd_24848_validation.xml.gz | 16.5 KB | 表示 | |
| CIF形式データ |  emd_24848_validation.cif.gz emd_24848_validation.cif.gz | 21.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24848 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24848 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24848 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24848 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24848.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24848.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Native-form oocyte/egg Alpha-2-Macroglobulin (A2Moo) tetramer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.47 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
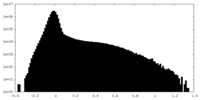
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Native-form oocyte/egg Alpha-2-Macroglobulin (A2Moo) tetramer
| ファイル | emd_24848_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Native-form oocyte/egg Alpha-2-Macroglobulin (A2Moo) tetramer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
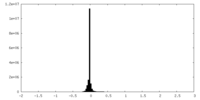
| 密度ヒストグラム |
-ハーフマップ: Native-form oocyte/egg Alpha-2-Macroglobulin (A2Moo) tetramer
| ファイル | emd_24848_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Native-form oocyte/egg Alpha-2-Macroglobulin (A2Moo) tetramer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
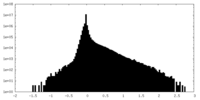
| 密度ヒストグラム |
-ハーフマップ: Native-form oocyte/egg Alpha-2-Macroglobulin (A2Moo) tetramer
| ファイル | emd_24848_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Native-form oocyte/egg Alpha-2-Macroglobulin (A2Moo) tetramer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
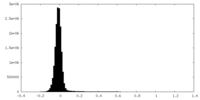
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : native-form oocyte/egg Alpha-2-Macroglobulin (A2Moo) tetramer
| 全体 | 名称: native-form oocyte/egg Alpha-2-Macroglobulin (A2Moo) tetramer |
|---|---|
| 要素 |
|
-超分子 #1: native-form oocyte/egg Alpha-2-Macroglobulin (A2Moo) tetramer
| 超分子 | 名称: native-form oocyte/egg Alpha-2-Macroglobulin (A2Moo) tetramer タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種: |
-分子 #1: Alpha 2-Macroglobulin
| 分子 | 名称: Alpha 2-Macroglobulin / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 159.480812 KDa |
| 配列 | 文字列: MSPNRFLLCV CILGLIAGGA AKVQYALTIP ALLKSGETQR ACVNLIGYHQ PLALSVVLEH QRVNISIFSE KVQPPHYFKC NKFMVPTVI TNAPDFVTLS VSGGGEDIKD RKAVVIAPLN TICLIQMDKP VYKPGCKVRF RLISLNTMLL PISEKYTAVY L EDPSGSRI ...文字列: MSPNRFLLCV CILGLIAGGA AKVQYALTIP ALLKSGETQR ACVNLIGYHQ PLALSVVLEH QRVNISIFSE KVQPPHYFKC NKFMVPTVI TNAPDFVTLS VSGGGEDIKD RKAVVIAPLN TICLIQMDKP VYKPGCKVRF RLISLNTMLL PISEKYTAVY L EDPSGSRI AQWQNQESVG GVVQLEFPLI SDAAPGSYTI TAEGESCESA RQGFTVDEYI LPRFSVIVDP PNTISILDDI LT LNVSAIY TYGQPVPGSV TIKCCREASS YYGRKGNCFK GNRGICTNIT GELGPDGAFY GVVSLLPFQM GQSGFQMSLG VAL TVTEEG TGIQVTHQFF IMITSQLATL IFDYDALKEF YKRGIPYLVK VILTDANDNP MANEQVEVEL AGKTIGAVLT DKEG RAEYA IDTSSFVQEN FTVVVSYENP HQCYYTEWEG PDFPTAQHFV MRFYSETGSF LDIQGSSVEL NCGQVHNISV RYILS LDGM GEGATTATFY YLAMSRAKIV QHGQRDVHLN QSKSGLFNIG LNVTSDLAPG AELIVYCILD LELIADTISL DIEKCF QNQ VSLSFSDDLG PTASNVSLNL SAAPGSLCGV KVIDSSLLLI NPYESLSASG VYYSIPYLSL FGYNYGGFNL EEPEPPC ED PNTVIFCKGR YYLPVSSSTE GDTYQNLRRV GLVLGTSSKI RKPVVCGMEA KFSVPRKSSG ESDFGSSLSN GHVETLRK N FSETFLWRLV SVDSEGQNTI TETVPDTITK WQGSMFCVSE KEGFGITKYS ANFTSFLPFF VELSLPYSLT REEILVMKA FVSNYLEECI KIIVTLQPSA DFEVIPQDVK QDQCICSGGR SSYSWNIIAS SLGRISFIVS AETTHIGASC DGPSDQSQST RKDTVIQTI LVQPEGIRKE ETSSNLVCVE DSNVEMPINL TLPENIVQGS ASAFVTFVGD VLGLPLSNLQ NLLQMPYGCG E QNLARMAP IPYVLEYLNN TNQLTDELLQ TAVQFLNEGY YRQLRYKLPS GAYDAFWSSP SDGSSWLSAY TFKTFEKAKK YI YVDGKIQ QQTLLYLQTS QKLDNGCFKA EGNLFMRQCG QERDLCFTAY LAIALLESNY SSGMTLLDDA LGCLEAAMSS AST LYFKSY TVYVFTLVQN WEIRNTLLNE LKSKVVSERG TLHWEREDKL GQEGIPLYYP NYSPAEVEIT AYMLLSIAKG SDPT HDDLT YMAQISVWLI QQQNSYGGFR STQDTVVALQ ALAFYAQLLF KSNAHHNVFL RSEYGDVGQL NLSEHNRLVV QRLQL PEVS GNYSISINGT GCCLVQSTIR YNIPVPKENS AFYVAADSVS KNCLNGVAYT ITITVSVSYR GLRNETNMVI IDIQML SGY QADYPSLRQL ENSQQVSKTE EQDNHVFLYL NAVPLKTIQL SFKVLIGSRV LNVKSASVYV YDYYETGENG FASYSQP C UniProtKB: Uncharacterized protein LOC431886 isoform X1 |
-分子 #2: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 2 / コピー数: 68 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #0 - 検出モード: SUPER-RESOLUTION / #0 - 平均電子線量: 33.11 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #1 - 検出モード: SUPER-RESOLUTION / #1 - 平均電子線量: 38.34 e/Å2 / #2 - Image recording ID: 3 #2 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #2 - 検出モード: SUPER-RESOLUTION / #2 - 平均電子線量: 35.27 e/Å2 / #3 - Image recording ID: 4 #3 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #3 - 検出モード: SUPER-RESOLUTION / #3 - 平均電子線量: 34.2 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)