+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24424 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Thermus thermophilus reiterative transcription complex with 11nt oligo-G RNA | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Thermus thermophilus / transcription initiation / TRANSCRIPTION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報sigma factor activity / DNA-directed RNA polymerase complex / DNA-templated transcription initiation / ribonucleoside binding / : / : / : / : / : / : ...sigma factor activity / DNA-directed RNA polymerase complex / DNA-templated transcription initiation / ribonucleoside binding / : / : / : / : / : / : / DNA-directed RNA polymerase / protein dimerization activity / magnesium ion binding / DNA binding / zinc ion binding / metal ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   Thermus thermophilus HB8 (バクテリア) / synthetic construct (人工物) Thermus thermophilus HB8 (バクテリア) / synthetic construct (人工物) | |||||||||
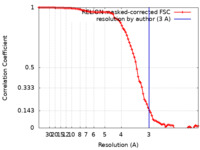
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Liu Y / Ebright RH | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2022 ジャーナル: Proc Natl Acad Sci U S A / 年: 2022タイトル: Structural and mechanistic basis of reiterative transcription initiation. 著者: Yu Liu / Libing Yu / Chirangini Pukhrambam / Jared T Winkelman / Emre Firlar / Jason T Kaelber / Yu Zhang / Bryce E Nickels / Richard H Ebright /   要旨: Reiterative transcription initiation, observed at promoters that contain homopolymeric sequences at the transcription start site, generates RNA products having 5' sequences noncomplementary to the ...Reiterative transcription initiation, observed at promoters that contain homopolymeric sequences at the transcription start site, generates RNA products having 5' sequences noncomplementary to the DNA template. Here, using crystallography and cryoelectron microscopy to define structures, protein-DNA photocrosslinking to map positions of RNAP leading and trailing edges relative to DNA, and single-molecule DNA nanomanipulation to assess RNA polymerase (RNAP)-dependent DNA unwinding, we show that RNA extension in reiterative transcription initiation 1) occurs without DNA scrunching; 2) involves a short, 2- to 3-bp, RNA-DNA hybrid; and 3) generates RNA that exits RNAP through the portal by which scrunched nontemplate-strand DNA exits RNAP in standard transcription initiation. The results establish that, whereas RNA extension in standard transcription initiation proceeds through a scrunching mechanism, RNA extension in reiterative transcription initiation proceeds through a slippage mechanism, with slipping of RNA relative to DNA within a short RNA-DNA hybrid, and with extrusion of RNA from RNAP through an alternative RNA exit. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24424.map.gz emd_24424.map.gz | 48.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24424-v30.xml emd-24424-v30.xml emd-24424.xml emd-24424.xml | 26.2 KB 26.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_24424_fsc.xml emd_24424_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24424.png emd_24424.png | 119.3 KB | ||
| Filedesc metadata |  emd-24424.cif.gz emd-24424.cif.gz | 8.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24424 http://ftp.pdbj.org/pub/emdb/structures/EMD-24424 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24424 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24424 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24424.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24424.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.038 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : reiterative transcription initiation complex with 11nt oligo-G RNA
+超分子 #1: reiterative transcription initiation complex with 11nt oligo-G RNA
+分子 #1: DNA-directed RNA polymerase subunit alpha
+分子 #2: DNA-directed RNA polymerase subunit beta
+分子 #3: DNA-directed RNA polymerase subunit beta'
+分子 #4: DNA-directed RNA polymerase subunit omega
+分子 #5: RNA polymerase sigma factor SigA
+分子 #6: DNA (31-MER) nontemplate strand
+分子 #7: DNA (31-MER) template strand
+分子 #8: RNA (5'-R(P*GP*GP*GP*GP*GP*GP*GP*GP*GP*GP*G)-3')
+分子 #9: MAGNESIUM ION
+分子 #10: ZINC ION
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.69 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.9 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.027 kPa | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-30 / 撮影したグリッド数: 1 / 実像数: 3424 / 平均露光時間: 0.2 sec. / 平均電子線量: 0.994 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): -2.8000000000000003 µm 最小 デフォーカス(公称値): -1.2 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-7rdq: |
-原子モデル構築 2
| 初期モデル | PDB ID: Chain - Chain ID: B / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 得られたモデル |  PDB-7rdq: |
-原子モデル構築 3
| 初期モデル | PDB ID: Chain - Chain ID: C / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 得られたモデル |  PDB-7rdq: |
-原子モデル構築 4
| 初期モデル | PDB ID: Chain - Chain ID: D / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 得られたモデル |  PDB-7rdq: |
-原子モデル構築 5
| 初期モデル | PDB ID: Chain - Chain ID: E / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 得られたモデル |  PDB-7rdq: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)