+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2423 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 3-dimensional structure of the toxin-delivery particle antifeeding prophage of Serratia entomophila | |||||||||
 マップデータ マップデータ | Reconstruction of the Afp tube-baseplate complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | tube-baseplate complex / tailocin / type VI secretion system / helical sheath / phage tail-like | |||||||||
| 生物種 |  Serratia entomophila (バクテリア) Serratia entomophila (バクテリア) | |||||||||
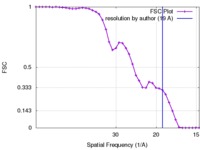
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 19.0 Å | |||||||||
 データ登録者 データ登録者 | Heymann JB / Bartho JD / Rybakova D / Venugopal HP / Winkler DC / Sen A / Hurst MRH / Mitra AK | |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2013 ジャーナル: J Biol Chem / 年: 2013タイトル: Three-dimensional structure of the toxin-delivery particle antifeeding prophage of Serratia entomophila. 著者: J Bernard Heymann / Joseph D Bartho / Daria Rybakova / Hari P Venugopal / Dennis C Winkler / Anindito Sen / Mark R H Hurst / Alok K Mitra /   要旨: The Serratia entomophila antifeeding prophage (Afp) is a bullet-shaped toxin-delivery apparatus similar to the R-pyocins of Pseudomonas aeruginosa. Morphologically it resembles the sheathed tail of ...The Serratia entomophila antifeeding prophage (Afp) is a bullet-shaped toxin-delivery apparatus similar to the R-pyocins of Pseudomonas aeruginosa. Morphologically it resembles the sheathed tail of bacteriophages such as T4, including a baseplate at one end. It also shares features with the type VI secretion systems. Cryo-electron micrographs of tilted Afp specimens (up to 60 degrees) were analyzed to determine the correct cyclic symmetry to overcome the limitation imposed by exclusively side views in nominally untilted specimens. An asymmetric reconstruction shows clear 6-fold cyclic symmetry contrary to a previous conclusion of 4-fold symmetry based on analysis of only the preferred side views (Sen, A., Rybakova, D., Hurst, M. R., and Mitra, A. K. (2010) J. Bacteriol. 192, 4522-4525). Electron tomography of negatively stained Afp revealed right-handed helical striations in many of the particles, establishing the correct hand. Higher quality micrographs of untilted specimens were processed to produce a reconstruction at 2.0-nm resolution with imposed 6-fold symmetry. The helical parameters of the sheath were determined to be 8.14 nm for the subunit rise along and 40.5° for the rotation angle around the helix. The sheath is similar to that of the T4 phage tail but with a different arrangement of the subdomain of the polymerizing sheath protein(s). The central tube is similar to the diameter and axial width of the Hcp1 hexamer of P. aeruginosa type VI secretion system. The tube extends through the baseplate into a needle resembling the "puncture device" of the T4 tail. The tube contains density that may be the toxin and/or a length-determining protein. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2423.map.gz emd_2423.map.gz | 15.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2423-v30.xml emd-2423-v30.xml emd-2423.xml emd-2423.xml | 9 KB 9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_2423_fsc.xml emd_2423_fsc.xml | 3.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_2423.png emd_2423.png | 133.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2423 http://ftp.pdbj.org/pub/emdb/structures/EMD-2423 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2423 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2423 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2423_validation.pdf.gz emd_2423_validation.pdf.gz | 214.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2423_full_validation.pdf.gz emd_2423_full_validation.pdf.gz | 213.8 KB | 表示 | |
| XML形式データ |  emd_2423_validation.xml.gz emd_2423_validation.xml.gz | 7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2423 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2423 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2423 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2423 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2423.map.gz / 形式: CCP4 / 大きさ: 16.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2423.map.gz / 形式: CCP4 / 大きさ: 16.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of the Afp tube-baseplate complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Tube-baseplate complex of the antifeeding prophage of Serratia en...
| 全体 | 名称: Tube-baseplate complex of the antifeeding prophage of Serratia entomophila |
|---|---|
| 要素 |
|
-超分子 #1000: Tube-baseplate complex of the antifeeding prophage of Serratia en...
| 超分子 | 名称: Tube-baseplate complex of the antifeeding prophage of Serratia entomophila タイプ: sample / ID: 1000 詳細: Tube-baseplate complex produced from 15 ORF products Number unique components: 1 |
|---|
-分子 #1: Antifeeding prophage tube-baseplate complex
| 分子 | 名称: Antifeeding prophage tube-baseplate complex / タイプ: protein_or_peptide / ID: 1 / Name.synonym: TBC 詳細: Product of the expression of 15 ORF's of the pADAP plasmid of Serratia entomophila コピー数: 1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Serratia entomophila (バクテリア) Serratia entomophila (バクテリア) |
| 組換発現 | 生物種:  組換プラスミド: pAF1-15 |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 詳細: 25 mM TBS, 130 mM NaCl |
|---|---|
| 染色 | タイプ: NEGATIVE / 詳細: 2% Uranyl acetate |
| グリッド | 詳細: Carbon film |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 12 |
|---|---|
| 日付 | 2013年2月11日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM デジタル化 - スキャナー: NIKON SUPER COOLSCAN 9000 デジタル化 - サンプリング間隔: 12.7 µm / 実像数: 75 / ビット/ピクセル: 16 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 倍率(補正後): 50364 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 2.3 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 52000 |
| 試料ステージ | 試料ホルダーモデル: OTHER |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera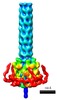



 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)