+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2409 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The architecture of yeast DNA polymerase 'zeta'. Electron microscopy reconstruction of the heterotetrameric complex Rev3-Rev7-Pol31-Pol32. | |||||||||
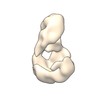
 マップデータ マップデータ | Map of Pol-zeta-d heterotetramer by negative stain | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Translesion DNA synthesis / DNA repair / DNA replication / DNA polymerase zeta / genome integrity / cancer | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報delta DNA polymerase complex / H3-H4 histone complex chaperone activity / DNA amplification / zeta DNA polymerase complex / RNA-templated DNA biosynthetic process / Processive synthesis on the lagging strand / Removal of the Flap Intermediate / Translesion synthesis by REV1 / Translesion synthesis by POLK / Translesion synthesis by POLI ...delta DNA polymerase complex / H3-H4 histone complex chaperone activity / DNA amplification / zeta DNA polymerase complex / RNA-templated DNA biosynthetic process / Processive synthesis on the lagging strand / Removal of the Flap Intermediate / Translesion synthesis by REV1 / Translesion synthesis by POLK / Translesion synthesis by POLI / DNA replication, removal of RNA primer / lagging strand elongation / double-strand break repair via break-induced replication / postreplication repair / error-free translesion synthesis / DNA metabolic process / DNA strand elongation involved in DNA replication / leading strand elongation / error-prone translesion synthesis / mismatch repair / double-strand break repair via homologous recombination / nucleotide-excision repair / base-excision repair / 4 iron, 4 sulfur cluster binding / DNA-directed DNA polymerase / DNA-directed DNA polymerase activity / DNA replication / nucleotide binding / chromatin / mitochondrion / DNA binding / zinc ion binding / nucleus / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 23.0 Å | |||||||||
 データ登録者 データ登録者 | Gomez-Llorente Y / Malik R / Jain R / Roy Choudhury J / Johnson RE / Prakash L / Prakash S / Ubarretxena-Belandia I / Aggarwal AK | |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2013 ジャーナル: Cell Rep / 年: 2013タイトル: The architecture of yeast DNA polymerase ζ. 著者: Yacob Gómez-Llorente / Radhika Malik / Rinku Jain / Jayati Roy Choudhury / Robert E Johnson / Louise Prakash / Satya Prakash / Iban Ubarretxena-Belandia / Aneel K Aggarwal /  要旨: DNA polymerase ζ (Polζ) is specialized for the extension step of translesion DNA synthesis (TLS). Despite its central role in maintaining genome integrity, little is known about its overall ...DNA polymerase ζ (Polζ) is specialized for the extension step of translesion DNA synthesis (TLS). Despite its central role in maintaining genome integrity, little is known about its overall architecture. Initially identified as a heterodimer of the catalytic subunit Rev3 and the accessory subunit Rev7, yeast Polζ has recently been shown to form a stable four-subunit enzyme (Polζ-d) upon the incorporation of Pol31 and Pol32, the accessory subunits of yeast Polδ. To understand the 3D architecture and assembly of Polζ and Polζ-d, we employed electron microscopy. We show here how the catalytic and accessory subunits of Polζ and Polζ-d are organized relative to each other. In particular, we show that Polζ-d has a bilobal architecture resembling the replicative polymerases and that Pol32 lies in proximity to Rev7. Collectively, our study provides views of Polζ and Polζ-d and a structural framework for understanding their roles in DNA damage bypass. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2409.map.gz emd_2409.map.gz | 1.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2409-v30.xml emd-2409-v30.xml emd-2409.xml emd-2409.xml | 14.9 KB 14.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2409.jpg emd_2409.jpg | 20.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2409 http://ftp.pdbj.org/pub/emdb/structures/EMD-2409 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2409 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2409 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2409_validation.pdf.gz emd_2409_validation.pdf.gz | 222.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2409_full_validation.pdf.gz emd_2409_full_validation.pdf.gz | 221.4 KB | 表示 | |
| XML形式データ |  emd_2409_validation.xml.gz emd_2409_validation.xml.gz | 5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2409 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2409 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2409 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2409 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2409.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2409.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map of Pol-zeta-d heterotetramer by negative stain | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.78 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Polymerase Zeta heterotetramer of Saccharomyces cerevisiae
| 全体 | 名称: Polymerase Zeta heterotetramer of Saccharomyces cerevisiae |
|---|---|
| 要素 |
|
-超分子 #1000: Polymerase Zeta heterotetramer of Saccharomyces cerevisiae
| 超分子 | 名称: Polymerase Zeta heterotetramer of Saccharomyces cerevisiae タイプ: sample / ID: 1000 / 詳細: Rev3:Rev7:Pol31:Pol32N 1:1:1:1 / 集合状態: Heterotetramer / Number unique components: 4 |
|---|---|
| 分子量 | 実験値: 300 KDa / 理論値: 298 KDa |
-分子 #1: DNA Polymerase 'Zeta' processivity subunit
| 分子 | 名称: DNA Polymerase 'Zeta' processivity subunit / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Rev7 / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 28 KDa / 理論値: 28 KDa |
| 組換発現 | 生物種:  |
| 配列 | UniProtKB: DNA polymerase zeta processivity subunit GO: error-free translesion synthesis, error-prone translesion synthesis InterPro: HORMA domain |
-分子 #2: DNA Polymerase 'Delta' small subunit
| 分子 | 名称: DNA Polymerase 'Delta' small subunit / タイプ: protein_or_peptide / ID: 2 / Name.synonym: Pol31 / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 55 KDa / 理論値: 55 KDa |
| 組換発現 | 生物種:  |
| 配列 | UniProtKB: DNA polymerase delta small subunit GO: DNA replication, removal of RNA primer, mismatch repair, base-excision repair InterPro: DNA polymerase alpha/delta/epsilon, subunit B, DNA polymerase delta/II small subunit family |
-分子 #3: DNA Polymerase 'Delta' subunit 3
| 分子 | 名称: DNA Polymerase 'Delta' subunit 3 / タイプ: protein_or_peptide / ID: 3 / Name.synonym: Pol32 詳細: We are using the N-terminal part of this protein. For further details, check the associated publication. コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 40 KDa / 理論値: 40 KDa |
| 組換発現 | 生物種:  |
| 配列 | UniProtKB: DNA polymerase delta subunit 3 GO: DNA replication, removal of RNA primer, mismatch repair, base-excision repair |
-分子 #4: DNA Polymerase 'Zeta' catalytic subunit
| 分子 | 名称: DNA Polymerase 'Zeta' catalytic subunit / タイプ: protein_or_peptide / ID: 4 / Name.synonym: Rev3 / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 173 KDa / 理論値: 173 KDa |
| 組換発現 | 生物種:  |
| 配列 | UniProtKB: DNA polymerase zeta catalytic subunit GO: error-free translesion synthesis, error-prone translesion synthesis, DNA replication InterPro: DNA-directed DNA polymerase, family B, DNA-directed DNA polymerase, family B, conserved site, DNA-directed DNA polymerase, family B, exonuclease domain, DNA-directed DNA polymerase, family ...InterPro: DNA-directed DNA polymerase, family B, DNA-directed DNA polymerase, family B, conserved site, DNA-directed DNA polymerase, family B, exonuclease domain, DNA-directed DNA polymerase, family B, multifunctional domain, DNA polymerase, palm domain superfamily, Ribonuclease H-like superfamily, C4-type zinc-finger of DNA polymerase delta |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 / 詳細: 25mM Tris pH 8.0, 250mM NaCl |
|---|---|
| 染色 | タイプ: NEGATIVE 詳細: 2 ul aliquots were adsorbed onto glow-discharged carbon coated copper grids, and negatively stained with 2% uranyl acetate. |
| グリッド | 詳細: Glow discharged carbon coated copper grids. |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2100F |
|---|---|
| 温度 | 最低: 80 K / 最高: 298 K / 平均: 293 K |
| 日付 | 2011年1月1日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GENERIC TVIPS / 平均電子線量: 10 e/Å2 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 63450 / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 40000 |
| 試料ステージ | 試料ホルダーモデル: JEOL |
- 画像解析
画像解析
| CTF補正 | 詳細: Each particle |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 23.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Xmipp, EMAN2, Spider / 使用した粒子像数: 14844 |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















