+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 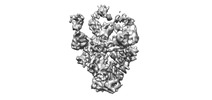 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
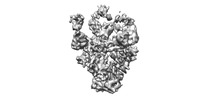
| タイトル | CryoEM map of monoclonal antibody Fab DH1025.1 bound to bound to CH505.M5 SOSIP trimer | |||||||||
 マップデータ マップデータ | CryoEM map of DH1025.1 bound to CH505.M5 SOSIP trimer | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.8 Å | |||||||||
 データ登録者 データ登録者 | Manne K / Acharya P | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: HIV-1 vaccine induction of protective tier 2 neutralizing antibodies in nonhuman primates is augmented by TLR7/8 agonism 著者: Tilahun K / Haynes BF | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24065.map.gz emd_24065.map.gz | 117.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24065-v30.xml emd-24065-v30.xml emd-24065.xml emd-24065.xml | 15.8 KB 15.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_24065.png emd_24065.png | 34.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24065 http://ftp.pdbj.org/pub/emdb/structures/EMD-24065 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24065 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24065 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24065_validation.pdf.gz emd_24065_validation.pdf.gz | 397.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24065_full_validation.pdf.gz emd_24065_full_validation.pdf.gz | 397 KB | 表示 | |
| XML形式データ |  emd_24065_validation.xml.gz emd_24065_validation.xml.gz | 6.8 KB | 表示 | |
| CIF形式データ |  emd_24065_validation.cif.gz emd_24065_validation.cif.gz | 7.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24065 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24065 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24065 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24065 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7mw9 M: このマップから作成された原子モデル |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24065.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24065.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM map of DH1025.1 bound to CH505.M5 SOSIP trimer | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : DH1025.1 Fab bound to CH505.M5 SOSIP trimer
| 全体 | 名称: DH1025.1 Fab bound to CH505.M5 SOSIP trimer |
|---|---|
| 要素 |
|
-超分子 #1: DH1025.1 Fab bound to CH505.M5 SOSIP trimer
| 超分子 | 名称: DH1025.1 Fab bound to CH505.M5 SOSIP trimer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: 293F Homo sapiens (ヒト) / 組換細胞: 293F |
-分子 #1: CH505.M5 gp120
| 分子 | 名称: CH505.M5 gp120 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 50.965219 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: ENLWVTVYYG VPVWKEAKTT LFCASDAKAY EKEVHNVWAT HACVPTDPNP QEMVLKNVTE NFNMWKNDMV DQMHEDVISL WDQSLKPCV KLTPLCVTLN CTNATASNSS IIEGMKNCSF NITTELRDKR EKKNALFYKL DIVQLDGNSS QYRLINCNTS V ITQACPKV ...文字列: ENLWVTVYYG VPVWKEAKTT LFCASDAKAY EKEVHNVWAT HACVPTDPNP QEMVLKNVTE NFNMWKNDMV DQMHEDVISL WDQSLKPCV KLTPLCVTLN CTNATASNSS IIEGMKNCSF NITTELRDKR EKKNALFYKL DIVQLDGNSS QYRLINCNTS V ITQACPKV SFDPIPIHYC APAGYAILKC NNKTFTGTGP CNNVSTVQCT HGIKPVVSTQ LLLNGSLAEG EIIIRSENIT KN VKTIIVH LNESVKIECT RPNNKTRTSI RIGPGQAFYA TGQVIGDIRE AYCNINESKW NETLQRVSKK LKEYFPHKNI TFQ PSSGGD LEITTHSFNC GGEFFYCNTS SLFNRTYMAN NSTRTITIHC RIKQIINMWQ EVGRAMYAPP IAGNITCISN ITGL LLTRD YGKNNTETFR PGGGNMKDNW RSELYKYKVV KIEPLGVAPT RCKRRVV |
-分子 #2: CH505.M5 gp41
| 分子 | 名称: CH505.M5 gp41 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 11.238608 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: VWGIKQLQAR VLAVERYLRD QQLLGIWGCS GKLICCTNVP WNSSWSNRNL SEIWDNMTWL QWDKEISNYT QIIYGLLEES QNQQEKNEQ DLLALD |
-分子 #3: DH1025.1 Fab Heavy Chain
| 分子 | 名称: DH1025.1 Fab Heavy Chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.238191 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVHLQESGPG LVKPSETLSL TCAVSGGSVS YYNWWSWIRQ PPGKGLEWIG YISGSSGNTY YSPSLKSRVT ISTDTSKNQF SLRLSSVTA ADTAVYYCAS VLQYLDWVLP DRVGEVHWFD VWGPGVLVSV SSASTKGPSV FPLAPSSRST SESTAALGCL V KDYFPEPV ...文字列: QVHLQESGPG LVKPSETLSL TCAVSGGSVS YYNWWSWIRQ PPGKGLEWIG YISGSSGNTY YSPSLKSRVT ISTDTSKNQF SLRLSSVTA ADTAVYYCAS VLQYLDWVLP DRVGEVHWFD VWGPGVLVSV SSASTKGPSV FPLAPSSRST SESTAALGCL V KDYFPEPV TVSWNSGSLT SGVHTFPAVL QSSGLYSLSS VVTVPSSSLG TQTYVCNVNH KPSNTKVDKR VEIKTCGG |
-分子 #4: DH1025.1 Fab Light Chain
| 分子 | 名称: DH1025.1 Fab Light Chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.12957 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIQMTQSPSS LSASVGDRVT ISCQASQGIS SRLAWYQQRP GKAPKLLIHT ASSLQSGVPS RFSGSGSGTE FTLTISSLQP EDFATYYCQ QHHSHPLTFG GGTKVEIKRA VAAPSVFIFP PSEDQVKSGT VSVVCLLNNF YPREASVKWK VDGVLKTGNS Q ESVTEQDS ...文字列: DIQMTQSPSS LSASVGDRVT ISCQASQGIS SRLAWYQQRP GKAPKLLIHT ASSLQSGVPS RFSGSGSGTE FTLTISSLQP EDFATYYCQ QHHSHPLTFG GGTKVEIKRA VAAPSVFIFP PSEDQVKSGT VSVVCLLNNF YPREASVKWK VDGVLKTGNS Q ESVTEQDS KDNTYSLSST LTLSNTDYQS HNVYACEVTH QGLSSPVTKS FNRGEC |
-分子 #7: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 7 / コピー数: 9 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 98 % / チャンバー内温度: 293 K / 装置: LEICA EM GP / 詳細: Blot for 2.5 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 93.15 K / 最高: 93.15 K |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 3056 / 平均電子線量: 62.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.75 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル | 
PDB-7mw9: |
 ムービー
ムービー コントローラー
コントローラー