+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23933 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
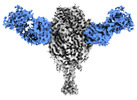
| タイトル | Cryo-EM structure of Prefusion-stabilized RSV F (DS-Cav1) in complex with Fab AM14 | |||||||||
 マップデータ マップデータ | Cryo-EM structure of RSV DS-Cav1 in complex with FabAM14. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | antigen / antibody / epitope / vaccine / structural vaccinology / IMMUNE SYSTEM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated induction of syncytium formation / host cell Golgi membrane / entry receptor-mediated virion attachment to host cell / fusion of virus membrane with host plasma membrane / viral envelope / symbiont entry into host cell / host cell plasma membrane / virion membrane / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Respiratory syncytial virus A2 (ウイルス) / Respiratory syncytial virus A2 (ウイルス) /  Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Abeyrathne PD / Malito E | |||||||||
 引用 引用 |  ジャーナル: MAbs / 年: 2021 ジャーナル: MAbs / 年: 2021タイトル: Improved epitope resolution of the prefusion trimer-specific antibody AM14 bound to the RSV F glycoprotein. 著者: Wayne Harshbarger / Priyanka D Abeyrathne / Sai Tian / Ying Huang / Sumana Chandramouli / Matthew James Bottomley / Enrico Malito /  要旨: Respiratory syncytial virus (RSV) is the most common cause of acute lower respiratory tract infections resulting in medical intervention and hospitalizations during infancy and early childhood, and ...Respiratory syncytial virus (RSV) is the most common cause of acute lower respiratory tract infections resulting in medical intervention and hospitalizations during infancy and early childhood, and vaccination against RSV remains a public health priority. The RSV F glycoprotein is a major target of neutralizing antibodies, and the prefusion stabilized form of F (DS-Cav1) is under investigation as a vaccine antigen. AM14 is a human monoclonal antibody with the exclusive capacity of binding an epitope on prefusion F (PreF), which spans two F protomers. The quality of recognizing a trimer-specific epitope makes AM14 valuable for probing PreF-based immunogen conformation and functionality during vaccine production. Currently, only a low-resolution (5.5 Å) X-ray structure is available of the PreF-AM14 complex, revealing few reliable details of the interface. Here, we perform complementary structural studies using X-ray crystallography and cryo-electron microscopy (cryo-EM) to provide improved resolution structures at 3.6 Å and 3.4 Å resolutions, respectively. Both X-ray and cryo-EM structures provide clear side-chain densities, which allow for accurate mapping of the AM14 epitope on DS-Cav1. The structures help rationalize the molecular basis for AM14 loss of binding to RSV F monoclonal antibody-resistant mutants and reveal flexibility for the side chain of a key antigenic residue on PreF. This work provides the basis for a comprehensive understanding of RSV F trimer specificity with implications in vaccine design and quality assessment of PreF-based immunogens. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23933.map.gz emd_23933.map.gz | 228.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23933-v30.xml emd-23933-v30.xml emd-23933.xml emd-23933.xml | 16.7 KB 16.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23933.png emd_23933.png | 93.3 KB | ||
| Filedesc metadata |  emd-23933.cif.gz emd-23933.cif.gz | 6.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23933 http://ftp.pdbj.org/pub/emdb/structures/EMD-23933 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23933 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23933 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23933_validation.pdf.gz emd_23933_validation.pdf.gz | 590.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23933_full_validation.pdf.gz emd_23933_full_validation.pdf.gz | 589.6 KB | 表示 | |
| XML形式データ |  emd_23933_validation.xml.gz emd_23933_validation.xml.gz | 7.3 KB | 表示 | |
| CIF形式データ |  emd_23933_validation.cif.gz emd_23933_validation.cif.gz | 8.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23933 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23933 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23933 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23933 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23933.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23933.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of RSV DS-Cav1 in complex with FabAM14. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Homo sapiens respiratory syncytial virus A
| 全体 | 名称: Homo sapiens respiratory syncytial virus A |
|---|---|
| 要素 |
|
-超分子 #1: Homo sapiens respiratory syncytial virus A
| 超分子 | 名称: Homo sapiens respiratory syncytial virus A / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 分子量 | 理論値: 285 KDa |
-超分子 #2: Fusion glycoprotein F1 and F2 fused with Fibritin trimerization d...
| 超分子 | 名称: Fusion glycoprotein F1 and F2 fused with Fibritin trimerization domain. タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Respiratory syncytial virus A2 (ウイルス) Respiratory syncytial virus A2 (ウイルス) |
-超分子 #3: Fab AM14 heavy and light chain
| 超分子 | 名称: Fab AM14 heavy and light chain / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Fusion glycoprotein F0,Envelope glycoprotein
| 分子 | 名称: Fusion glycoprotein F0,Envelope glycoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 54.806496 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QNITEEFYQS TCSAVSKGYL SALRTGWYTS VITIELSNIK ENKCNGTDAK VKLIKQELDK YKNAVTELQL LMQSTPATNN RAFLGFLLG VGSAIASGVA VCKVLHLEGE VNKIKSALLS TNKAVVSLSN GVSVLTFKVL DLKNYIDKQL LPILNKQSCS I SNIETVIE ...文字列: QNITEEFYQS TCSAVSKGYL SALRTGWYTS VITIELSNIK ENKCNGTDAK VKLIKQELDK YKNAVTELQL LMQSTPATNN RAFLGFLLG VGSAIASGVA VCKVLHLEGE VNKIKSALLS TNKAVVSLSN GVSVLTFKVL DLKNYIDKQL LPILNKQSCS I SNIETVIE FQQKNNRLLE ITREFSVNAG VTTPVSTYML TNSELLSLIN DMPITNDQKK LMSNNVQIVR QQSYSIMCII KE EVLAYVV QLPLYGVIDT PCWKLHTSPL CTTNTKEGSN ICLTRTDRGW YCDNAGSVSF FPQAETCKVQ SNRVFCDTMN SLT LPSEVN LCNVDIFNPK YDCKIMTSKT DVSSSVITSL GAIVSCYGKT KCTASNKNRG IIKTFSNGCD YVSNKGVDTV SVGN TLYYV NKQEGKSLYV KGEPIINFYD PLVFPSDEFD ASISQVNEKI NQSLAFIRKS DELLSAIGGY IPEAPRDGQA YVRKD GEWV LLSTFLGGLV PR UniProtKB: Fusion glycoprotein F0, Envelope glycoprotein |
-分子 #2: AM14 Fab Heavy Chain
| 分子 | 名称: AM14 Fab Heavy Chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 26.380352 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: CVQLVESGGG VVQPGRSLRL SCAASGFSFS HYAMHWVRQA PGKGLEWVAV ISYDGENTYY ADSVKGRFSI SRDNSKNTVS LQMNSLRPE DTALYYCARD RIVDDYYYYG MDVWGQGATV TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA ...文字列: CVQLVESGGG VVQPGRSLRL SCAASGFSFS HYAMHWVRQA PGKGLEWVAV ISYDGENTYY ADSVKGRFSI SRDNSKNTVS LQMNSLRPE DTALYYCARD RIVDDYYYYG MDVWGQGATV TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA LTSGVHTFPA VLQSSGLYSL SSVVTVPSSS LGTQTYICNV NHKPSNTKVD KKVEPKSCDK GSENLYFQGS HH HHHH |
-分子 #3: AM14 Fab Light Chain
| 分子 | 名称: AM14 Fab Light Chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.870906 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: METPAELLFL LLLWLPDTTG DIQMTQSPSS LSASVGDRVT ITCQASQDIK KYLNWYHQKP GKVPELLMHD ASNLETGVPS RFSGRGSGT DFTLTISSLQ PEDIGTYYCQ QYDNLPPLTF GGGTKVEIKR TVAAPSVFIF PPSDEQLKSG TASVVCLLNN F YPREAKVQ ...文字列: METPAELLFL LLLWLPDTTG DIQMTQSPSS LSASVGDRVT ITCQASQDIK KYLNWYHQKP GKVPELLMHD ASNLETGVPS RFSGRGSGT DFTLTISSLQ PEDIGTYYCQ QYDNLPPLTF GGGTKVEIKR TVAAPSVFIF PPSDEQLKSG TASVVCLLNN F YPREAKVQ WKVDNALQSG NSQESVTEQD SKDSTYSLSS TLTLSKADYE KHKVYACEVT HQGLSSPVTK SFNRGEC |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 3 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV | |||||||||
| 詳細 | DS-Cav1-FabAM14. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 1 / 実像数: 1078 / 平均露光時間: 60.0 sec. / 平均電子線量: 26.7 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-7mpg: |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















