+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2352 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Bunyamwera virus glycoprotein spike determined by electron cryo-tomography and sub-volume averaging | |||||||||
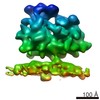
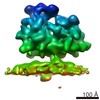
 マップデータ マップデータ | Sub-tomogram average of Bunyamwera virus glycoprotein spike | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Bunyavirus / viral glycoprotein / reconstruction / viral fusion / orthobunyavirus | |||||||||
| 生物種 |  Bunyamwera virus (ブニヤンベラウイルス) Bunyamwera virus (ブニヤンベラウイルス) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 30.0 Å | |||||||||
 データ登録者 データ登録者 | Bowden TA / Bitto D / McLees A / Yeromonahos C / Elliott RM / Huiskonen JT | |||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2013 ジャーナル: PLoS Pathog / 年: 2013タイトル: Orthobunyavirus ultrastructure and the curious tripodal glycoprotein spike. 著者: Thomas A Bowden / David Bitto / Angela McLees / Christelle Yeromonahos / Richard M Elliott / Juha T Huiskonen /  要旨: The genus Orthobunyavirus within the family Bunyaviridae constitutes an expanding group of emerging viruses, which threaten human and animal health. Despite the medical importance, little is known ...The genus Orthobunyavirus within the family Bunyaviridae constitutes an expanding group of emerging viruses, which threaten human and animal health. Despite the medical importance, little is known about orthobunyavirus structure, a prerequisite for understanding virus assembly and entry. Here, using electron cryo-tomography, we report the ultrastructure of Bunyamwera virus, the prototypic member of this genus. Whilst Bunyamwera virions are pleomorphic in shape, they display a locally ordered lattice of glycoprotein spikes. Each spike protrudes 18 nm from the viral membrane and becomes disordered upon introduction to an acidic environment. Using sub-tomogram averaging, we derived a three-dimensional model of the trimeric pre-fusion glycoprotein spike to 3-nm resolution. The glycoprotein spike consists mainly of the putative class-II fusion glycoprotein and exhibits a unique tripod-like arrangement. Protein-protein contacts between neighbouring spikes occur at membrane-proximal regions and intra-spike contacts at membrane-distal regions. This trimeric assembly deviates from previously observed fusion glycoprotein arrangements, suggesting a greater than anticipated repertoire of viral fusion glycoprotein oligomerization. Our study provides evidence of a pH-dependent conformational change that occurs during orthobunyaviral entry into host cells and a blueprint for the structure of this group of emerging pathogens. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2352.map.gz emd_2352.map.gz | 3.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2352-v30.xml emd-2352-v30.xml emd-2352.xml emd-2352.xml | 9.2 KB 9.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-2352.png EMD-2352.png | 129.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2352 http://ftp.pdbj.org/pub/emdb/structures/EMD-2352 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2352 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2352 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2352_validation.pdf.gz emd_2352_validation.pdf.gz | 211.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2352_full_validation.pdf.gz emd_2352_full_validation.pdf.gz | 211 KB | 表示 | |
| XML形式データ |  emd_2352_validation.xml.gz emd_2352_validation.xml.gz | 5.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2352 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2352 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2352 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2352 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2352.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2352.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sub-tomogram average of Bunyamwera virus glycoprotein spike | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5.4 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Purified Bunyamwera virus particles
| 全体 | 名称: Purified Bunyamwera virus particles |
|---|---|
| 要素 |
|
-超分子 #1000: Purified Bunyamwera virus particles
| 超分子 | 名称: Purified Bunyamwera virus particles / タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|
-分子 #1: Bunyamwera virus glycoprotein Gn-Gc membrane complex
| 分子 | 名称: Bunyamwera virus glycoprotein Gn-Gc membrane complex タイプ: protein_or_peptide / ID: 1 / Name.synonym: Bunyamwera viral glycoprotein spike 詳細: One central trimeric spike surrounded by six neighbouring spikes 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  Bunyamwera virus (ブニヤンベラウイルス) Bunyamwera virus (ブニヤンベラウイルス) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: 10 mM Tris pH 7.4, 100 mM NaCl |
|---|---|
| グリッド | 詳細: C-flat CF-2/1/2C |
| 凍結 | 凍結剤: ETHANE-PROPANE MIXTURE / チャンバー内温度: 103 K / 装置: GATAN CRYOPLUNGE 3 / 手法: Blot both sides for 2 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 温度 | 最低: 100 K / 最高: 120 K / 平均: 100 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF2002 エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 詳細 | Low dose tomography |
| 日付 | 2012年3月28日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GENERIC CCD / 平均電子線量: 100 e/Å2 詳細: Thirteen single-axis tilt series were collected over an angular range of -60 to 60 at 3 degree increments. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 111111 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 4.5 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダー: Liquid nitrogen cooled / 試料ホルダーモデル: OTHER / Tilt series - Axis1 - Min angle: -60 ° / Tilt series - Axis1 - Max angle: 60 ° |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | 2,000 subtomograms were selected using template matching in Jsubtomo |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C3 (3回回転対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 30.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Jsubtomo 詳細: Final map was calculated by averaging the two half maps. A low pass filter to 30 A was applied. 使用したサブトモグラム数: 2000 |
| CTF補正 | 詳細: Low pass filter was applied after the first zero of the CTF |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















