+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23251 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of a ferrichrome importer FhuCDB from E. coli | |||||||||
 マップデータ マップデータ | ferrichrome importer FhuCDB | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ABC importer / siderophore / cryo-EM / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ABC-type ferric hydroxamate transporter / ABC-type ferric hydroxamate transporter activity / ferric iron transmembrane transporter activity / ferric-hydroxamate import into cell / iron ion import across plasma membrane / siderophore-iron import into cell / siderophore uptake transmembrane transporter activity / transmembrane transporter activity / ATP-binding cassette (ABC) transporter complex, substrate-binding subunit-containing / ATP-binding cassette (ABC) transporter complex ...ABC-type ferric hydroxamate transporter / ABC-type ferric hydroxamate transporter activity / ferric iron transmembrane transporter activity / ferric-hydroxamate import into cell / iron ion import across plasma membrane / siderophore-iron import into cell / siderophore uptake transmembrane transporter activity / transmembrane transporter activity / ATP-binding cassette (ABC) transporter complex, substrate-binding subunit-containing / ATP-binding cassette (ABC) transporter complex / outer membrane-bounded periplasmic space / ATP hydrolysis activity / ATP binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Hu W / Zheng H | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2021 ジャーナル: Commun Biol / 年: 2021タイトル: Cryo-EM reveals unique structural features of the FhuCDB Escherichia coli ferrichrome importer. 著者: Wenxin Hu / Hongjin Zheng /  要旨: As one of the most elegant biological processes developed in bacteria, the siderophore-mediated iron uptake demands the action of specific ATP-binding cassette (ABC) importers. Although extensive ...As one of the most elegant biological processes developed in bacteria, the siderophore-mediated iron uptake demands the action of specific ATP-binding cassette (ABC) importers. Although extensive studies have been done on various ABC importers, the molecular basis of these iron-chelated-siderophore importers are still not fully understood. Here, we report the structure of a ferrichrome importer FhuCDB from Escherichia coli at 3.4 Å resolution determined by cryo electron microscopy. The structure revealed a monomeric membrane subunit of FhuB with a substrate translocation pathway in the middle. In the pathway, there were unique arrangements of residues, especially layers of methionines. Important residues found in the structure were interrogated by mutagenesis and functional studies. Surprisingly, the importer's ATPase activity was decreased upon FhuD binding, which deviated from the current understanding about bacterial ABC importers. In summary, to the best of our knowledge, these studies not only reveal a new structural twist in the type II ABC importer subfamily, but also provide biological insights in the transport of iron-chelated siderophores. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23251.map.gz emd_23251.map.gz | 117.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23251-v30.xml emd-23251-v30.xml emd-23251.xml emd-23251.xml | 12.2 KB 12.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
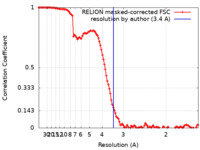
| FSC (解像度算出) |  emd_23251_fsc.xml emd_23251_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23251.png emd_23251.png | 200.2 KB | ||
| Filedesc metadata |  emd-23251.cif.gz emd-23251.cif.gz | 5.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23251 http://ftp.pdbj.org/pub/emdb/structures/EMD-23251 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23251 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23251 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23251_validation.pdf.gz emd_23251_validation.pdf.gz | 581.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23251_full_validation.pdf.gz emd_23251_full_validation.pdf.gz | 581.5 KB | 表示 | |
| XML形式データ |  emd_23251_validation.xml.gz emd_23251_validation.xml.gz | 12.2 KB | 表示 | |
| CIF形式データ |  emd_23251_validation.cif.gz emd_23251_validation.cif.gz | 16.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23251 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23251 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23251 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23251 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23251.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23251.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ferrichrome importer FhuCDB | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.799 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : holocomplex of ferrichrome importer FhuCDB
| 全体 | 名称: holocomplex of ferrichrome importer FhuCDB |
|---|---|
| 要素 |
|
-超分子 #1: holocomplex of ferrichrome importer FhuCDB
| 超分子 | 名称: holocomplex of ferrichrome importer FhuCDB / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Iron(3+)-hydroxamate import system permease protein FhuB
| 分子 | 名称: Iron(3+)-hydroxamate import system permease protein FhuB タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 71.574867 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSKRIALFPA LLLALLVIVA TALTWMNFSQ ALPRSQWAQA AWSPNINVIE QMIFHYSLLP RLAISLLVGA GLGLVGVLFQ QVLRNPLAE PTTLGVATGA QLGITVTTLW AIPGAMASQF AALAGACVVG LIVFGVAWGK RLSPVTLILA GLVVSLYCGA I NQLLVIFH ...文字列: MSKRIALFPA LLLALLVIVA TALTWMNFSQ ALPRSQWAQA AWSPNINVIE QMIFHYSLLP RLAISLLVGA GLGLVGVLFQ QVLRNPLAE PTTLGVATGA QLGITVTTLW AIPGAMASQF AALAGACVVG LIVFGVAWGK RLSPVTLILA GLVVSLYCGA I NQLLVIFH HDQLQSMFLW STGTLTQTDW GGVERLWPQL LGGVMLTLLL LRPLTLMGLD DGVARNLGLA LSLARLAALS LA IVISALL VNAVGIIGFI GLFAPLLAKM LGARRLLPRL MLASLIGALI LWLSDQIILW LTRVWMEVST GSVTALIGAP LLL WLLPRL RSISAPDMKV NDRVATERQH VLAFALAGGV LLLMAVVVAL SFGRDAHGWT WASGALLDDL MPWRWPRIMA ALFA GVMLA VAGCIIQRLT GNPMASPEVL GISSGAAFGV VLMLFLVPGN AFGWLLPAGS LGAAVTLLII MIAAGRGGFS PHRML LAGM ALSTAFTMLL MMLQASGDPR MAQVLTWISG STYNATDAQV WRTGIVMVIL LAITPLCRRW LTILPLGGDT ARAVGM ALT PTRIALLLLA ACLTATATMT IGPLSFVGLM APHIARMMGF RRTMPHIVIS ALVGGLLLVF ADWCGRMVLF PFQIPAG LL STFIGAPYFI YLLRKQSRHH HHHHHH UniProtKB: Iron(3+)-hydroxamate import system permease protein FhuB |
-分子 #2: Iron(3+)-hydroxamate-binding protein FhuD
| 分子 | 名称: Iron(3+)-hydroxamate-binding protein FhuD / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 33.030258 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSGLPLISRR RLLTAMALSP LLWQMNTAHA AAIDPNRIVA LEWLPVELLL ALGIVPYGVA DTINYRLWVS EPPLPDSVID VGLRTEPNL ELLTTMRPSF MVWSAGYGPS PEMLARIAPG RGFNFSDGKQ PLAMARKSLT EMADLLNLQS AAETHLAQYE D FIRSMKPR ...文字列: MSGLPLISRR RLLTAMALSP LLWQMNTAHA AAIDPNRIVA LEWLPVELLL ALGIVPYGVA DTINYRLWVS EPPLPDSVID VGLRTEPNL ELLTTMRPSF MVWSAGYGPS PEMLARIAPG RGFNFSDGKQ PLAMARKSLT EMADLLNLQS AAETHLAQYE D FIRSMKPR FVKRGARPLL LTTLIDPRHM LVFGPNSLFQ EILDEYGIPN AWQGETNFWG STAVSIDRLA AYKDVDVLCF DH DNSKDMD ALMATPLWQA MPFVRAGRFQ RVPAVWFYGA TLSAMHFVRV LDNAIGGKA UniProtKB: Iron(3+)-hydroxamate-binding protein FhuD |
-分子 #3: Iron(3+)-hydroxamate import ATP-binding protein FhuC
| 分子 | 名称: Iron(3+)-hydroxamate import ATP-binding protein FhuC タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO / EC番号: ABC-type ferric hydroxamate transporter |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 28.921424 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MQEYTNHSDT TFALRNISFR VPGRTLLHPL SLTFPAGKVT GLIGHNGSGK STLLKMLGRH QPPSEGEILL DAQPLESWSS KAFARKVAY LPQQLPPAEG MTVRELVAIG RYPWHGALGR FGAADREKVE EAISLVGLKP LAHRLVDSLS GGERQRAWIA M LVAQDSRC ...文字列: MQEYTNHSDT TFALRNISFR VPGRTLLHPL SLTFPAGKVT GLIGHNGSGK STLLKMLGRH QPPSEGEILL DAQPLESWSS KAFARKVAY LPQQLPPAEG MTVRELVAIG RYPWHGALGR FGAADREKVE EAISLVGLKP LAHRLVDSLS GGERQRAWIA M LVAQDSRC LLLDEPTSAL DIAHQVDVLS LVHRLSQERG LTVIAVLHDI NMAARYCDYL VALRGGEMIA QGTPAEIMRG ET LEMIYGI PMGILPHPAG AAPVSFVY UniProtKB: Iron(3+)-hydroxamate import ATP-binding protein FhuC |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 65.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)