+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22441 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The bacteriophage Phi-29 viral genome packaging motor assembly | |||||||||
 マップデータ マップデータ | Focused asymmetric reconstruction of the bacteriophage phi29 dsDNA packaging motor assembly at 4.1 A. | |||||||||
 試料 試料 | Bacillus virus phi29 viral genome packaging motor assembly != Bacillus virus phi29 Bacillus virus phi29 viral genome packaging motor assembly
| |||||||||
 キーワード キーワード | packaging motor / ATPase / MOTOR PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral DNA genome packaging / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / ATP hydrolysis activity / DNA binding / RNA binding / ATP binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   Bacillus phage phi29 (ファージ) / Bacillus phage phi29 (ファージ) /  Bacillus virus phi29 (ウイルス) Bacillus virus phi29 (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.1 Å | |||||||||
 データ登録者 データ登録者 | White MA / Woodson M | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2021 ジャーナル: Sci Adv / 年: 2021タイトル: A viral genome packaging motor transitions between cyclic and helical symmetry to translocate dsDNA. 著者: Michael Woodson / Joshua Pajak / Bryon P Mahler / Wei Zhao / Wei Zhang / Gaurav Arya / Mark A White / Paul J Jardine / Marc C Morais /  要旨: Molecular segregation and biopolymer manipulation require the action of molecular motors to do work by applying directional forces to macromolecules. The additional strand conserved E (ASCE) ring ...Molecular segregation and biopolymer manipulation require the action of molecular motors to do work by applying directional forces to macromolecules. The additional strand conserved E (ASCE) ring motors are an ancient family of molecular motors responsible for diverse biological polymer manipulation tasks. Viruses use ASCE segregation motors to package their genomes into their protein capsids and provide accessible experimental systems due to their relative simplicity. We show by cryo-EM-focused image reconstruction that ASCE ATPases in viral double-stranded DNA (dsDNA) packaging motors adopt helical symmetry complementary to their dsDNA substrates. Together with previous data, our results suggest that these motors cycle between helical and planar configurations, providing a possible mechanism for directional translocation of DNA. Similar changes in quaternary structure have been observed for proteasome and helicase motors, suggesting an ancient and common mechanism of force generation that has been adapted for specific tasks over the course of evolution. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22441.map.gz emd_22441.map.gz | 113.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22441-v30.xml emd-22441-v30.xml emd-22441.xml emd-22441.xml | 17.6 KB 17.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
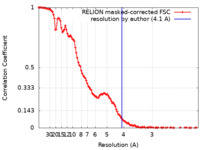
| FSC (解像度算出) |  emd_22441_fsc.xml emd_22441_fsc.xml | 11.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22441.png emd_22441.png | 251.7 KB | ||
| Filedesc metadata |  emd-22441.cif.gz emd-22441.cif.gz | 6.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22441 http://ftp.pdbj.org/pub/emdb/structures/EMD-22441 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22441 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22441 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22441_validation.pdf.gz emd_22441_validation.pdf.gz | 714.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22441_full_validation.pdf.gz emd_22441_full_validation.pdf.gz | 713.6 KB | 表示 | |
| XML形式データ |  emd_22441_validation.xml.gz emd_22441_validation.xml.gz | 12.1 KB | 表示 | |
| CIF形式データ |  emd_22441_validation.cif.gz emd_22441_validation.cif.gz | 16.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22441 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22441 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22441 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22441 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22441.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22441.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused asymmetric reconstruction of the bacteriophage phi29 dsDNA packaging motor assembly at 4.1 A. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Bacillus virus phi29 viral genome packaging motor assembly
| 全体 | 名称: Bacillus virus phi29 viral genome packaging motor assembly |
|---|---|
| 要素 |
|
-超分子 #1: Bacillus virus phi29
| 超分子 | 名称: Bacillus virus phi29 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 / NCBI-ID: 10756 / 生物種: Bacillus virus phi29 / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
-分子 #1: DNA packaging protein
| 分子 | 名称: DNA packaging protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 5 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:   Bacillus phage phi29 (ファージ) Bacillus phage phi29 (ファージ) |
| 分子量 | 理論値: 39.010406 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDKSLFYNPQ KMLSYDRILN FVIGARGIGK SYAMKVYPIN RFIKYGEQFI YVRRYKPELA KVSNYFNDVA QEFPDHELVV KGRRFYIDG KLAGWAIPLS VWQSEKSNAY PNVSTIVFDE FIREKDNSNY IPNEVSALLN LMDTVFRNRE RVRCICLSNA V SVVNPYFL ...文字列: MDKSLFYNPQ KMLSYDRILN FVIGARGIGK SYAMKVYPIN RFIKYGEQFI YVRRYKPELA KVSNYFNDVA QEFPDHELVV KGRRFYIDG KLAGWAIPLS VWQSEKSNAY PNVSTIVFDE FIREKDNSNY IPNEVSALLN LMDTVFRNRE RVRCICLSNA V SVVNPYFL FFNLVPDVNK RFNVYDDALI EIPDSLDFSS ERRKTRFGRL IDGTEYGEMS LDNQFIGDSQ VFIEKRSKDS KF VFSIVYN GFTLGVWVDV NQGLMYIDTA HDPSTKNVYT LTTDDLNENM MLITNYKNNY HLRKLASAFM NGYLRFDNQV IRN IAYELF RKMRIQ UniProtKB: DNA packaging protein |
-分子 #2: pRNA (117-MER)
| 分子 | 名称: pRNA (117-MER) / タイプ: rna / ID: 2 / コピー数: 5 |
|---|---|
| 由来(天然) | 生物種:  Bacillus virus phi29 (ウイルス) Bacillus virus phi29 (ウイルス) |
| 分子量 | 理論値: 37.469012 KDa |
| 配列 | 文字列: GGAAUGGUAC GGUACUUCCA UUGUCAUGUG UAUGUUGGGG AUUAAACCCU GAUUGAGUUC AGCCCACAUA CUUUGUUGAU UGGUUGUCA AUCAUGGCAA AAGUGCACGC UACUUUCC |
-分子 #3: DNA (60-MER)
| 分子 | 名称: DNA (60-MER) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Bacillus virus phi29 (ウイルス) Bacillus virus phi29 (ウイルス) |
| 分子量 | 理論値: 18.491848 KDa |
| 配列 | 文字列: (DG)(DT)(DC)(DA)(DG)(DT)(DC)(DA)(DG)(DT) (DC)(DA)(DG)(DT)(DC)(DA)(DG)(DT)(DC)(DA) (DG)(DT)(DC)(DA)(DG)(DT)(DC)(DA)(DG) (DT)(DC)(DA)(DG)(DT)(DC)(DA)(DG)(DT)(DC) (DA) (DG)(DT)(DC)(DA)(DG) ...文字列: (DG)(DT)(DC)(DA)(DG)(DT)(DC)(DA)(DG)(DT) (DC)(DA)(DG)(DT)(DC)(DA)(DG)(DT)(DC)(DA) (DG)(DT)(DC)(DA)(DG)(DT)(DC)(DA)(DG) (DT)(DC)(DA)(DG)(DT)(DC)(DA)(DG)(DT)(DC) (DA) (DG)(DT)(DC)(DA)(DG)(DT)(DC)(DA) (DG)(DT)(DC)(DA)(DG)(DT)(DC)(DA)(DG)(DT) (DC)(DA) |
-分子 #4: DNA (60-MER)
| 分子 | 名称: DNA (60-MER) / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Bacillus virus phi29 (ウイルス) Bacillus virus phi29 (ウイルス) |
| 分子量 | 理論値: 18.49185 KDa |
| 配列 | 文字列: (DT)(DG)(DA)(DC)(DT)(DG)(DA)(DC)(DT)(DG) (DA)(DC)(DT)(DG)(DA)(DC)(DT)(DG)(DA)(DC) (DT)(DG)(DA)(DC)(DT)(DG)(DA)(DC)(DT) (DG)(DA)(DC)(DT)(DG)(DA)(DC)(DT)(DG)(DA) (DC) (DT)(DG)(DA)(DC)(DT) ...文字列: (DT)(DG)(DA)(DC)(DT)(DG)(DA)(DC)(DT)(DG) (DA)(DC)(DT)(DG)(DA)(DC)(DT)(DG)(DA)(DC) (DT)(DG)(DA)(DC)(DT)(DG)(DA)(DC)(DT) (DG)(DA)(DC)(DT)(DG)(DA)(DC)(DT)(DG)(DA) (DC) (DT)(DG)(DA)(DC)(DT)(DG)(DA)(DC) (DT)(DG)(DA)(DC)(DT)(DG)(DA)(DC)(DT)(DG) (DA)(DC) |
-分子 #5: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 5 / コピー数: 3 / 式: AGS |
|---|---|
| 分子量 | 理論値: 523.247 Da |
| Chemical component information |  ChemComp-AGS: |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 3 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY | ||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF 2002 |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 7676 pixel / デジタル化 - サイズ - 縦: 7420 pixel / デジタル化 - 画像ごとのフレーム数: 0-44 / 平均電子線量: 41.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 4.2 µm / 最小 デフォーカス(補正後): 1.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.2 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)