+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22379 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Herpes Simplex Virus Type 1 A-capsid with Portal Vertex | |||||||||
 マップデータ マップデータ | Herpes sipmlex virus Type 1 A-capsid with the portal vertex | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Human alphaherpesvirus 1 (ヘルペスウイルス) Human alphaherpesvirus 1 (ヘルペスウイルス) | |||||||||
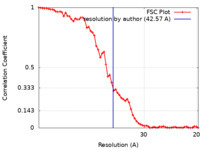
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 42.57 Å | |||||||||
 データ登録者 データ登録者 | Buch MHC / Heymann JB / Newcomb WW / Winkler DC / Steven AC | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: mBio / 年: 2021 ジャーナル: mBio / 年: 2021タイトル: Cryo-Electron Tomography of the Herpesvirus Procapsid Reveals Interactions of the Portal with the Scaffold and a Shift on Maturation. 著者: Michael H C Buch / William W Newcomb / Dennis C Winkler / Alasdair C Steven / J Bernard Heymann /  要旨: Herpes simplex virus 1 (HSV-1) requires seven proteins to package its genome through a vertex in its capsid, one of which is the portal protein, pUL6. The portal protein is also thought to facilitate ...Herpes simplex virus 1 (HSV-1) requires seven proteins to package its genome through a vertex in its capsid, one of which is the portal protein, pUL6. The portal protein is also thought to facilitate assembly of the procapsid. While the portal has been visualized in mature capsids, we aimed to elucidate its role in the assembly and maturation of procapsids using cryo-electron tomography (cryoET). We identified the portal vertex in individual procapsids, calculated a subtomogram average, and compared that with the portal vertex in empty mature capsids (A-capsids). The resulting maps show the portal on the interior surface with its narrower end facing outwards, while maintaining close contact with the capsid shell. In the procapsid, the portal is embedded in the underlying scaffold, suggesting that assembly involves a portal-scaffold complex. During maturation, the capsid shell angularizes with a corresponding outward movement of the vertices. We found that in A-capsids, the portal translocates outward further than the adjacent capsomers and strengthens its contacts with the capsid shell. Our methodology also allowed us to determine the number of portal vertices in each capsid, with most having one per capsid, but some none or two, and rarely three. The predominance of a single portal per capsid supports facilitation of the assembly of the procapsid. Herpes simplex virus 1 (HSV-1) infects a majority of humans, causing mostly mild disease but in some cases progressing toward life-threatening encephalitis. Understanding the life cycle of the virus is important to devise countermeasures. Production of the virion starts with the assembly of an icosahedral procapsid, which includes DNA packaging proteins at a vertex, one of which is the dodecameric portal protein. The procapsid then undergoes maturation and DNA packaging through the portal, driven by a terminase complex. We used cryo-electron tomography to visualize the portal in procapsids and compare them to mature empty capsids. We found the portal located inside one vertex interacting with the scaffold protein in the procapsid. On maturation, the scaffold is cleaved and dissociates, the capsid angularizes, and the portal moves outward, interacting closely with the capsid shell. These transformations may provide a basis for the development of drugs to prevent HSV-1 infections. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22379.map.gz emd_22379.map.gz | 304.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22379-v30.xml emd-22379-v30.xml emd-22379.xml emd-22379.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22379_fsc.xml emd_22379_fsc.xml | 6.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22379.png emd_22379.png | 108.8 KB | ||
| マスクデータ |  emd_22379_msk_1.map emd_22379_msk_1.map | 347.6 MB |  マスクマップ マスクマップ | |
| その他 |  emd_22379_half_map_1.map.gz emd_22379_half_map_1.map.gz emd_22379_half_map_2.map.gz emd_22379_half_map_2.map.gz | 313.4 MB 314.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22379 http://ftp.pdbj.org/pub/emdb/structures/EMD-22379 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22379 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22379 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22379.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22379.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Herpes sipmlex virus Type 1 A-capsid with the portal vertex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.66 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
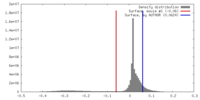
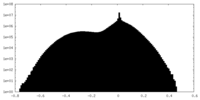
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_22379_msk_1.map emd_22379_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
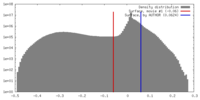
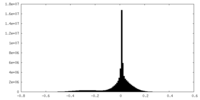
| 密度ヒストグラム |
-ハーフマップ: Herpes Simplex Virus Type 1 A-capsid with Portal Vertex
| ファイル | emd_22379_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Herpes Simplex Virus Type 1 A-capsid with Portal Vertex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
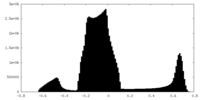
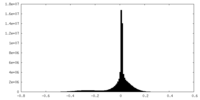
| 密度ヒストグラム |
-ハーフマップ: Herpes Simplex Virus Type 1 A-capsid with Portal Vertex
| ファイル | emd_22379_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Herpes Simplex Virus Type 1 A-capsid with Portal Vertex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
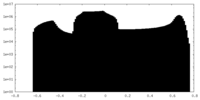
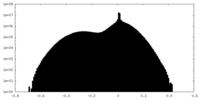
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human alphaherpesvirus 1
| 全体 | 名称:   Human alphaherpesvirus 1 (ヘルペスウイルス) Human alphaherpesvirus 1 (ヘルペスウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Human alphaherpesvirus 1
| 超分子 | 名称: Human alphaherpesvirus 1 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Capsids were obtained from infected Vero cells. Purification steps were carried out at 4 deg. Harvested cells were resuspended in TNE buffer and lysed by sonication. Nuclei were lysed with 1. ...詳細: Capsids were obtained from infected Vero cells. Purification steps were carried out at 4 deg. Harvested cells were resuspended in TNE buffer and lysed by sonication. Nuclei were lysed with 1.5% TX-100, and a mix of A-, B-, and C-capsids was pelleted through a 30% sucrose cushion. The resuspended capsid mix was applied to a 20-50% sucrose gradient, and the band containing A-capsids was isolated. NCBI-ID: 10298 / 生物種: Human alphaherpesvirus 1 / Sci species strain: KOS / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| Host system | 生物種:  Chlorocebus aethiops (ミドリザル) Chlorocebus aethiops (ミドリザル) |
| ウイルス殻 | Shell ID: 1 / 名称: A-capsid / 直径: 1250.0 Å / T番号(三角分割数): 16 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER 詳細: Grids were plasma-cleaned in a Model 1020 plasma cleaner (Fischione, Export, PA). | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / 装置: LEICA EM GP 詳細: Sample was mixed with Aurion Anionic 10 nm BSA gold tracer (EMS) at a 1:1 ratio, and 3 uL were applied to Quantifoil copper grids covered with 300-mesh holey carbon (EMS). Excess liquid was ...詳細: Sample was mixed with Aurion Anionic 10 nm BSA gold tracer (EMS) at a 1:1 ratio, and 3 uL were applied to Quantifoil copper grids covered with 300-mesh holey carbon (EMS). Excess liquid was blotted off for 2.5 s, and grids were flash-frozen in liquid ethane.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2200FS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 平均露光時間: 1.0 sec. / 平均電子線量: 3.1 e/Å2 詳細: Tilts were imaged in movie mode with 3 movies at on average 0.5 seconds each. |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 20000 |
| 試料ステージ | 試料ホルダーモデル: GATAN 914 HIGH TILT LIQUID NITROGEN CRYO TRANSFER TOMOGRAPHY HOLDER ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)