+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22041 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Asterix/Gtsf1 from mouse (residues 1-45; zinc finger 1) bound to co-purifying tRNA | |||||||||
 マップデータ マップデータ | Asterix/Gtsf1 from mouse (residues 1-45; zinc finger 1) bound to co-purifying tRNA | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   | |||||||||
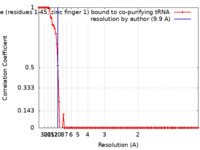
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 9.9 Å | |||||||||
 データ登録者 データ登録者 | Ipsaro JJ / Joshua-Tor L | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2021 ジャーナル: Cell Rep / 年: 2021タイトル: Asterix/Gtsf1 links tRNAs and piRNA silencing of retrotransposons. 著者: Jonathan J Ipsaro / Paul A O'Brien / Shibani Bhattacharya / Arthur G Palmer / Leemor Joshua-Tor /  要旨: The Piwi-interacting RNA (piRNA) pathway safeguards genomic integrity by silencing transposable elements (transposons) in the germline. While Piwi is the central piRNA factor, others including ...The Piwi-interacting RNA (piRNA) pathway safeguards genomic integrity by silencing transposable elements (transposons) in the germline. While Piwi is the central piRNA factor, others including Asterix/Gtsf1 have also been demonstrated to be critical for effective silencing. Here, using enhanced crosslinking and immunoprecipitation (eCLIP) with a custom informatic pipeline, we show that Asterix/Gtsf1 specifically binds tRNAs in cellular contexts. We determined the structure of mouse Gtsf1 by NMR spectroscopy and identified the RNA-binding interface on the protein's first zinc finger, which was corroborated by biochemical analysis as well as cryo-EM structures of Gtsf1 in complex with co-purifying tRNA. Consistent with the known dependence of long terminal repeat (LTR) retrotransposons on tRNA primers, we demonstrate that LTR retrotransposons are, in fact, preferentially de-repressed in Asterix mutants. Together, these findings link Asterix/Gtsf1, tRNAs, and LTR retrotransposon silencing and suggest that Asterix exploits tRNA dependence to identify transposon transcripts and promote piRNA silencing. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22041.map.gz emd_22041.map.gz | 68.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22041-v30.xml emd-22041-v30.xml emd-22041.xml emd-22041.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22041_fsc.xml emd_22041_fsc.xml | 9.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22041.png emd_22041.png | 58.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22041 http://ftp.pdbj.org/pub/emdb/structures/EMD-22041 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22041 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22041 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22041_validation.pdf.gz emd_22041_validation.pdf.gz | 309.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22041_full_validation.pdf.gz emd_22041_full_validation.pdf.gz | 309 KB | 表示 | |
| XML形式データ |  emd_22041_validation.xml.gz emd_22041_validation.xml.gz | 11.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22041 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22041 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22041 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22041 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22041.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22041.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Asterix/Gtsf1 from mouse (residues 1-45; zinc finger 1) bound to co-purifying tRNA | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.6262 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Complex of Asterix/Gtsf1 from mouse (residues 1-45; zinc finger 1...
| 全体 | 名称: Complex of Asterix/Gtsf1 from mouse (residues 1-45; zinc finger 1) bound to co-purifying tRNA |
|---|---|
| 要素 |
|
-超分子 #1: Complex of Asterix/Gtsf1 from mouse (residues 1-45; zinc finger 1...
| 超分子 | 名称: Complex of Asterix/Gtsf1 from mouse (residues 1-45; zinc finger 1) bound to co-purifying tRNA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Co-purifying RNA in this sample was derived from the expression host (Sf9) and represents a heterogenous population. For more details on the identities of these nucleic acids, please see the original publication. |
|---|---|
| 分子量 | 理論値: 30 KDa |
-超分子 #2: Gametocyte-specific factor 1 (residues 1-45; zinc finger 1)
| 超分子 | 名称: Gametocyte-specific factor 1 (residues 1-45; zinc finger 1) タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: Co-purifying tRNA (heterogeneous population)
| 超分子 | 名称: Co-purifying tRNA (heterogeneous population) / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Gametocyte-specific factor 1
| 分子 | 名称: Gametocyte-specific factor 1 / タイプ: protein_or_peptide / ID: 1 詳細: Truncation construct Asterix/Gtsf1 from mouse (residues 1-45; zinc finger 1), with C-terminal TEV-Strep tag 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEDTYIDSLD PEKLLQCPYD KNHQIRACRF PYHLIKCRKN HPDVAENLYF QGASAWSHPQ FEKGGGSGGG SGGSAWSHPQ FEK |
-分子 #2: tRNA
| 分子 | 名称: tRNA / タイプ: rna / ID: 2 詳細: The sequence above represents the most abundant RNA as assessed by next-generation sequencing bound to the full-length protein (see related entry). This sequence made up approximately 15% of ...詳細: The sequence above represents the most abundant RNA as assessed by next-generation sequencing bound to the full-length protein (see related entry). This sequence made up approximately 15% of the sequencing library, with additional sequences only 1 nucleotide different making up approximately 10% more of the library. |
|---|---|
| 由来(天然) | 生物種:  組織: ovary |
| 配列 | 文字列: TCTTCGGTAG TATAGTGGTC AGTATCCCCG CCTGTCACGC GGGAGACCGG GGTTCGATTC CCCGCCGGAG AGCCA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.25 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 4.0 kPa 詳細: Grids were purchased from Electron Microscopy Sciences. Lacey carbon film, 300 mesh. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 298 K / 装置: LEICA EM GP / 詳細: Blotted for 2.5 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 2461 / 平均露光時間: 4.0 sec. / 平均電子線量: 76.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 215000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)