+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21941 | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | KIF14[391-748] - ADP-AlFx in complex with a microtubule | |||||||||||||||||||||||||||
 マップデータ マップデータ | Main map. Locally refined single unit. Not helically averaged. | |||||||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||||||||
 キーワード キーワード | KIF14 / kinesin / motility / microtubule / tubulin / MOTOR PROTEIN | |||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cerebellar granular layer structural organization / regulation of cell maturation / RHO GTPases activate CIT / cerebellar Purkinje cell layer structural organization / negative regulation of integrin activation / RND2 GTPase cycle / RND1 GTPase cycle / cell proliferation in forebrain / regulation of Rap protein signal transduction / cerebellar cortex development ...cerebellar granular layer structural organization / regulation of cell maturation / RHO GTPases activate CIT / cerebellar Purkinje cell layer structural organization / negative regulation of integrin activation / RND2 GTPase cycle / RND1 GTPase cycle / cell proliferation in forebrain / regulation of Rap protein signal transduction / cerebellar cortex development / activation of protein kinase activity / olfactory bulb development / Flemming body / plus-end-directed microtubule motor activity / Microtubule-dependent trafficking of connexons from Golgi to the plasma membrane / Resolution of Sister Chromatid Cohesion / Hedgehog 'off' state / Cilium Assembly / Intraflagellar transport / COPI-dependent Golgi-to-ER retrograde traffic / Mitotic Prometaphase / Carboxyterminal post-translational modifications of tubulin / RHOH GTPase cycle / EML4 and NUDC in mitotic spindle formation / Sealing of the nuclear envelope (NE) by ESCRT-III / Kinesins / PKR-mediated signaling / Separation of Sister Chromatids / The role of GTSE1 in G2/M progression after G2 checkpoint / Aggrephagy / RHO GTPases activate IQGAPs / RHO GTPases Activate Formins / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / MHC class II antigen presentation / Recruitment of NuMA to mitotic centrosomes / regulation of myelination / COPI-mediated anterograde transport / SCF-dependent proteasomal ubiquitin-dependent protein catabolic process / microtubule-based movement / mitotic metaphase chromosome alignment / positive regulation of cytokinesis / spindle midzone / regulation of G1/S transition of mitotic cell cycle / regulation of neuron apoptotic process / regulation of cell adhesion / microtubule-based process / regulation of G2/M transition of mitotic cell cycle / substrate adhesion-dependent cell spreading / tubulin binding / regulation of cell migration / hippocampus development / PDZ domain binding / regulation of cell growth / establishment of protein localization / cerebral cortex development / structural constituent of cytoskeleton / microtubule cytoskeleton organization / mitotic cell cycle / microtubule cytoskeleton / midbody / microtubule binding / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / proteasome-mediated ubiquitin-dependent protein catabolic process / negative regulation of neuron apoptotic process / microtubule / cell division / GTPase activity / positive regulation of cell population proliferation / protein kinase binding / negative regulation of apoptotic process / GTP binding / ATP hydrolysis activity / ATP binding / metal ion binding / nucleus / membrane / plasma membrane / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||||||||||||||||||||
| 生物種 |   | |||||||||||||||||||||||||||
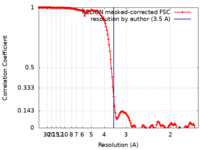
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||||||||||||||||||||
 データ登録者 データ登録者 | Benoit MPMH / Asenjo AB | |||||||||||||||||||||||||||
| 資金援助 |  米国, 米国,  カナダ, 8件 カナダ, 8件
| |||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Structural basis of mechano-chemical coupling by the mitotic kinesin KIF14. 著者: Matthieu P M H Benoit / Ana B Asenjo / Mohammadjavad Paydar / Sabin Dhakal / Benjamin H Kwok / Hernando Sosa /   要旨: KIF14 is a mitotic kinesin whose malfunction is associated with cerebral and renal developmental defects and several cancers. Like other kinesins, KIF14 couples ATP hydrolysis and microtubule binding ...KIF14 is a mitotic kinesin whose malfunction is associated with cerebral and renal developmental defects and several cancers. Like other kinesins, KIF14 couples ATP hydrolysis and microtubule binding to the generation of mechanical work, but the coupling mechanism between these processes is still not fully clear. Here we report 20 high-resolution (2.7-3.9 Å) cryo-electron microscopy KIF14-microtubule structures with complementary functional assays. Analysis procedures were implemented to separate coexisting conformations of microtubule-bound monomeric and dimeric KIF14 constructs. The data provide a comprehensive view of the microtubule and nucleotide induced KIF14 conformational changes. It shows that: 1) microtubule binding, the nucleotide species, and the neck-linker domain govern the transition between three major conformations of the motor domain; 2) an undocked neck-linker prevents the nucleotide-binding pocket to fully close and dampens ATP hydrolysis; 3) 13 neck-linker residues are required to assume a stable docked conformation; 4) the neck-linker position controls the hydrolysis rather than the nucleotide binding step; 5) the two motor domains of KIF14 dimers adopt distinct conformations when bound to the microtubule; and 6) the formation of the two-heads-bound-state introduces structural changes in both motor domains of KIF14 dimers. These observations provide the structural basis for a coordinated chemo-mechanical kinesin translocation model. | |||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21941.map.gz emd_21941.map.gz | 7.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21941-v30.xml emd-21941-v30.xml emd-21941.xml emd-21941.xml | 35.2 KB 35.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21941_fsc.xml emd_21941_fsc.xml | 14.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21941.png emd_21941.png | 156.4 KB | ||
| マスクデータ |  emd_21941_msk_1.map emd_21941_msk_1.map emd_21941_msk_2.map emd_21941_msk_2.map emd_21941_msk_3.map emd_21941_msk_3.map emd_21941_msk_4.map emd_21941_msk_4.map emd_21941_msk_5.map emd_21941_msk_5.map emd_21941_msk_6.map emd_21941_msk_6.map | 1.1 GB 1.1 GB 274.6 MB 274.6 MB 1.1 GB 274.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-21941.cif.gz emd-21941.cif.gz | 8.3 KB | ||
| その他 |  emd_21941_additional_1.map.gz emd_21941_additional_1.map.gz emd_21941_additional_2.map.gz emd_21941_additional_2.map.gz emd_21941_additional_3.map.gz emd_21941_additional_3.map.gz emd_21941_half_map_1.map.gz emd_21941_half_map_1.map.gz emd_21941_half_map_2.map.gz emd_21941_half_map_2.map.gz | 210.9 MB 827.6 MB 827.6 MB 218.1 MB 218 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21941 http://ftp.pdbj.org/pub/emdb/structures/EMD-21941 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21941 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21941 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21941_validation.pdf.gz emd_21941_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21941_full_validation.pdf.gz emd_21941_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_21941_validation.xml.gz emd_21941_validation.xml.gz | 22.1 KB | 表示 | |
| CIF形式データ |  emd_21941_validation.cif.gz emd_21941_validation.cif.gz | 29.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21941 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21941 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21941 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21941 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6wwnMC  6wweC  6wwfC  6wwgC  6wwhC  6wwiC  6wwjC  6wwkC  6wwlC  6wwmC  6wwoC  6wwpC  6wwqC  6wwrC  6wwsC  6wwtC  6wwuC  6wwvC  7lvqC  7lvrC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21941.map.gz / 形式: CCP4 / 大きさ: 274.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21941.map.gz / 形式: CCP4 / 大きさ: 274.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main map. Locally refined single unit. Not helically averaged. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.825 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
+マスク #1
+マスク #2
+マスク #3
+マスク #4
+マスク #5
+マスク #6
+追加マップ: Helical reconstruction.
+追加マップ: Helical reconstruction half map 2 (gold standard).
+追加マップ: Helical reconstruction half map 1 (gold standard).
+ハーフマップ: Half map 1 (gold standard). Locally refined single...
+ハーフマップ: Half map 2 (gold standard). Locally refined single...
- 試料の構成要素
試料の構成要素
+全体 : Kif14[391-748] - ADP-AlFx in complex with a microtubule
+超分子 #1: Kif14[391-748] - ADP-AlFx in complex with a microtubule
+超分子 #2: KIF14[391-748] - ADP-AlFx
+超分子 #3: Microtubule
+分子 #1: Tubulin alpha-1B chain
+分子 #2: Tubulin beta-2B chain
+分子 #3: Kinesin-like protein KIF14
+分子 #4: GUANOSINE-5'-TRIPHOSPHATE
+分子 #5: MAGNESIUM ION
+分子 #6: GUANOSINE-5'-DIPHOSPHATE
+分子 #7: TAXOL
+分子 #8: ALUMINUM FLUORIDE
+分子 #9: ADENOSINE-5'-DIPHOSPHATE
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 6.8 構成要素:
| ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: UltrAuFoil / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING | ||||||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-40 / 平均電子線量: 70.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 60606 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-6wwn: |
 ムービー
ムービー コントローラー
コントローラー































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)