+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21150 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Xenopus tropicalis pannexin 1 channel | |||||||||
 マップデータ マップデータ | Sharpened map of a pannexin1 channel | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | channel / ATP release / heptamer / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報wide pore channel activity / positive regulation of interleukin-1 production / gap junction / monoatomic cation transport / cell-cell signaling / monoatomic ion transmembrane transport / endoplasmic reticulum membrane / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 | ||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.02 Å | |||||||||
 データ登録者 データ登録者 | Syrjanen JL / Furukawa H | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2020 ジャーナル: Elife / 年: 2020タイトル: The Cryo-EM structure of pannexin 1 reveals unique motifs for ion selection and inhibition. 著者: Kevin Michalski / Johanna L Syrjanen / Erik Henze / Julia Kumpf / Hiro Furukawa / Toshimitsu Kawate /  要旨: Pannexins are large-pore forming channels responsible for ATP release under a variety of physiological and pathological conditions. Although predicted to share similar membrane topology with other ...Pannexins are large-pore forming channels responsible for ATP release under a variety of physiological and pathological conditions. Although predicted to share similar membrane topology with other large-pore forming proteins such as connexins, innexins, and LRRC8, pannexins have minimal sequence similarity to these protein families. Here, we present the cryo-EM structure of a frog pannexin 1 (Panx1) channel at 3.0 Å. We find that Panx1 protomers harbor four transmembrane helices similar in arrangement to other large-pore forming proteins but assemble as a heptameric channel with a unique constriction formed by Trp74 in the first extracellular loop. Mutating Trp74 or the nearby Arg75 disrupt ion selectivity, whereas altering residues in the hydrophobic groove formed by the two extracellular loops abrogates channel inhibition by carbenoxolone. Our structural and functional study establishes the extracellular loops as important structural motifs for ion selectivity and channel inhibition in Panx1. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21150.map.gz emd_21150.map.gz | 59.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21150-v30.xml emd-21150-v30.xml emd-21150.xml emd-21150.xml | 11.9 KB 11.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_21150.png emd_21150.png | 153.5 KB | ||
| Filedesc metadata |  emd-21150.cif.gz emd-21150.cif.gz | 5.2 KB | ||
| その他 |  emd_21150_additional.map.gz emd_21150_additional.map.gz | 59.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21150 http://ftp.pdbj.org/pub/emdb/structures/EMD-21150 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21150 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21150 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21150_validation.pdf.gz emd_21150_validation.pdf.gz | 641.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21150_full_validation.pdf.gz emd_21150_full_validation.pdf.gz | 640.8 KB | 表示 | |
| XML形式データ |  emd_21150_validation.xml.gz emd_21150_validation.xml.gz | 6.5 KB | 表示 | |
| CIF形式データ |  emd_21150_validation.cif.gz emd_21150_validation.cif.gz | 7.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21150 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21150 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21150 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21150 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21150.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21150.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map of a pannexin1 channel | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
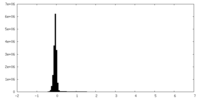
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: unsharpened map of a pannexin1 channel
| ファイル | emd_21150_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map of a pannexin1 channel | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
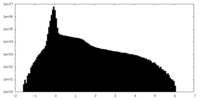
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Heptameric pannexin1 complex assembly
| 全体 | 名称: Heptameric pannexin1 complex assembly |
|---|---|
| 要素 |
|
-超分子 #1: Heptameric pannexin1 complex assembly
| 超分子 | 名称: Heptameric pannexin1 complex assembly / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種: |
-分子 #1: Pannexin
| 分子 | 名称: Pannexin / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 39.424148 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAIAHIATEY VFSDFLLKDP PESKYKGLRL ELAVDKLVSC IAVGLPLLLI SLAFAQEITL GSQISCFAPT SFSWRQAAYV DSFCWAAVQ QKHLSQSDSG NVPLWLHKFF PYILLLVAVL LYLPNLFWRF TAAPHLSSDL KFVMEELDKC YNRDIKDIKA A NNLNSSDK ...文字列: MAIAHIATEY VFSDFLLKDP PESKYKGLRL ELAVDKLVSC IAVGLPLLLI SLAFAQEITL GSQISCFAPT SFSWRQAAYV DSFCWAAVQ QKHLSQSDSG NVPLWLHKFF PYILLLVAVL LYLPNLFWRF TAAPHLSSDL KFVMEELDKC YNRDIKDIKA A NNLNSSDK RDYPIVEQYL KTKNNSYGLI IKYLICRVVT LIIVFTACIY LGYYISLFSL TDEFTCNIRT GILRNDTALP PL VQCKLIA VGVFRLLSYI NLIIYVLIMP FIIYAMLVPF RKTANVLKVY EVLPTFSVQQ APSKTYDDHS LFLLFLEENV SEL KSYKFL KVLENIKASS WSHPQFEK UniProtKB: Pannexin |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 85 % / チャンバー内温度: 288.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 57.3 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C7 (7回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.02 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cisTEM / 使用した粒子像数: 90185 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)