+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20911 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Group 1 H1 influenza stem nanoparticle with a group 2-like N-linked glycosylation site at position 38 | |||||||||
 マップデータ マップデータ | Map after postprocessing | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス) | |||||||||
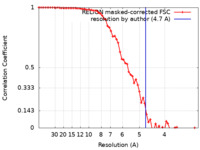
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.7 Å | |||||||||
 データ登録者 データ登録者 | Tsybovsky Y / Stephens T / Kanekiyo M / Graham BS | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Glycan repositioning of influenza hemagglutinin stem facilitates the elicitation of protective cross-group antibody responses. 著者: Seyhan Boyoglu-Barnum / Geoffrey B Hutchinson / Jeffrey C Boyington / Syed M Moin / Rebecca A Gillespie / Yaroslav Tsybovsky / Tyler Stephens / John R Vaile / Julia Lederhofer / Kizzmekia S ...著者: Seyhan Boyoglu-Barnum / Geoffrey B Hutchinson / Jeffrey C Boyington / Syed M Moin / Rebecca A Gillespie / Yaroslav Tsybovsky / Tyler Stephens / John R Vaile / Julia Lederhofer / Kizzmekia S Corbett / Brian E Fisher / Hadi M Yassine / Sarah F Andrews / Michelle C Crank / Adrian B McDermott / John R Mascola / Barney S Graham / Masaru Kanekiyo /  要旨: The conserved hemagglutinin (HA) stem has been a focus of universal influenza vaccine efforts. Influenza A group 1 HA stem-nanoparticles have been demonstrated to confer heterosubtypic protection in ...The conserved hemagglutinin (HA) stem has been a focus of universal influenza vaccine efforts. Influenza A group 1 HA stem-nanoparticles have been demonstrated to confer heterosubtypic protection in animals; however, the protection does not extend to group 2 viruses, due in part to differences in glycosylation between group 1 and 2 stems. Here, we show that introducing the group 2 glycan at Asn38 to a group 1 stem-nanoparticle (gN38 variant) based on A/New Caledonia/20/99 (H1N1) broadens antibody responses to cross-react with group 2 HAs. Immunoglobulins elicited by the gN38 variant provide complete protection against group 2 H7N9 virus infection, while the variant loses protection against a group 1 H5N1 virus. The N38 glycan thus is pivotal in directing antibody responses by controlling access to group-determining stem epitopes. Precise targeting of stem-directed antibody responses to the site of vulnerability by glycan repositioning may be a step towards achieving cross-group influenza protection. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20911.map.gz emd_20911.map.gz | 8.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20911-v30.xml emd-20911-v30.xml emd-20911.xml emd-20911.xml | 19.3 KB 19.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20911_fsc.xml emd_20911_fsc.xml | 6.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20911.png emd_20911.png | 112 KB | ||
| マスクデータ |  emd_20911_msk_1.map emd_20911_msk_1.map | 22.2 MB |  マスクマップ マスクマップ | |
| その他 |  emd_20911_additional.map.gz emd_20911_additional.map.gz emd_20911_additional_1.map.gz emd_20911_additional_1.map.gz emd_20911_half_map_1.map.gz emd_20911_half_map_1.map.gz emd_20911_half_map_2.map.gz emd_20911_half_map_2.map.gz | 14.2 MB 14.2 MB 14.6 MB 14.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20911 http://ftp.pdbj.org/pub/emdb/structures/EMD-20911 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20911 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20911 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20911_validation.pdf.gz emd_20911_validation.pdf.gz | 78.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20911_full_validation.pdf.gz emd_20911_full_validation.pdf.gz | 78 KB | 表示 | |
| XML形式データ |  emd_20911_validation.xml.gz emd_20911_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20911 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20911 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20911 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20911 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20911.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20911.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map after postprocessing | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.58 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_20911_msk_1.map emd_20911_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Map before postprocessing
| ファイル | emd_20911_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map before postprocessing | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Map before postprocessing
| ファイル | emd_20911_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map before postprocessing | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map 1
| ファイル | emd_20911_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map 2
| ファイル | emd_20911_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : H1ssF gN38
| 全体 | 名称: H1ssF gN38 |
|---|---|
| 要素 |
|
-超分子 #1: H1ssF gN38
| 超分子 | 名称: H1ssF gN38 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Ferritin-based nanoparticle displaying Influenza hemagglutinin stems |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: 293 Expi Homo sapiens (ヒト) / 組換細胞: 293 Expi |
| 分子量 | 理論値: 1.031 MDa |
-分子 #1: H1ssF gN38
| 分子 | 名称: H1ssF gN38 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKAKLLVLLC TFTATYADTI CIGYHANNST DTVDTVLEKN VTVTNSTNLG SGLRMVTGLR NIPQRETRGL FGAIAGFIEG GWTGMVDGWY GYHHQNEQGS GYAADQKSTQ NAINGITNMV NSVIEKMGSG GSGTDLAELL VLLLNERTLD FHDSNVKNLY EKVKSQLKNN ...文字列: MKAKLLVLLC TFTATYADTI CIGYHANNST DTVDTVLEKN VTVTNSTNLG SGLRMVTGLR NIPQRETRGL FGAIAGFIEG GWTGMVDGWY GYHHQNEQGS GYAADQKSTQ NAINGITNMV NSVIEKMGSG GSGTDLAELL VLLLNERTLD FHDSNVKNLY EKVKSQLKNN AKEIGNGCFE FYHKCNNECM ESVKNGTYDY PKYSEESKLN REKIDSGGDI IKLLNEQVNK EMQSSNLYMS MSSWCYTHSL DGAGLFLFDH AAEEYEHAKK LIIFLNENNV PVQLTSISAP EHKFEGLTQI FQKAYEHEQH ISESINNIVD HAIKSKDHAT FNFLQWYVAE QHEEEVLFKD ILDKIELIGN ENHGLYLADQ YVKGIAKSRK SGS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: PBS |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 297 K / 装置: FEI VITROBOT MARK IV / 詳細: Drop volume: 2.7 microliters. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS TALOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 撮影したグリッド数: 1 / 実像数: 248 / 平均露光時間: 2.0 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 92000 |
| 試料ステージ | 試料ホルダーモデル: GATAN 626 SINGLE TILT LIQUID NITROGEN CRYO TRANSFER HOLDER ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)