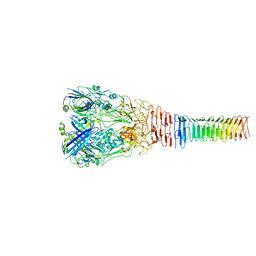
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20698 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Francisella PdpA-VgrG Complex, half-lidded | |||||||||
 マップデータ マップデータ | Francisella PdpA-VgrG Complex, half-lidded | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | T6SS Central Spike / Complex / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 | symbiont cell surface / host cell cytoplasm / Uncharacterized protein / Uncharacterized protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Francisella tularensis subsp. novicida U112 (バクテリア) / Francisella tularensis subsp. novicida U112 (バクテリア) /  Francisella tularensis subsp. novicida (strain U112) (バクテリア) Francisella tularensis subsp. novicida (strain U112) (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.98 Å | |||||||||
 データ登録者 データ登録者 | Yang X / Clemens DL | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2019 ジャーナル: Structure / 年: 2019タイトル: Atomic Structure of the Francisella T6SS Central Spike Reveals a Unique alpha-Helical Lid and a Putative Cargo. 著者: Yang X / Clemens DL / Lee BY / Cui Y / Zhou ZH / Horwitz MA | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20698.map.gz emd_20698.map.gz | 9.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20698-v30.xml emd-20698-v30.xml emd-20698.xml emd-20698.xml | 12.7 KB 12.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20698.png emd_20698.png | 69.4 KB | ||
| Filedesc metadata |  emd-20698.cif.gz emd-20698.cif.gz | 5.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20698 http://ftp.pdbj.org/pub/emdb/structures/EMD-20698 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20698 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20698 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20698_validation.pdf.gz emd_20698_validation.pdf.gz | 350.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20698_full_validation.pdf.gz emd_20698_full_validation.pdf.gz | 350.2 KB | 表示 | |
| XML形式データ |  emd_20698_validation.xml.gz emd_20698_validation.xml.gz | 6.8 KB | 表示 | |
| CIF形式データ |  emd_20698_validation.cif.gz emd_20698_validation.cif.gz | 7.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20698 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20698 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20698 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20698 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20698.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20698.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Francisella PdpA-VgrG Complex, half-lidded | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : PdpA-VgrG complex
| 全体 | 名称: PdpA-VgrG complex |
|---|---|
| 要素 |
|
-超分子 #1: PdpA-VgrG complex
| 超分子 | 名称: PdpA-VgrG complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Francisella tularensis subsp. novicida U112 (バクテリア) Francisella tularensis subsp. novicida U112 (バクテリア) |
-分子 #1: PdpA
| 分子 | 名称: PdpA / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Francisella tularensis subsp. novicida (strain U112) (バクテリア) Francisella tularensis subsp. novicida (strain U112) (バクテリア)株: U112 |
| 分子量 | 理論値: 95.469961 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MIAVKDITDL NIQDIISQLT SEVINGDTTP SSAKFACEIN SYIINYNLSN INLINTQLKN TKILYRKGLI SKLDYEKYKR YCIISRFKN NIDEFILYFS TNYKDSQSLK IAIKELQNSC SSSLILELPH DYIRKIDVLL TSIDSAIQRS SDLNKTIIKQ L NKLRSSLS ...文字列: MIAVKDITDL NIQDIISQLT SEVINGDTTP SSAKFACEIN SYIINYNLSN INLINTQLKN TKILYRKGLI SKLDYEKYKR YCIISRFKN NIDEFILYFS TNYKDSQSLK IAIKELQNSC SSSLILELPH DYIRKIDVLL TSIDSAIQRS SDLNKTIIKQ L NKLRSSLS RYIGYNNVLQ KQEITINIKP INKNFELEDI SFVSTRNKQY FKHNSLTLKN PHIEKLEVCE NIYGINGWLT FD LAYINNH KDFNFLLSPN QPILLDIQIN DSFNFYKKES KKDHHKRTTR FIAIGFNSNS IDIHENFEYS IYSYTKNVSS GVK KFKIQF HDPLKALWTK HKPSYIALNK SLDDIFKDNF FFDSLFSLDT NKSNNLKIRI PQAFISTVNR NFYDFFIQQL EQNK CYLKY FCDKKSGKVS YHVVDQVDND LQRNIVNSDE DLKDKLSPYD ISCFKKQILI SNKSNFYVKE KNICPDVTLN TQRKE DRKI SDTLVKPFSS ILKDNLQSVE YIQSNNDDKQ EIITTGFEIL LTSRNTLPFL DTEITLSKLD NDQNYLLGAT DIKSLY ISQ RKLLFKRSKY CSKQLYENLH NFHYKSDSES DVYEKIAFTK YPSLTHDNSI TYKIKDYSNL TPEYPKYKSF SNFYING RI TIGENVNNDS KKAYKFFKNH KPEESSIAEF QENGEKGTSA ILNSKADILY AIEIAKEMLS DKSSDKPIIY LPLKVNIN S ANNQFIPLRN DDIILIEIQS FTKGEIIELI SNSAISTKKA QQQLLQRQLL GSKENCEMAY TQTSDSETFS LTQVNEDCE NSFLINDKKG IFLRYKSKGN UniProtKB: Uncharacterized protein |
-分子 #2: VgrG
| 分子 | 名称: VgrG / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Francisella tularensis subsp. novicida (strain U112) (バクテリア) Francisella tularensis subsp. novicida (strain U112) (バクテリア)株: U112 |
| 分子量 | 理論値: 20.539779 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDYKDDDDKD YKDDDDKDYK DDDDKGSKAD HIFNLEEQGL LIDIKDDSKG CTTKLESSGK ITHNATESIE SSADKQIIEN VKDSKISIT EKEILLATKK SSIMLSEDKI VIKIGNSLII LDDSNISLES ATINIKSSAN INIQASQNID IKSLNNSIKA D VNLNAEGL ...文字列: MDYKDDDDKD YKDDDDKDYK DDDDKGSKAD HIFNLEEQGL LIDIKDDSKG CTTKLESSGK ITHNATESIE SSADKQIIEN VKDSKISIT EKEILLATKK SSIMLSEDKI VIKIGNSLII LDDSNISLES ATINIKSSAN INIQASQNID IKSLNNSIKA D VNLNAEGL DVNIKGSVTA SIKGSAATMV G UniProtKB: Uncharacterized protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 47.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C3 (3回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.98 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 10343 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル | 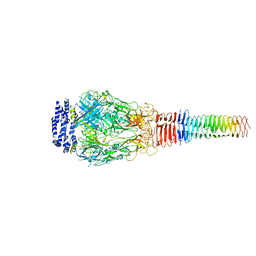 PDB-6u9g: |
 ムービー
ムービー コントローラー
コントローラー