+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20477 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Attachment of STIV to Sulfolobus pili | |||||||||
 マップデータ マップデータ | Attachment of Sulfolobus Turreted Icosahedral Virus (STIV) to Sulfolobus pili | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |    Sulfolobus turreted icosahedral virus 1 (ウイルス) Sulfolobus turreted icosahedral virus 1 (ウイルス) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 20.0 Å | |||||||||
 データ登録者 データ登録者 | Lawrence CM / Young MA / Hartman RA | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2019 ジャーナル: Structure / 年: 2019タイトル: The Molecular Mechanism of Cellular Attachment for an Archaeal Virus. 著者: Ross Hartman / Brian J Eilers / Daniel Bollschweiler / Jacob H Munson-McGee / Harald Engelhardt / Mark J Young / C Martin Lawrence /   要旨: Sulfolobus turreted icosahedral virus (STIV) is a model archaeal virus and member of the PRD1-adenovirus lineage. Although STIV employs pyramidal lysis structures to exit the host, knowledge of the ...Sulfolobus turreted icosahedral virus (STIV) is a model archaeal virus and member of the PRD1-adenovirus lineage. Although STIV employs pyramidal lysis structures to exit the host, knowledge of the viral entry process is lacking. We therefore initiated studies on STIV attachment and entry. Negative stain and cryoelectron micrographs showed virion attachment to pili-like structures emanating from the Sulfolobus host. Tomographic reconstruction and sub-tomogram averaging revealed pili recognition by the STIV C381 turret protein. Specifically, the triple jelly roll structure of C381 determined by X-ray crystallography shows that pilus recognition is mediated by conserved surface residues in the second and third domains. In addition, the STIV petal protein (C557), when present, occludes the pili binding site, suggesting that it functions as a maturation protein. Combined, these results demonstrate a role for the namesake STIV turrets in initial cellular attachment and provide the first molecular model for viral attachment in the archaeal domain of life. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20477.map.gz emd_20477.map.gz | 20.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20477-v30.xml emd-20477-v30.xml emd-20477.xml emd-20477.xml | 10.1 KB 10.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20477.png emd_20477.png | 180.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20477 http://ftp.pdbj.org/pub/emdb/structures/EMD-20477 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20477 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20477 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20477_validation.pdf.gz emd_20477_validation.pdf.gz | 78 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20477_full_validation.pdf.gz emd_20477_full_validation.pdf.gz | 77.1 KB | 表示 | |
| XML形式データ |  emd_20477_validation.xml.gz emd_20477_validation.xml.gz | 495 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20477 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20477 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20477 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20477 | HTTPS FTP |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20477.map.gz / 形式: CCP4 / 大きさ: 3.7 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20477.map.gz / 形式: CCP4 / 大きさ: 3.7 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Attachment of Sulfolobus Turreted Icosahedral Virus (STIV) to Sulfolobus pili | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
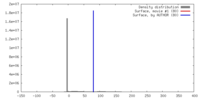
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Sulfolobus turreted icosahedral virus 1
| 全体 | 名称:    Sulfolobus turreted icosahedral virus 1 (ウイルス) Sulfolobus turreted icosahedral virus 1 (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Sulfolobus turreted icosahedral virus 1
| 超分子 | 名称: Sulfolobus turreted icosahedral virus 1 / タイプ: virus / ID: 1 / 親要素: 0 / NCBI-ID: 269145 / 生物種: Sulfolobus turreted icosahedral virus 1 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Saccharolobus solfataricus P2 (古細菌) / 株: P2^3 Saccharolobus solfataricus P2 (古細菌) / 株: P2^3 |
| ウイルス殻 | Shell ID: 1 / 直径: 740.0 Å / T番号(三角分割数): 31 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 緩衝液 | pH: 2.5 / 構成要素 - 濃度: 5.0 mM / 構成要素 - 式: C6H8O7 / 構成要素 - 名称: Citric Acid |
|---|---|
| グリッド | モデル: Quantifoil R2/4 / 材質: COPPER / メッシュ: 200 |
| 凍結 | 凍結剤: ETHANE-PROPANE / 装置: FEI VITROBOT MARK III |
| 詳細 | 1 ml S. solfactaricus cells at approximately 5E10^8 cells/ml was combined with 100 ul of 1.88 mg/ml virus and incubated for 20 minutes at 80C. Unbound virus was removed by centrifugation. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 位相板: VOLTA PHASE PLATE |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称) / 解像度のタイプ: BY AUTHOR / 解像度: 20.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: PEET / 使用したサブトモグラム数: 7 |
|---|---|
| 抽出 | トモグラム数: 1 / 使用した粒子像数: 3 / ソフトウェア - 名称: PEET |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)