+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the full-length Pseudomonas aeruginosa bacteriophytochrome in its Pr state | |||||||||
 マップデータ マップデータ | Composite map generated with phenix combine maps. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Photosensor / Photoreceptor / Phytochrome / Bacterial protein / CYTOSOLIC PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報osmosensory signaling via phosphorelay pathway / detection of visible light / phosphorelay response regulator activity / phosphorelay sensor kinase activity / histidine kinase / photoreceptor activity / protein kinase activator activity / regulation of DNA-templated transcription / ATP binding / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
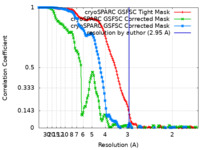
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.95 Å | |||||||||
 データ登録者 データ登録者 | Bodizs S / Westenhoff S | |||||||||
| 資金援助 |  スウェーデン, 1件 スウェーデン, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2024 ジャーナル: Structure / 年: 2024タイトル: Cryo-EM structures of a bathy phytochrome histidine kinase reveal a unique light-dependent activation mechanism. 著者: Szabolcs Bódizs / Petra Mészáros / Lukas Grunewald / Heikki Takala / Sebastian Westenhoff /   要旨: Phytochromes are photoreceptor proteins in plants, fungi, and bacteria. They can adopt two photochromic states with differential biochemical responses. The structural changes transducing the signal ...Phytochromes are photoreceptor proteins in plants, fungi, and bacteria. They can adopt two photochromic states with differential biochemical responses. The structural changes transducing the signal from the chromophore to the biochemical output modules are poorly understood due to challenges in capturing structures of the dynamic, full-length protein. Here, we present cryoelectron microscopy (cryo-EM) structures of the phytochrome from Pseudomonas aeruginosa (PaBphP) in its resting (Pfr) and photoactivated (Pr) state. The kinase-active Pr state has an asymmetric, dimeric structure, whereas the kinase-inactive Pfr state opens up. This behavior is different from other known phytochromes and we explain it with the unusually short connection between the photosensory and output modules. Multiple sequence alignment of this region suggests evolutionary optimization for different modes of signal transduction in sensor proteins. The results establish a new mechanism for light-sensing by phytochrome histidine kinases and provide input for the design of optogenetic phytochrome variants. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19981.map.gz emd_19981.map.gz | 230.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19981-v30.xml emd-19981-v30.xml emd-19981.xml emd-19981.xml | 16 KB 16 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19981_fsc.xml emd_19981_fsc.xml emd_19981_fsc_2.xml emd_19981_fsc_2.xml emd_19981_fsc_3.xml emd_19981_fsc_3.xml | 13.3 KB 13.3 KB 13.3 KB | 表示 表示 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19981.png emd_19981.png | 69.3 KB | ||
| Filedesc metadata |  emd-19981.cif.gz emd-19981.cif.gz | 6.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19981 http://ftp.pdbj.org/pub/emdb/structures/EMD-19981 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19981 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19981 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_19981_validation.pdf.gz emd_19981_validation.pdf.gz | 496 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_19981_full_validation.pdf.gz emd_19981_full_validation.pdf.gz | 495.6 KB | 表示 | |
| XML形式データ |  emd_19981_validation.xml.gz emd_19981_validation.xml.gz | 14.2 KB | 表示 | |
| CIF形式データ |  emd_19981_validation.cif.gz emd_19981_validation.cif.gz | 18.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19981 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19981 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19981 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19981 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9eutMC  9euyC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19981.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19981.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Composite map generated with phenix combine maps. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.828 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Homodimeric complex of the protein bacteriophytochrome containing...
| 全体 | 名称: Homodimeric complex of the protein bacteriophytochrome containing its cofactor biliverdin |
|---|---|
| 要素 |
|
-超分子 #1: Homodimeric complex of the protein bacteriophytochrome containing...
| 超分子 | 名称: Homodimeric complex of the protein bacteriophytochrome containing its cofactor biliverdin タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 160 KDa |
-分子 #1: Bacteriophytochrome
| 分子 | 名称: Bacteriophytochrome / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: histidine kinase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 82.094867 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTSITPVTLA NCEDEPIHVP GAIQPHGALV TLRADGMVLA ASENIQALLG FVASPGSYLT QEQVGPEVLR MLEEGLTGNG PWSNSVETR IGEHLFDVIG HSYKEVFYLE FEIRTADTLS ITSFTLNAQR IIAQVQLHND TASLLSNVTD ELRRMTGYDR V MAYRFRHD ...文字列: MTSITPVTLA NCEDEPIHVP GAIQPHGALV TLRADGMVLA ASENIQALLG FVASPGSYLT QEQVGPEVLR MLEEGLTGNG PWSNSVETR IGEHLFDVIG HSYKEVFYLE FEIRTADTLS ITSFTLNAQR IIAQVQLHND TASLLSNVTD ELRRMTGYDR V MAYRFRHD DSGEVVAESR REDLESYLGQ RYPASDIPAQ ARRLYIQNPI RLIADVAYTP MRVFPALNPE TNESFDLSYS VL RSVSPIH CEYLTNMGVR ASMSISIVVG GKLWGLFSCH HMSPKLIPYP VRMSFQIFSQ VCSAIVERLE QGRIAELLRV STE RRLALA RRARDADDLF GALAHPDDGI AALIPCDGAL VMLGGRTLSI RGDFERQAGN VLQRLQRDPE RDIYHTDNWP QPSE DSPDG GDCCGVLAIR FHRQESGWIF WFRHEEVHRI RWGGKPEKLL TIGPSGPRLT PRGSFEAWEE VVRGHSTPWS ETDLA IAEK LRLDLMELCL NHAAEVDRMR QRLIAVLGHD LRNPLQSISM AAALLSSSDT RTTELRQHIS ASSSRMERLV SQILDM SRL QSGIGLTVNP VDTDVSQLVR QIVCETDVAY PGLVIEIAID PQVRAVVDPD RYAQVAANLL SNARHHGLPG RPVLVTL TR QGDEVCLSVL NETSGLSEAQ LANLFEPFKR ESADNQRNRN GLGIGLYISQ AIAQAHQGRI DVDCRDDVIT FCLRLPVR Q AETGSSSLEH HHHHH UniProtKB: Bacteriophytochrome |
-分子 #2: 3-[2-[(Z)-[3-(2-carboxyethyl)-5-[(Z)-(4-ethenyl-3-methyl-5-oxidan...
| 分子 | 名称: 3-[2-[(Z)-[3-(2-carboxyethyl)-5-[(Z)-(4-ethenyl-3-methyl-5-oxidanylidene-pyrrol-2-ylidene)methyl]-4-methyl-pyrrol-1-ium -2-ylidene]methyl]-5-[(Z)-[(3E)-3-ethylidene-4-methyl-5-oxidanylidene- ...名称: 3-[2-[(Z)-[3-(2-carboxyethyl)-5-[(Z)-(4-ethenyl-3-methyl-5-oxidanylidene-pyrrol-2-ylidene)methyl]-4-methyl-pyrrol-1-ium -2-ylidene]methyl]-5-[(Z)-[(3E)-3-ethylidene-4-methyl-5-oxidanylidene-pyrrolidin-2-ylidene]methyl]-4-methyl-1H-pyrrol-3- yl]propanoic acid タイプ: ligand / ID: 2 / コピー数: 2 / 式: LBV |
|---|---|
| 分子量 | 理論値: 585.67 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.8 構成要素:
詳細: 80 mM Tris, 10 mM MgCl2, 150 mM CH3CO2K, pH 7.8 | ||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 50 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: OTHER / 前処理 - 気圧: 0.04 kPa / 詳細: Current: 20 mA | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: Blotting for 5 s from both sides. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 2 / 実像数: 30336 / 平均露光時間: 1.7 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 2.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 得られたモデル |  PDB-9eut: |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)